Integration von Signalen
Werbung
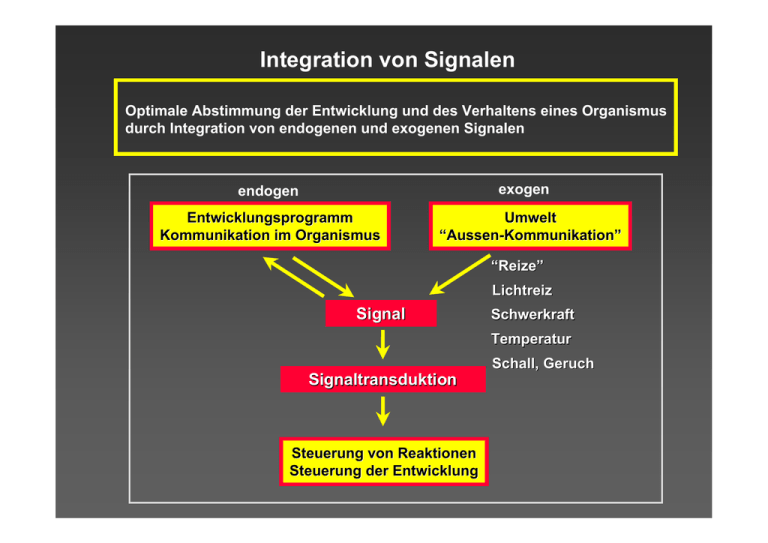
Integration von Signalen Optimale Abstimmung der Entwicklung und des Verhaltens eines Organismus durch Integration von endogenen und exogenen Signalen exogen endogen Entwicklungsprogramm Kommunikation im Organismus Umwelt “Aussen-Kommunikation” “Reize” Lichtreiz Signal Schwerkraft Temperatur Schall, Geruch Signaltransduktion Steuerung von Reaktionen Steuerung der Entwicklung Exogene Signale exogene Signale Tier Pflanze Mikroorg.men + + + +/- - - (Perzeption der Gasphase) + + + Geschmacksstoffe + + + + + + + + + + + + Licht Schall Geruchsstoffe (Perzeption der Flüssigphase) Widerstand (Fühlen) (Perzeption der Festphase) Temperatur Schwerkraft Endogene Signale/ Botenstoffe bei Säuger und Pflanze endogene Signale Tier/Mensch Pflanze + + Wachstumshormone + Adrenalin Auxin (P) + + Östrogen Brassinosteroide (P) + + Jasmonat (P) + + Acetylcholin Histamin + + Ca, cGMP, Beispiel Hormone-Endokrine / Phytohorm. (intraorganism. intraorganism. Kommunikation, speziell der Organe) Organe) Peptid/Protein Aminosäurederivate Terpenderivate (u.a. Steroide) + weitere Botenstoffe Pheromone (interorgamism. interorgamism. Kommunikation über chemische Signale) Transmitter (interzelluläre, interzelluläre, lokale Kommunikation) sekundäre Botenstoffe (intrazelluläre Kommunikation, Bildung nach SignalSignal-RezeptorRezeptorInteraktion) Phytohormone als zellübergreifende endogene Signale exogen endogen Entwicklungsprogramm Kommunikation im Organismus Umwelt Integration von Außenfaktoren Endogenes Signal Phytohormon Signaltransduktion Ionenflüsse Enzymaktivitäten Genexpression Steuerung von Reaktionen und der Entwicklung Cytoskelett Definition Phytohormone: sind niedermolekulare pflanzliche Botenstoffe, die in geringen Konzentration (< 10 -5 M) eine regulatorische, in der Regel zellübergreifende, Wirkung zeigen ohne hierbei selbst chemisch verändert zu werden. Hormonrezeptoren: sind hochaffine und spezifische Bindestellen für Hormone, die durch eine reversible Bindung des Liganden gekennzeichnet sind und dieses Signal im Sinne einer Reizleitung transduzieren. Einteilung der Phytohormone wachstumsstimulierende Phytohormone Auxine Cytokinine Gibberelline Brassinosteroide wachstumsinhibierende Phytohormone Ethylen Abscisinsäure Jasmonsäure Salicylsäure wachstumstimulierende Phytohormone (Übersicht) elongation growth cell division apical dominance adventitous roots -CH2-COOH indol acetic acid N H _ HN-CH2-CH=C Cytokinin cell division adventitous buds antagonist of senescence _ CH2OH _ Auxin CH3 N N zeatin N H N _ O CO Gibberellin HO _ _ _ H3C HOOC CH2 gibberellic acid 1 Brassinosteroid OH _ elongation growth germination flower control _ CH3 elongation growth _ CH3 HO _ campesterol wachstumsinhibierende Phytohormone (Übersicht) Ethylene Abscisic acid Jasmonic acid fruit ripening senecence leaf abscission stomatal closure dormancy desiccation tolerance cold tolerance pathogen response wound response tendril coiling H C C H H H OH COOH O O COOH COOH Salicylic acid thermogenesis pathogen response OH Regulation endogener Signale Transport Konjugierung Speicherung Biosynthese Katabolismus Physiologisch aktives Signal Signaltransduktion Def. Signaltransduktion: Perzeption und Übertragung eines Signals, d.h. exogenen Reizes oder endogenen Botenstoffes, in einer molekularen Wirkkette -CH2-COOH Auxin N H indol acetic acid wichtigstes Auxin: Indolessigsäure vorwiegende Bildungsorte: Transport Meristeme Embryonen junge Blätter basipetal: Spross, vom Apex zur Basis Wurzel unter Beteiligung des Auxinefflux-Carriers PIN1, der über Vesikel ortsselektiv in das Plasmalemma inseriert wird, und AUX1 (Auxin-Influx-Carrier) z.T. ungerichtete Diffusion Phyllotaxis induced by exogenous auxin pin1 mutant Reinhardt et al, Nature 426, 255 (2003) Secondary Root Formation Auxin-Verteilung (Auxin-induzierte GUS Expression, blau, und Lokalisation uber Auxin-spez. Antikörper, braun) Benkova et al., Cell 115, 591 (2003) PIN1 Expression Wirkung Auxin fördert: Streckungswachstum (i.d.R.) Spross u. Wurzel unterschiedliche Sensitivität (Überversorgung hemmt!) Teilungsaktivität des Kambiums induziert Entwicklung des interfascikulären Kambiums (sek. Dickenwachstum) Usambaraveilchen Seitenwurzelbildung Auxin wirkt hier antagonistisch zu Cytokinin 10 Tage Stecklingsbewurzelung durch Auxin (v.a. Indolbuttersäure) gefördert Adventivwurzeln + Auxin Apikaldominaz Entwicklung von Seitensprossen wird unterdrückt - Auxin Fruchtentwicklung wird durch Auxin induziert Kontrolle Samen entfernt bis auf einen ! Samen entfernt bis auf drei ! Samen entfernt + Auxin - Auxin Samenlose Frucht (parthenokarp) Auxinapplikation auf Fruchtknoten löst vielfach samenlose (parthenokarpe) Fruchtentwicklung aus z.B. bei Erdbeere, Tomate, Wassermelone Biosynthese über Tryptophan - C=N Indol-3-acetonitril 4 Tao et al., 2008 Cell 133,164-176 1) Trp-deaminase 2) Indol-3-pyruvat-Decarboxylase 3) Indol-3-acetaldehyd-DH 4) Indol-3-acetonitril-Nitrilase npr Agrobacterium tumefaciens Gram-negatives Bodenbakterium Pflanzenpathogen: Verursacher des Wurzelhalstumors bei Dicotylen ca. 90 Pflanzenfamilen (>600 Spezies) unter Kulturbedingungen auch Monocotyle wie Reis und Mais Pflanzenkontakt: Einschleusen der einzelsträngigen T-DNA (ca. 23 kb), Region des Ti-Plasmids, Integration ins Wirtgenom, T-DNA Gene für Auxin- und Cytokininbiosynthese Gene für Opinbiosynthese eukaryotische Transkription ! transgene Tabakpflanze mit grün-fluoreszierendem Quallenprotein (GFP) Genome of Agrobacterium tumefaciens strain C58 and mechanism of plant-cell transformation. npr The four genetic elements of the Agrobacterium genome are represented in relative scale to each other. The circular chromosome (purple) is 2.84 Mb and contains 2,750 ORFs; the linear chromosome (blue) is 2.08 Mb and contains 1,850 ORFs; plasmid pAtC58 (green) is 543 kb and contains 550 ORFs; and plasmid pTiC58 (red) is 214 kb and contains 198 ORFs. Agrobacterium transfers the T-DNA as a complex with the VirD2 protein to the plant cell via a type IV transporter system encoded on pTiC58. The T-DNA is imported to the nucleus, where it becomes integrated into the plant-cell genome. Expression of wild-type T-DNA– encoded genes results in tumorigenesis. In the laboratory, replacement of T-DNA sequences with recombinant DNA allows genetic engineering of susceptible species. Zupan J, Ward D, Zambryski P. (2002) Nat Biotechnol. 20:129-131. npr Gen 1 Auxin-Inaktivierung Oxidation: IAA-Oxidase Konjugation an IAA-Carboxygruppe: - von Glucose über C1 (reversibel) - von Aspartat über N (irreversibel Abbau) Inaktivierung von Auxin initiiert durch IAAIAA-Oxidase Auxin-Signaltransduktion Targets? Analyse? Auxin-abhängige Genregulation über ARF Transkriptionsfaktoren Transkriptionsfaktoren ARF (auxin-response factors, sind Aktivatoren) AUX/IAA homodimerisieren bzw. ARF heterodimerisieren mit Transkriptionsregulatoren AUX Inaktives Heterodimer bzw. IAA (wirken als Repressor, 24 Vertreter in Arabidopsis) ARF-AUX/IAA Heterodimer inaktiv! Aktivierung der ARF durch Abbau des Inhibitors IAA/AUX über Ubiquitinierung von AUX/IAA (Dömäne II interagiert mit Ubiquitinligase) Experimenteller Beweis: Auxin-insensitive IAA/AUX Mutanten mit Defekt im Abbau (spezifisch für Auxinwirkung!) ARF ARF Aktives Dimer Abbau von AUX/IAA beseitigt negative Regulation der Auxin-Antwort, d.h. stimuliert Auxin-Signalisation Gray et al. (2001) Nature 414, 271 Fusionsproteine von IAA-Glucuronidase sind stabilisiert in der Auxin-insensitiven Mutante axr2 mit mutiertem IAA7-Gen (=AXR2) und axr3 (=mutiertes IAA17) Ubiquitin - 76 Aminosäuren - C-terminales Gly wird reversibel an Lysin-Reste anderer Proteine konjugiert (Isopeptidbindung) - hochkonserviert (> 95 % Identität zwischen Homo sapiens und Arabidopsis thaliana) Funktionen • Monoubiquitinierung: Stabilisierung • Polyubiquitinierung: Abbau der Proteine durch Proteolyse RUB und SUMO mit struktureller Homologie zu Ubiquitin 60 % identity Ubiquitin RUB1 (NEDD8) SUMO Ubiquitinierung + ATP SH X E1 C O - O O C AMP O C S NH3+ Monoubiquitinierung: ¾ Histone ¾ Proteolyse E2 E3 O C SH S AMP E2 E1 E1: UbIquitin-aktivierendes Enzym E2: UbIquitin-konjugierendes Enzym E3: UbIquitin-Ligase (SCF-Komplex) 2 >20 C O N X PPi Polyubiquitinierung und Proteolyse > 500 Gene in Arabidopsis Ubiquitin und strukturverwandtes RUB in der Auxin-Signaltransduktion Auxin AXR1 (RUB-E1 UE) RCE1 E1 E2 Cullin SKP F-box ARF AUX/IAA AUX/IAA ARF Ubi RUB AUX/IAA E3 (SCFTIR1 Complex) inaktiv E3: Ubiquitin-Ligas heterotrimer Untereinheiten SCF SKP1, Cullin, F-box Cullin Kontakt mit E aktiviert durc SKP: Kontakt mit C F-box: Substraterkennung (TIR1 bei Au antwort) AUX/IAA 26S Proteasome TIR1: Auxinrezep Dharmasine et al a Nature 435, 441 Kepinski and Ley Nature 435, 446 Nemhauser and Cell 112, 970 a, An overall view of the TIR1 surface pocket occupied by auxin (green) at the bottom and the highly coiled IAA7 peptide (orange) on top. The surface of the three long top surface loops of TIR1 responsible for ligand binding is coloured red. b, A close-up side view of the central GWPPV motif in the IAA7 peptide upon binding to TIR1. Interacting residues of the substrate peptide and TIR1 are shown as orange and yellow stick models, respectively. Auxin is shown as a green stick model. To clarify the view, two hydrophobic residues, one from the peptide and the other from loop-2 of TIR1, are not shown. c, A slab view of the TIR1–auxin–IAA7 peptide complex, showing that auxin fills a cavity between two proteins. The molecular surface of TIR1 is shown in grey mesh. The IAA7 peptide and auxin are shown in orange surface representation and green CPK, respectively. Xu et al. Nature 446, 640-645 (2007) Proteolyse-abhängige Genregulation der Phytohormone über F-Box Proteine Auxin-Antwort Aktivierung der ARF Transkriptionsfaktoren durch Abbau der inhibitorischen IAA/AUX Gibberellin-Antwort GA-Bindung an Rezeptor: Rezeptor interagiert mit F-Box und aktiviert den Abbau der inhibitorischen DELLA Proteine (konserviert in Kormophyten nicht in Moosen, Algen) Jasmonat-Antwort JA-Isoleucin Konjugat perzipiert durch Rezeptor COI (F-Box ), das den Abbau der inhibitorischen JAZ Proteine einleitet, Aktivierung der Transkriptionsfaktoren (MYC2) Jasmonat-Isoleucin-Signaltransduktion a, JAZ proteins are normally bound to transcription factors and inhibit their activity. b, In response to attack, jasmonoyl–isoleucine (JA–Ile, marked with a star) stabilizes the interaction between COI1 and JAZ. c, The JAZ protein is probably then modified by ubiquitin (U), so marking it for destruction. d, JAZ is destroyed, liberating the transcription factors; e, this allows transcription of genes that produce proteins involved in defence and development, as well as of JAZ genes to restrain the jasmonate response. (Only the COI1 component of the SCFCOI1 enzyme complex of which it is a part is shown.) Edward E. Farmer Nature 448, 659-660 (2007) Yan et al. Plant Cell 8, 2220 (2009) N N Cytokinin _ _ HN-CH2-CH=C _ CH2OH CH3 zeatin N H wichtigstes Cytokinin: Zeatin = Isopentenyladenin (Purinbiosyntheseweg) N vorwiegende Bildungsorte: Transport Wirkung Wurzelspitze keimende Samen junge Gewebebereiche Biosynthese Wirkung eng gekoppelt unpolar Cytokinin fördert: Zellteilung und Streckungswachstum Cytokinin aktiviert u.a. Cyclin-cdk-Komplex (siehe Vorlesung PD. Torres) Stoffwechsel und verhindert Seneszenz Beispiel: Herbstlaub mit grünen Inseln (Cytokinin produziert von Mikroorganismus) Differenzierung u. Entwicklung im Zusammenspiel mit Auxin Cytokinin fördert Seitensprossbildung (Auxin unterdrückt) Bildung von “Hexenbesen” extrem: fascierte (verwachsene) Sprosse Cytokinin unterdrückt Seitenwurzelbildung (Auxin fördert) Cytokinin/Auxin regulierte Organentwicklung Beispiel: Organregeneration aus Kallus fascierte Sprosse von Salix Organdifferenzierung aus Kalli der Tabakpflanze (Nicotiana tabacum) Indolessigsäure (mg/l) 0 0.005 Kallus Wurzelinduktion 0 Kinetin (mg/l) 0.03 Kallusproliferation 0.2 Sprossinduktion 1 0.18 1 3 Biosynthese über AMP Nucleosidase Hydroxylierung Reduktion Signalkette 2- Komponentensystem Cytokininrezeptoren CRE 1, AHK2, AHK3, sind strukturell ähnlich dem Ethylen-Rezeptor ETR1 (To u. Kieber, 2008, Trends Plant Science 13, 85) Schematische Darstellung der Primärstriktur von CRE1 (CKI1) und ETR1 aus Arabidopsis Inoue et al. 2001, Nature 409,1060 Klonierung von CRE1 (CKI1) durch AktivierungsAktivierungs-Tagging Insertion der TT-DNA aktiviert Transkription benachbarter Gene Überexpression von CRE1 (C, D, G, H) führt zur CytokininCytokinin-hypersensitiven Sprossinduktion bei Arabidopsis Kalluskulturen (D, H). Kontrolle (B, F) AHK2,3 u. CRE1 sind Cytokininrezeptoren mit struktureller Homologie zu bakteriellen „2-Komponenten-Systemen“ bakterielle „2-Komponenten-Systeme bestehen aus Sensor (membranst. Rezeptor) und Regulator ähnlich den Cytokinin-u. Ethylenrezeptoren, Fusion von Sensor und Regulator (Transduzent) 2-Komponenten System (Enzym-gekoppelter Rezeptor) charakteristisch für prokaryotische Signalwege Hefe: Osmoregulation Pflanze: Ethylen- und Cytokininrezeptoren Histidin-spezifische PROTEINKINASE als Sensor überträgt Phosphatrest auf REGULATOR Protein (Phosphoaspartat-Bildung) Regulator häufig Transkriptionsregulator =Transduzent Sensor-histidinkinase z.B. CheA Regulator z.B. CheY Bakterielle Chemotaxis Histidin-Phosphotransmitter AHP (Arabidopsis Histidine-Phosphotransmitter) binden an Rezeptoren und werden phosphoryliert AHP1, AHP2 u. AHP5 zeigen Cytokinin-abhängige Lokalisation in Zellkern Responseregulator ARR (Arabidopsis Response Regulator) sind Transkriptionsfaktoren ARR1, ARR2 u. ARR10 sind Aktivatoren der Genexpression und der CytokininAntwort (TFs, ARR B-Typ) werden durch AHP1 u. AHP2 de-reprimiert ARR4, ARR5, ARR6 u. ARR7 (ARR A-Typ) sind negative Regulatoren der Cytokinin Antwort, werden durch Cytokinin induziert aus Hwang und Sheen 2001, Nature 413,383 Ektopische Expression von CKI oder ARR2 verstärkt die Cytokinin-Antwort aus Hwang und Sheen 2001, Nature











