Was ist ein Enzym? - Fachdidaktik Chemie ETH
Werbung

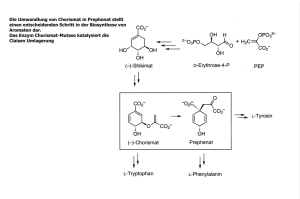
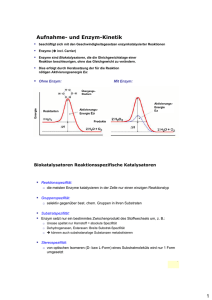
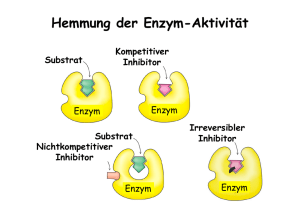
Wirkstoffentwicklung durch strukturbasiertes Design Medizinalchemie Dr. Andri Schütz Definitionen Ligand: (kleines) Molekül, welches an ein biologisches Makromolekül (z.B. Enzym) bindet Inhibitor: Ligand, der die Bindung des Substrats an ein Enzym verhindert IC50 Wert: Inhibitorkonzentration (in M, mM, μM, nM), bei welcher ein Enzym zu 50% «gebremst» wird. Analogie: H.-J. Böhm, G. Klebe, H. Kubinyi, Wirkstoffdesign, Spektrum, 2002. Proteine bestehen aus Aminosäuren-Ketten Das Beispiel Myoglobin – ein Muskelprotein Protein (Aminosäurenkette) Zusätzliche Moleküle (hier Porphin mit gebundenem O2) Ein Sulfat-Ion oder Phosphat-Ion Beispiel Myoglobin – ein Muskelprotein Ziel des strukturbasierten Ligandendesigns Werkzeug für die Entwicklung von medizinischen Wirkstoffen. Liganden/Inhibitor-Entwicklung. Liganden müssen selektiv und mit hoher Affinität an ein definiertes Zielenzym binden, und dadurch dessen Funktion beeinflussen. Ligand Zielenzym anderes Biomolekül/Enzym Ligand bindet selektiv Das Schlüssel—Schloss-Prinzip Emil Fischer (Nobelpreis 1902) Schloss Schlüssel Schlüssel—Schloss-Komplex Was sind die Folgen, wenn der Ligand kein «guter Schlüssel» ist? Was ist ein Enzym? Enzyme sind Biokatalysatoren. Sie beschleunigen chemische Reaktionen und ermöglichen so die Synthese aller benötigten „Bausteine“ eines biologischen Systems. Enzyme sind in der Regel Proteine, bestehen also aus einer langen Aufeinanderfolge von miteinander verbundenen Aminosäuren. Substrat bindet an Enzym Substrat Enzym Übergangszustand bildet sich aus Substrat Substrat Enzym Produkte verlassen das Enzym Enzym Produkte Produkte Enzym Reaktion findet statt Wie funktioniert ein Inhibitor? Inhibitoren binden an die aktive Tasche eines Enzyms und blockieren damit die enzymatische Reaktion. Inhibitor bindet ans Enzym Inhibitor Enzym Substrat kann nicht ans Enzym binden Inhibitor Enzym Inhibitor Substrat Enzym Produkte Reaktion findet nicht statt Das Schlüssel—Schloss-Prinzip Emil Fischer (Nobelpreis 1902) Schloss Schlüssel Schlüssel—Schloss-Komplex + Enzym Inhibitor Enzym—Inhibitor-Komplex Einen Schlüssel herzustellen,… ... ohne das Schloss zu kennen, ist schwer! Der Designzyklus von Liganden Krankheit „Biologie“ Ligandendesign (Computer) Strukturaufklärung Zielenzym Kristallstruktur Designter Ligand Biologisch aktive Moleküle (Assaydaten) Assay Klinische Studien, Medikamente Synthese Synthetisierter Ligand Strategien im strukturbasierten Ligandendesign Derivate des Substrats oder in der Natur vorkommender Liganden (Chemie) de-novo-Design (noch keine Liganden bekannt) (Computer & Chemie) Strukturen aus 3D-Datenbank-Suche (Computer) Strukturen aus Screening-Hits (Chemie) z.B. Fragment screening: Fragmente weiterentwickeln, verknüpfen, optimieren IC50-Wert: Konzentration des Inhibitors, welche das Enzym zu 50% inhibiert/verlangsamt. Projekt: Malaria/Plasmepsine Malaria: eine Tropenkrankheit (auch “Sumpffieber”) Der Erreger: Plasmodium Die Enzyme: Plasmepsine 1–4 ChlorochinResistenz SulfadoxinPyrimethaminResistenz Projekt: Malaria/Plasmepsine R2 IC50-Wert: Konzentration des Inhibitors, welche das Enzym zu 50% inhibiert/verlangsamt. O2S R1 N H N H H O O O Asp 34 IC50 = 50 nM O Asp 214 “Diaminnadel” Aminosäuren (21 natürlich vorkommende AS) Projekt: Malaria/Plasmepsine R2 O2S R1 N H N H H O O O Asp 34 O Asp 214 IC50 = 50 nM “Diaminnadel” Übungsaufgabe: Inhibitordesign Sie arbeiten auf dem Plasmepsin-Projekt als Student und diskutieren mit einem Kollegen über “Ihre” aktive Tasche und die zwischenmolekularen Kräfte zwischen ihrem Inhibitor (rot) und dem Enzym (schwarz) Der eine Kollege schlägt Ihnen vor, anstelle der Hexylgruppe einen Ether einzuführen. Was entgegnen Sie? Mögliche Diskussionspunkte: Intermolekulare WW Partitionierung zwischen Enzym und Wasser Affinität und Selektivität Haben Sie einen besseren Vorschlag anstelle des Ethers? Aufgabe: Resistenzen Ihr Inhibitor wird in Afrika als Antimalariawirkstoff eingesetzt. Plötzlich wird durch Ärzte festgestellt, dass das Medikament viel weniger zu wirken scheint. (= Entwicklung einer Resistenz). Ein Biologenteam untersucht die Erreger, gegen welche Ihr Wirkstoff schlechter wirkt und erklärt Ihnen, dass eine Mutation der Aminosäure Valin82 zu Tyrosin in der aktiven Tasche gefunden wurde. Wie erklären Sie sich die schlechtere Inhibition und wie würden sie die Seitenkette ihres Inhibitors anpassen, um diese Resistenz «umgehen» zu können? Mögliche Diskussionspunkte: Sterische Aspekte Platzverhältnisse in einer Enzymtasche Zwischenmolekulare WW WW zu Wasser Zusatzaufgabe: Selektive Bindung? ATP (AdenosinTriPhosphat) ist eine chemische Speicherform für Energie. Durch die Umwandlung von ATP zu ADP (AdenosinDiPhospat) im Körper können andere (endotherme/ungünstige) Reaktionen ablaufen. Das veränderte ATP wird als Inhibitor für Bakterienwachstum im Labor verwendet. Kann γ-S-ATP als Medikament gegen eine bakterielle Infektion eingesetzt werden?