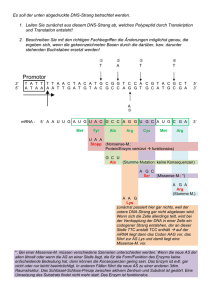
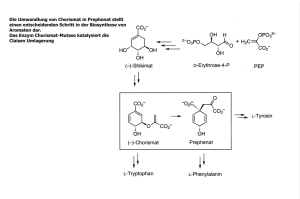
katalysiert besitzt
Werbung

Wissensmanagement in der Bioinformatik
Prof. Dr. Ulf Leser, Silke Trißl
Übungen zur Vorlesung „Molekularbiologische Datenbanken“
Lösungsblatt 1:
Datenbanksuche
Symptome
1.Ein Kind kommt in die Praxis und hat einen schweren geistigen Defekt (mental retardation).
Außerdem erzählt die Mutter, dass es an epileptischen Anfällen (seizures) leidet und auch oft
sehr übererregt (irritability) ist. Dem Arzt fällt auf, dass das Kind Pigmentstörungen auf der Haut
hat und außerdem einen unangenehmen Geruch wie Mäusekot ('mousy' odor) hat. Der Arzt
nimmt an, dass es sich bei der Krankheit um eine Erbkrankheit handeln könnte, da die Mutter
erzählt, dass eine Tante ähnliche Symptome aufgewiesen hat.
(OMIM http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?db=OMIM)
Um welche Krankheit kann es sich bei diesem Kind handeln und wie kann das überprüft werden?
Geben Sie ausserdem an, um welches um welches Gen bzw. das daraus resultierende Protein (auch
mit dem 2. Namen) es sich handelt mit der dazugehörigen EC Nummer.
Antwort:
OMIM-Acc: 261600, Phenyketonuria (PKU)
http://www.ncbi.nlm.nih.gov/entrez/dispomim.cgi?id=261600
RFLP mit Phenyalanine hydroxylase / (=Phenyalanine 4-monooxygenase) Gen
(EC 1.14.16.1)
2.Auf welchem Chromosom ist der Defekt lokalisiert und wie wird er vererbt und bei wem tritt er
auf?
Antwort:
Auf Chromosom 12, Vererbung erfolgt autosomal (nicht auf Geschlechtschromosomen (XY)) rezessiv, das heißt nur wenn Mutter und Vater beide das defekte
Gen besitzen und es auch an das Kind weitervererben tritt die Krankeit auf.
Von http://www.m-ww.de/krankheiten/erbkrankheiten/phenylketonurie.html
3.Ein Protein, das in einer Zelle einen biologischen Vorgang ermöglicht oder beschleunigt
(katalysiert) nennt man auch Enzym. Die Frage ist nun, welche Biochemische Reaktion wird
durch dieses Enzym katalysiert und in welchem biochemischen Pathway wird es benötigt
(KEGG: http://www.genome.ad.jp/kegg/kegg2.html)? Was bedeutet es, wenn dieses Enzym
nicht funktionell ist, d. h. die Reaktion nicht katalysiert?
Antwort:
L-phenylalanine + tetrahydrobiopterin + O(2) ->
L-tyrosine + dihydrobiopterin + H(2)O
Biochemical Pathway: phenylalanin, tyrosin and tryptophan pathway
http://www.genome.ad.jp/dbget-bin/get_pathway?org_name=hsa&mapno=00400
Phenylalanin wird nicht zu Thyrosin umgewandelt und bleibt somit im Körper
vorhanden. Dies führ zu den erwähnten Schäden.
4.Suchen Sie die Accession-Number der genomischen DNA und der mRNA (jeweils komplett) für
das entsprechende Gen beim Menschen (organism=human) aus EMBL heraus und notieren Sie
jeweils die Länge. Was ist der Grund für die Abweichung bei der Länge zwischen Genomischer
DNA und mRNA?
Antwort:
Genomische DNA:
EMBL-Accession: AF404777
Länge: 171 266 bp
mRNA:
EMBL-Accession: U49897 (ID: HS49897)
Länge: 2 680 bp
Introns in der genomischen DNA, die in der mRNA entfernt wurden.
5.Suchen Sie mit Hilfe der SPTREMBL Accession-Number heraus, welche molekularen
Funktionen dieses Enzyms besitzt und an welchen biologischen Prozessen es laut Gene Ontology
(QuickGo http://www.ebi.ac.uk/ego/index.html) beteiligt ist.
Antwort:
Biologischer Prozeß:
GO0009072
aromatic amino acid family metabolism
GO0008152
metabolism
GO0006559
phenylalanine catabolism
Molekulare Funktion:
GO0016597
amino acid binding activity
GO0005506
iron ion binding activity
GO0004497
monooxygenase activity
GO0004505
phenylalanine 4-monooxygenase activity
6.Finden Sie über eine Literatursuche heraus, (PubMed,
http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?db=PubMed) ob es eine eigene Datenbank über
Mutationen in diesem Gen gibt.
Antwort: Ja, http://www.pahdb.mcgill.ca/
7.Für welche Organismen sind Proteinsequenzen dem Enzym mit diesem Namen in Swissprot
vorhanden?
Antwort:
Sequenzen in SWISS-PROT
PH4H_CAEEL
Caenorhabditis elegans
PH4H_CAUCR
Caulobacter crescentus
PH4H_CHRVO
Chromobacterium violaceum
PH4H_DROME
Drosophila melanogaster (Fruit fly)
PH4H_HUMAN
Homo sapiens (Human)
PH4H_MOUSE
Mus musculus (Mouse)
PH4H_PSEAE
Pseudomonas aeruginosa
PH4H_RALSO
Ralstonia solanacearum (Pseudomonas solanacearum)
PH4H_RAT
Rattus norvegicus (Rat)
PH4H_RHILO
Rhizobium loti (Mesorhizobium loti)
PH4H_VIBCH
Vibrio cholerae
8.Für welche dieser Organismen aus SWISSPROT gibt es auch eine experimentell bestimmte
Struktur in PDB? Zeigen Sie diese mit dem Liganden FeIII.
Antwort:
1phz
1pah
1ltv
Abbildung 2: 1phz (Rat)
Rattus norvegicus (Rat)
Homo sapiens (Human)
Chromobacterium violaceum
Abbildung 3: 1pah (Human)
Abbildung 1: Überlappung der 3 Proteine durch SwissPDB Viewer
Abbildung 4: 1ltv (Chromobacterium)
9. Für alle, die noch etwas Zeit und Lust haben: Finden Sie mit einer Similaritätssuche (BLAST)
heraus, ob das Enzym noch zu weiteren Proteingruppen zu dem Behandelten Enzym homolog
sind.
Antwort:
Homolog zu Proteingruppen: Tryptophan 5-monooxygenase und
Tyrosine 3-monooxygenase