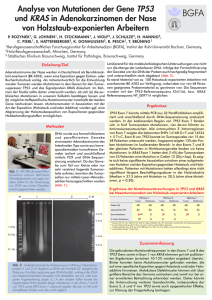
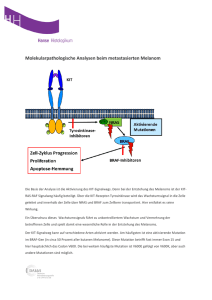
Thesis_Kett
Werbung

Universität Ulm Zentrum für Innere Medizin Klinik für Innere Medizin III Professor Dr. med. Hartmut Döhner Mutationsanalyse des Tumorsuppressorgens TP53 bei der akuten myeloischen Leukämie mit komplexem Karyotyp Dissertation zur Erlangung des Doktorgrades der Medizin der Medizinischen Fakultät der Universität Ulm vorgelegt durch Helena Kett Ulm 2016 Amtierender Dekan: Herr Prof. Dr. T. Wirth 1. Berichtserstatter: Frau apl. Prof. Dr. K. Döhner 2. Berichtserstatter: Herr apl. Prof. Dr. W. Kratzer Tag der Promotion: 16.12.2016 II FÜR MEINEN VATER III Inhaltsverzeichnis Abkürzungsverzeichnis .......................................................................................... VI 1. Einleitung ............................................................................................................. 1 1.1 Die akute myeloische Leukämie ...................................................................... 1 1.2 Epidemiologie und Ätiologie ............................................................................ 1 1.3 Pathogenese...................................................................................................... 2 1.4 Klassifikation..................................................................................................... 3 1.6 Die akute myeloische Leukämie mit komplexem Karyotyp ........................... 6 1.7 Das Tumorsuppressorgen TP53 ...................................................................... 7 1.7.1 Funktionelle und strukturelle Domänen .............................................................. 8 1.7.2. Der N-Terminus von p53 ................................................................................... 8 1.7.3. Die Prolin-reiche Domäne ................................................................................. 8 1.7.4. Die zentrale DNA-Bindungsdomäne .................................................................. 8 1.7.5. Der C-Terminus von p53 ................................................................................... 9 1.7.6. Funktion............................................................................................................. 9 1.7.7. Mutationen....................................................................................................... 10 1.8 Zielsetzung ...................................................................................................... 12 2. Material und Methoden ..................................................................................... 13 2.1 Patienten und Patientenproben ..................................................................... 13 2.1.1. Zytogenetische und molekulargenetische Charakterisierung ......................... 13 2.2 Therapie ........................................................................................................... 14 2.3 Mutationsanalyse – methodische Grundlagen ............................................. 14 2.4 DNA-Extraktion ............................................................................................... 16 2.5 Herstellung von DNA-Arbeitslösungen und DNA-Platten ........................... 16 2.6 DNA-Amplifikation .......................................................................................... 17 2.6.1. Polymerase-Kettenreaktion (PCR) .................................................................. 17 2.6.2. Auswahl der Polymerase und Oligonukleotide ................................................ 17 2.6.3. Primer .............................................................................................................. 18 2.7 Gelelektrophorese der PCR-Produkte ........................................................... 19 2.8 Zumischung von Wildtyp-DNA ...................................................................... 20 2.9 Heteroduplex ................................................................................................... 21 2.10 Mutationsanalyse am Wave-Gerät ................................................................. 21 2.10.1 Prinzip der Auftrennung von DNA-Fragmenten .............................................. 22 2.10.2 Puffer .............................................................................................................. 24 2.10.3 Analysevorgang .............................................................................................. 25 2.10.4 Sensitivitätsbestimmung ................................................................................. 26 IV 2.11 Auswertung ..................................................................................................... 26 2.12 Amplifikation der selektierten DNA-Fragmente mittels PCR....................... 26 2.12.1 Aufreinigung der PCR-Produkte ..................................................................... 27 2.12.2 Cycle Sequencing Reaction (CSR) ................................................................. 28 2.12.3 Aufreinigung der DNA mittels DyeEx Spin Kit 30 ............................................ 29 2.12.4 Sequenzierung ............................................................................................... 29 2.12.5 Statistische Auswertung ................................................................................. 30 2.12.6 Reagenzien, Chemikalien und Geräte ............................................................ 30 3. Ergebnisse ......................................................................................................... 31 3.1 Inzidenz von TP53-Mutationen ....................................................................... 31 3.2 TP53 Mutationstypen ...................................................................................... 31 3.2.1 Missense Mutationen ...................................................................................... 32 3.2.2 Nonsense Mutationen ..................................................................................... 38 3.2.3 Frame-Shift ..................................................................................................... 39 3.2.4 Intronische Splice-Mutationen ........................................................................ 40 3.3 TP53 Sequenzvarianten .................................................................................. 41 3.4 Exonverteilung der Mutationen ..................................................................... 42 3.5 Exon- und Intronverteilung der Polymorphismen ........................................ 44 3.6 Korrelation des TP53-Status mit klinischen Daten ...................................... 45 3.7 Korrelation des TP53-Status mit genomischen Veränderungen ................ 46 3.8 Überlebensanalysen ....................................................................................... 48 4. Diskussion ......................................................................................................... 50 4.1 Das Tumorsuppressorgen ............................................................................. 50 4.2 Das Mutationsprofil des TP53 ........................................................................ 51 4.3 Mögliche Bedeutung von TP53 Polymorphismen ........................................ 52 4.4 Genetische Instabilität , Assoziation mit chromosomalen Aberrationen... 53 4.5 Klinische und prognostische Bedeutung von TP53 Mutationen bei der AML mit komplexem Karyotyp ............................................................................... 56 4.6 Methode ........................................................................................................... 58 4.7 Ausblick ........................................................................................................... 58 5. Zusammenfassung ............................................................................................ 60 6. Literaturverzeichnis .......................................................................................... 62 7. Anhang ............................................................................................................... 72 8. Lebenslauf.......................................................................................................... 74 V Abkürzungsverzeichnis 100 bp-L. 3' 5` 53kDA A AA Ala AML AML-ETO AMLSG Arg AS Asn Asp ATM BAI 1 BAX BCL2BL-60 BRCA1 C Caspase CBF CCD CEBPA C-kit CN_LOH CR CSR C-terminal Cyklin-CDK Cys dATP dCTP ddNTP dGTP DHPLC DNA dNTP DR 5 dTTP EFS et al. Fas FISH FLT3 FMS FS G G1-Phase G2 GAAD45 GD-AiF 100 Basenpaar-Längenstandard 3`-Ende 5`-Ende 53 kilo Dalton Adenin Aminoacid Alanin akute myeloische Leukämie Eight-twenty-one acute myelogenous leukemia study group Ulm Arginin Aminosäure Asparagin Asparaginsäure Ataxia telangiectasia mutated Brain specific angiogenesis inhibitor 1 proapoptotisches Bcl-2-Homolog B-Cell leukemia/lymphoma= B-ZellLeukämie/Lymphom 2 Burkitt Lymphom Zelllinie Breast Cancer 1 Cytosin Cystein-Aspartat spezifische Protease Core binding factor Charge-Coupled Device CCAAT/enhancer binding protein alpha v-kit Hardy-Zuckerman 4 feline sarcoma viral oncogene homolog Copy number-neutral loss of heterozygoty Complete remission Cycle Sequencing Reaction Carboxy-Terminus Cyclin-dependent Kinase Cystein Desoxyadenosintriphosphat Desoxycytidintriphosphat Didesoxynukleotidtriphosphat Desoxyguanosintriphosphat Denaturating high performance liquid chromatography Desoxyribonukleinsäure Desoxyribonukleotidtriphosphat Death receptor 5 Desoxythymidintriphosphat Event free survival (Ereignisfreies Überleben) et alii, et aliae, et alia F7-associated surface protein Fluoreszenz-in-situ Hybridisierung FMs-related tyrosine kinase 3-Ligand formerly McDonough feline sarcoma viral oncogene homolog Frame Shift Guanin Gap1-Phase Gap2-Phase Growth arrest and DNA damage inducible 45 Glioma- derived angiogenesis inhibitor factor VI Gln Glu Gly HAM HD His HLA HOX HPV HR IARC ICE Ile inv ISCN ITD JAK2 JVM kb kDa KM Leu LOH LSC LW Lys mdm 2 MDM2 MDR MDS Met MgCl MLL mRNA mut MYH11 NA NF1 NOXA NPM1 NR N-terminal OS p21WAF1 p53 PB PCR Phe Pidd PML PRIMA-1 Pro PUMA RAD51 RAF RARα RAS Reprimo RFS Glutamin Glutaminsäure Glycin Hochdosis - Cytarabin+ Mitoxantron Heidelberg Histidin Human leukocyte antigen Homöotisches Gen Humanes Papillomvirus Hochrisiko International Agency for Research on Cancer Idarubicin, Cytarabin, Etopsid Isoleucin invers International System for Cytogenetic Nomenclature Interne Tandemduplikation Januskinase Prolymphozytorische Zelllinie Kilobasenpaar Kilo Dalton Knochenmark Leucin loss of heterozygoty leukämische Stammzelle Leerwert Lysin mouse double minute 2 homolog murine double minute 2 Multidrug Resistance Myelodysplastisches Syndrom Methionin Magnesiumchlorid Mixed lineage Leukemia messenger Ribonukleinsäure mutiert myosin, heavy chain 11 Not available Neurofibromin 1 steht für "damage" Nucleophosmin 1 Niedrigrisiko Amino-Terminus Overall Survival (Gesamtüberleben) Cyclin-dependent Kinase inhibitor 1 Protein 53 peripheres Blut Polymerase chain reaction Phenylalanin P53-induced protein with a death domain Promyelozytenleukämie p53-dependent reactivation and induction of massive apoptosis Prolin p53 upregulated mediator of apoptosis recombinase found in eukaryotes rapidly growing fibrosarcoma retinoic acid receptor rat sarcoma viral oncogene homolog steht für "stop/repress" Relaps free Survival (Rezidivfreies Überleben) VII RITA RUNX1 s-AML SCT SEER Ser SH3 SH3-Domäne SNP SR T TAE t-AML TEAA Ter Thr TKD TNF Trp TSP-1 Tyr UMD Val vs WHO WT WT1 z.B. reactivation of p53 and induction of tumour cell apoptosis) Runt-related transcription factor-1 sekundäre akute myeloische Leukämie stem cell transplantation Surveillance Epidemiology and End Results Serin Src-homology 3 Src-homology 3 - Domäne single-nucleotide-polymorphisms Standardrisiko Thymin Tris-Acetet-EDTA-Puffer Therapie-assoziierte AML Triethylammoniumacetat Stop Threonin Tyrosinkinasedomäne Tumornekrosefaktor Tryptophan Thrombospondin-1 Tyrosin Universal Mutation Database Valin versus World Health Organization Wildtyp Wilms tumour 1 zum Beispiel VIII 1. Einleitung 1.1 Die akute myeloische Leukämie Die akute myeloische Leukämie (AML) ist eine maligne klonale Neoplasie der hämatopoetischen Stammzellen. Sie ist charakterisiert durch eine gesteigerte Zellproliferation der myeloiden Vorläuferzellen im Knochenmark und eine Blockade der Differenzierung dieser Zellen. (Lowenberg,Bob 1999) Sowohl genetisch als auch klinisch stellt sich ein heterogenes Erkrankungsbild dar. Auf molekularer hämatopoetischen Ebene Stammzellen, zeigt sich die die dies in normalen Veränderungen Mechanismen der des Zellwachstums, der Proliferation und der Differenzierung stören. (Frohling,S. 2005) Die klinischen Zeichen der AML sind divers und unspezifisch mit häufig akutem Beginn. Sie resultieren durch die Suppression der normalen Hämatopoese und der Insuffizienz der hämatopoetischen Systeme, und zeigen sich dementsprechend in Form von Anämie, Neutropenie und Thrombozytopenie. (Estey,E. 2006) 1.2 Epidemiologie und Ätiologie Das größte Krebsregister Amerikas, das Surveillance Epidemiology and End Results (SEER), gibt eine altersadjustierte Inzidenz der AML von 3,4/100000 Einwohner an. Diese Inzidenz bezog sich auf die Jahre 2003 – 2007 und war für Männer deutlich höher als für Frauen (4,3 vs. 2,9). Die Unterschiede bezüglich der ethnischen Zugehörigkeit waren mit einer Inzidenz von 3,7 bei weißen Amerikanern im Vergleich zu 2,7 bei Farbigen geringer. Die deutlichsten Inzidenzunterschiede finden sich für das Alter. Mit zunehmendem Alter steigt die Inzidenz der AML deutlich an und beträgt 15,1 bei den 55- bis 64-Jährigen, 19,9 bei den 65- bis 74-Jährigen und 24,5 bei den 75 bis 84-Jährigen. Das mediane Alter bei Diagnosestellung beträgt 67 Jahre, sodass die AML als eine Erkrankung des älteren Menschen angesehen werden muss. Die auslösenden Faktoren für die Erkrankung sind bei der großen Mehrzahl der Patienten nicht bekannt. Chemikalien wie z.B. Benzolverbindungen (Hayes,R.B. 1997) oder ionisierende Strahlung (Curtis,R.E. 1992), (Curtis,R.E. 1985) sind mit einer erhöhten Inzidenz assoziiert worden. Eine mögliche genetische Prädispostion zur Entwicklung der 1 AML wird durch Untersuchungen bei Patienten mit Downsyndrom (Hasle,H. 2000) und der Fanconi-Anämie diskutiert. (Auerbach,A.D. 1991) Auch exogene Ursachen wie die vorherige Behandlung mit zytostatischen Substanzen, insbesondere mit Alkylanzien oder Topoisomerase-II- Inhibitoren sind von Bedeutung. Die therapieassoziierte AML stellt mittlererweile 10-20 % aller Fälle mit AML dar. (Leone,G. 1999) 1.3 Pathogenese Durch die zytogenetische und molekulare Charakterisierung der leukämischen Zellpopulation bei AML Patienten konnten wesentliche Einblicke in die Pathogenese dieser Erkrankung ermöglicht werden und zeigen, dass kritische Veränderungen in Genen, die die Proliferations- und Differenzierungsprogramme in der normalen Hämatopoese steuern ursächlich für die Fehlregulation von Differenzierung und Proliferation des leukämischen Zellklons sind. So wurde nach einem Modell das Zusammenwirken zweier komplementärer Klassen an Mutationen postuliert, die zusammenwirken müssen, um eine akute Leukämie hervorzurufen. (Gilliland,D.G. 2004) Durch die Aktivierung von Signaltransduktionskaskaden der sogenannten Klasse1-Mutationen wird ein Proliferationsvorteil für hämatopoetische Vorläuferzellen geboten. Dazu gehören zum Beispiel FLT3-ITD, KIT und N-RAS. (Gilliland,D.G. 2002) Zu den Mutations-Formen der Klasse-2 gehören die Transkriptionsfaktoren oder Bestandteile des Koaktivierungskomplexes der Transkription, die überwiegend die Zelldifferenzierung beeinträchtigen. Beispiele hierfür sind NPM1Mutationen, HOX Fusionsgene (Dash,A. 2001) MLL Fusionsgene (Gilliland,D.G. 2002) und CEBPA Mutationen. (Pabst,T. 2001) Ein „in vivo“ Mausmodell konnte nachweisen, dass nur durch die Koexistenz von sowohl Klasse-1-, hier FLT3-ITD, als auch Klasse-2-Mutationen, hier das Fusionsgen RUNX1/RUNX1T1, eine akute Leukämie induziert werden konnte, durch eine einzelne Mutation jedoch nicht. (Schessl,C. 2005) Klinisch synergistische Effekte gibt es zwischen der internen Tandemduplikation (ITD) des FLT3-Gens (Klasse-1-Mutation) und NPM1-Mutationen (Klasse-2-Mutation) in der 2 ein günstiger NPM1-Effekt hinsichtlich der Prognose nur in Abwesenheit von FLT3-ITD beobachtet wird. (Dohner,K. 2005) 1.4 Klassifikation Die sogenannte French-American-British – (FAB)-Klassifikation zur Einteilung der AML wurde seit morphologische 1976 Kriterien verwendet. des Sie stützte Knochenmarks. sich hauptsächlich (Bennett,J.M. 1985) auf Diese Klassifikation war im klinischen Alltag sehr gut anwendbar, berücksichtigte jedoch neue diagnostische Verfahren wie Zytogenetik, Genetik Immunphänotypisierung, sowie klinische oder prognostische Aspekte nicht. oder Jene Lücke der FAB-Klassifikation will die seit 2001 vorliegende World-HealthOrganiziation-(WHO-)Klassifikation schließen [World Health Organization Classification of Tumours 2001]. Diese berücksichtigt erstmals zytologenetische und genetische Merkmale, um die AML in biologische Subtypen einzuteilen. Die WHO-Klassifikation wurde mittlerweile mehrfach überarbeitet, zuletzt im Sommer 2008. In dieser rezidivierten Aktualisierung wurden erstmals die beiden molekularen Marker NPM1 und CEBPA Mutation provisorisch zur weiteren Klassifizierung von AML Entitäten mit aufgenommen. 3 WHO-Klassifikation (Swerdlow, SH 2008) AML mit rekurrenten zytogenetischen Aberrationen AML mit balancierten Translokationen v.a. t(8;21)(q22;q22), t(15;17)(q22;q12), inv(16)(p13;q22)/t(16;16)(p13;q22) AML mit Genmutationen AML mit CEBPA Mutation AML mit NPM1 Mutation AML mit multilineärer Dysplasie (mindestens 2 Linien betroffen) AML mit vorausgegangener Myelodysplasie/myeloproliferatives Syndrom De-novo AML ohne vorausgegangenes Syndrom Therapie assoziierte AML und MDS AML/MDS nach Gabe von Alkylanzien AML/MDS nach sonstiger Chemo-/ Strahlentherapie AML/MDS nach Therapie mit Topoisomerase-II-Inhibitoren AML ohne weitere Spezifizierung AML minimal differenziert (FAB M0) AML mit Ausreifung (FAB M2) AML ohne Ausreifung (FAB M1) Akute myelomonozytäre Leukämie (FAB M4) Akute Erythroleukämie (FAB M6) Akute monozytäre Leukämie (FAB M5a, b) Akute Megakaryoblastenleukämie (FAB M7) Akute Basophilenleukämie Akute Panmyelose mit Myelofibrose Myelosarkom/Chlorom Für die Diagnosestellung werden >/= 20% Blasten im Knochenmark und/oder Blut verlangt. 4 1.5 Prognostische Bedeutung chromosomaler und molekulargenetischer Aberrationen bei der AML Die AML ist eine prognostisch sehr heterogene Erkrankung. Eine ganze Reihe klinischer und diagnostischer Kriterien wurden auf ihren prognostischen Einfluss hin untersucht. Aktuell können der Karyotyp, das Alter des Patienten, die Leukozytenzahl bei Diagnose, de novo versus sekundäre oder therapieassoziierte AML, (Lowenberg,B. 2001) sowie das Ansprechen auf die Induktionstherapie (Kern,W. 2004) als relevante prognostische Faktoren betrachtet werden, wovon zytogenetische Aberrationen (Grimwade,D. 1998) den bedeutensten Prognosefaktor darstellen. Mehrere Studien haben gezeigt, dass bestimmte chromosomale Aberrationen auch mit verschieden hohen Raten an kompletten Remissionen und dem Überleben einhergehen. (Byrd JC. Mrozek K. Dodge RK. Carroll AJ. Edwards CG. Arthur DC. Pettenati MJ. Patil SR. Rao KW. Watson MS. Koduru PR. Moore JO. Stone RM. Mayer RJ. Feldman EJ. Davey FR. Schiffer CA. Larson RA. Bloomfield CD. Cancer and Leukemia Group B (CALGB 8461) 2002), (Grimwade, D. 1998), (Mrozek,K. 2001) In Bezug auf das Therapieansprechen und die Gesamtüberlebensdauer erfolgt die Zuordnung in drei zytogenetische Risikogruppen: Niedrigrisiko (NR): t(8;21), inv(16)/t(16;16), t(15;17) Hochrisiko (HR): abn(3q), -5/5q-, t(6;9), -7, komplexer Karyotyp, Standardrisiko (SR): normaler Karyotyp, t(9;11), +8, andere FLT3-Mutation, MDR1-Expression Auch bei AML-Patienten ohne Nachweis von Karyotypanomalien ist die prognostische Heterogenität submikroskopischen beachtlich. genetischen Durch Alterationen die konnten Identifikation mit von modernen molekulargenetischen Untersuchungen eine Vielfalt an genetischen Markern identifiziert werden.(Dohner,K. 2005) Von prognostischem Interesse sind zum Beispiel NPM1- Genmutationen, interne Tandemduplikationen (ITD) des FLT3Gens, CEBPA-Genmutationen, sowie partielle Tandemduplikationen des MLL- Gens. (Schlenk,R.F. 2008) Mehrere Studien zeigen einheitlich, dass CEBPA5 Mutationen mit einer günstigen (Frohling,S. 2004), FLT3-ITD- Mutation dagegen mit einer ungünstigen Prognose hinsichtlich Rezidivfreien- und Gesamtüberleben assoziiert sind. (Frohling S. Skelin S. Liebisch C. Scholl C. Schlenk RF. Dohner H. Dohner K. Acute Myeloid Leukemia Study Group,Ulm 2002). Ein besseres Ansprechen auf die Induktionstherapie und ein längeres Rezidivfreies Überleben zeigen dagegen Patienten mit Mutationen im NPM1- Gen. Allerdings wurde dieser vorteilhafte Effekt nur in Abwesenheit einer FLT3-ITD Mutation beobachtet. (Dohner,K. 2005) Für einen tieferen Einblick in die Entwicklung der AML und die Etablierung molekularzielgerichteter Therapie ist es von grosser Bedeutung, mit Hilfe molekulargenetischer Methoden neue molekulare Marker zu identifizieren und diese in großen, multizentrischen Studien auf klinische und prognostische Relevanz hin zu testen. 1.6 Die akute myeloische Leukämie mit komplexem Karyotyp Zeigt eine AML mindestens drei strukturelle und/oder numerische Aberrationen, spricht man von einer AML mit komplexem Karyotyp. (Schoch,C. 2001) Häufig beinhalten die komplex aberranten Karyotypen Veränderungen an den Chromosomen 5 und 7, jedoch ist die Definition der Gruppe mit komplex aberranten Karyotyp nicht immer einheitlich und trägt zur Heterogenität dieser AML-Subgruppe bei. De novo beträgt dabei die Häufigkeit 10-15 % (Slovak, M.L. 2000), bei der sekundären AML 25 % und 26,9 % bei der therapieassoziierten AML (t-AML). (Schoch,C. 2005) Einige Studien befassten sich mit der Assoziation von einer Alkylanzientherapie mit unbalancierten Veränderungen, insbesondere an den Chromosomen 5 und 7 (Andersen, M.K. 1998), (Pedersen-Bjergaard,J. 1994), ohne jedoch Einblicke in die Mechanismen der Entstehung dieser Alterationen zu geben. Veränderungen an den Chromosomen 5 und 7 zählen zu den häufigsten Veränderungen, die innerhalb eines komplexen Karyotyps vorliegen. Das Vorhandensein einer Veränderung am Chromosom 5 oder am Chromosom 7 ist mit einer schlechten Prognose assoziiert und stratifiziert die Patienten in die Hoch-Risiko-Gruppe. 6 Eine wichtige Rolle innerhalb AML mit komplexem Karyotyp könnte das TP53 spielen, das eine Schlüsselrolle im Rahmen des Apoptosemechanismus darstellt. Es wurde im Zusammenhang mit der AML mit komplexem Karyotyp in bis zu 78 % der Fälle nachgewiesen. (Schoch, C. 2005), (Haferlach,C. 2008) Auch traten diese Mutationen gehäuft bei Patienten mit sekundärer AML auf und könnten als Grundlage für die hier klinisch häufig beobachtete Therapierefraktivität angesehen werden. (Castro, P.D. 2000), (Christiansen, D.H. 2001) Auffällig ist ebenso die häufige Assoziation von 17p13-Deletionen, die den p53-Locus beinhalten (~60%). (Rucker,F.G. 2006) Im Vergleich zur AML mit balancierten Chromosomenanomalien steigt die Inzidenz der AML mit komplexem Karyotyp altersabhängig an. (Schoch, C. 2001) und ist mit einer äußerst schlechten Prognose assoziiert. Die Rate an „Complete Remission“ (CR) liegt mit 14-56 % sehr niedrig und trägt entscheidend zu dem mit einem Median von 2,5-7 Monaten Überleben dieser Patienten bei. (Frohling,S. 2006) Inwieweit kumulierende genetische Alterationen und somit eine genetische und chromosomale Instabilität diesem Verhalten zugrunde liegt, werden künftige Untersuchungen klarstellen. Auch gab es bislang keine Arbeit, in der im Rahmen einer homogen behandelten Studienpopulation der p53- Mutationsstatus evaluiert wurde und die molekularen Daten mit klinischen Charakterstika korreliert wurde. 1.7 Das Tumorsuppressorgen TP53 Seit seiner Erstbeschreibung von LANE u. CRAWFORD (1979) (Lane, D.P. 1979) ist p53 ein intensiv untersuchtes Protein und wird als weitaus bedeutendster Tumorsuppressor angesehen. Verlust oder Mutation des Proteins führen zu einer höheren Tumoranfälligkeit. Im DNA- Schädigungssignalweg spielen Sensoren, Signaltransduktoren und – effektoren eine wichtige Rolle und vermitteln als ein verzahntes Regelwerk die Antwort auf eine DNA-Schädigung. Ein gutes Beispiel ist das Protein 53, welches im Bezug auf den Zellzyklusarrest, Apoptose und genetische Stabilität einen zentralen Platz einnimmt. (Kirsch,D.G. 1998) 7 1.7.1 Funktionelle und strukturelle Domänen P53 ist ein zelluläres Protein, das sich aus 393 Aminosäuren zusammensetzt und dessen Molekulargewicht ca. 53kDa beträgt. Das kodierende Gen liegt auf dem kurzen Arm des Chromosoms 17 und enthält strukturell fünf evolutionär konservierte Bereiche, die als Domänen 1-5 gekennzeichnet werden. (Soussi,T. 1990) Die Domäne 1 befindet sich im N-terminalen Bereich, die Domäne 2-5 in der zentralen Region von p53. Funktionell kann man p53 in vier verschiedene Domänen unterteilen: Transaktivierungsdomäne AS 1-42 Zentrale DNA – Bindungsdomäne AS 102-292 Prolinreiche Domäne AS 61-94 Oligomerisierungsdomäne AS 363 – 393 1.7.2. Der N-Terminus von p53 Der aminoterminale Bereich des Proteins wird aus den Aminosäuren 1-42 gebildet und enthält die Transaktivierungsdomäne. (Unger,T. 1992) Diese Domäne kann mit Transkriptionsfaktoren interagieren und somit die Transkription ermöglichen. Finden sich in diesen Bereichen Mutationen, so ist p53 nicht mehr in der Lage spezifische Zielgene zu aktivieren, aber auch Ausfälle bezüglich der Repression von Promotoren und der Induktion zur Apoptose werden beobachtet. (Roemer,K. 1996) 1.7.3. Die Prolin-reiche Domäne Diese Domäne reguliert die Induktion der Apoptose und wird von Proteinen erkannt, die eine SH3-Domäne besitzen. (Sakamuro,D. 1997) 1.7.4. Die zentrale DNA-Bindungsdomäne Die zentrale Region enthält die DNA-Bindungsdomäne und wird von den Aminosäuren 102-292 gebildet. Es enthält evolutionär hochkonservierte Regionen, die hohe Sequenzhomologien mit anderen Spezies aufweisen. Hier lokalisieren über 90% der TP53-Mutationen. (el-Deiry,W.S. 1992), (Hainaut,P. 1998) 8 1.7.5. Der C-Terminus von p53 Die Aminosäuren 363-393 bilden das C-terminale Ende und eine regulatorische Domäne. Durch Phosphorylierung und Acetylierung wird p53 stabilisiert und aktiviert und dient als negativer Regulator der Bindung zwischen p53 und der Ziel-DNA. (Hupp,T.R. 1993) Durch die Interaktion des C-Terminus mit einer anderen Region des p53-Moleküls wird der Zustand des Proteins inaktiviert und eine Bindung mit der Ziel-DNA verhindert. 1.7.6. Funktion Das vielfach als „Wächter des Genoms“ bezeichnete Protein (Lane,D.P. 1992) reguliert die Expression von Genen, die in verschiedenen zellulären Funktionen involviert sind, wie Zellzyklusarrest, DNA – Reparatur, Seneszenz und Apoptose (Vogelstein,B. 2000) aber auch Zellmotilität,- adhäsion, Antiangiogenese und Antimetastasierung. (Harms,K. 2004), (Wei,C.L. 2006) Untersuchungen über die p53-DNA-Bindungsstellen legen nahe, dass es mehr als 200-400 p53 Zielgene gibt. (el-Deiry,W.S. 1998) Mit der Expression und Repression verschiedener Zielgene antwortet p53 auf Stress und aktiviert oder reprimiert bestimmte Gene, um damit einen funktionellen Erfolg zu erzielen. (Liu,G. 2006) Hierbei bindet p53 an die DNA-Bindungsdomäne Promotorbereich des jeweiligen von p53-responsiven Zielgens und induziert Elementen so die im basale Transkriptionsmaschinerie, Co-Aktivatoren und Co-Repressoren. (Espinosa,J.M. 2003) P53 aktiviert Gene mit den unterschiedlichsten Funktionen. Eine der beiden wichtigsten Funktionen sind der Zellzyklusarrest und die Apoptose. Der normale Zellzyklus beinhaltet vier Phasen: die Mitose, S-Phase, G1-Phase (zwischen Mitose und S-Phase) und die G2-Phase (zwischen S-Phase und Mitose). p21WAF1 unterbricht den Zellzyklus in der G1- und der G2-Phase, (el-Deiry, W.S. 1993), (Zhan,Q. 1999) indem es nach DNA-Schäden durch wtp53, nicht aber durch mutiertes p53 transaktiviert wird. (el-Deiry, W.S. 1993) und dann an Cyklin-CDKKomplexe (Cyclin-dependent kinase) bindet und deren Kinaseaktivität hemmt. Weitere Proteine, die den Zellzyklus in der G2-Phase unterbinden, sind Reprimo, 14-3-3sigma und GADD 45. (Laronga, C. 2000), (Ohki,R. 2000), (Zhan,Q. 1999) 9 Die Apoptose, auch programmierter Zelltod genannt, kann ebenfalls durch p53 Zielgene ausgeführt werden. Eine Beteiligung zeigt hierbei zum Beispiel Bax, ein Protein der Bcl2-Familie, das an den Mitochondrien lokalisiert ist und als erster Mediator der p53-abhängigen Apoptose identifiziert wurde. (Miyashita,T. 1995) Bax begünstigt eine Cytochrom-c-Freisetzung, was wiederum zu einer Aktivierung der Caspase 9 führt und damit die Caspase-Kaskade auslöst, die in Apoptose endet. (Srinivasula,S.M. 1998) Gene, wie puma und noxa gehören ebenfalls zur BCL2-Familie und werden durch p53 transaktiviert. (Nakano,K. 2001), (Oda,E. 2000) Zusätzlich werden auch die Todesrezeptoren DR5 und Fas, Tumornekrosefaktor (TNF) und Pidd durch p53 aktiviert und provozieren die Apoptose. (Owen-Schaub,L.B. 1995), (Vogelstein,B. 2000), (Lin,Y. 2000) Auch in der Neubildung von Blutgefäßen für das Tumorwachstum ist p53 involviert, indem es durch die Aktivierung von Proteinen wie Thrombospondin1 (TSP-1), BAI1 oder GD-AiF eine Hemmung der Angiogenese bewirkt und dadurch die malignen Zellen nicht mit Nährstoffen versorgt werden können. (el-Deiry,W.S. 1998) Eine besondere Rolle spielt das Gen MDM2, da es durch p53 transaktiviert wird und zugleich ein p53-Inhibitor ist. So transaktiviert p53 MDM2, welches daraufhin im Kern an p53 bindet. (Vogelstein,B. 2000) 1.7.7. Mutationen Studien konnten belegen, dass TP53 das am häufigsten mutierte Gen bei malignen Erkrankungen ist, in mehr als 50% aller humanen Tumoren liegt es mutiert vor. (Vogelstein,B. 2000) Die meisten Tumorsuppressoren werden durch Frameshift oder Nonsense-Mutationen inaktiviert, während sich bei TP53 ca. 80% Missense-Mutationen finden, die wiederum zu 97 % in der DNA – Bindungsdomäne liegen. (Weisz,L. 2007) Diese Missense Mutationen führen zu einer Anhäufung des mutierten p53-Proteins, so dass mutiertes p53-Protein in Tumoren sehr viel höher exprimiert als wtp53 in gesunden Zellen. Nach Angaben der International Agency for Research on Cancer (IARC) finden sich die häufigsten TP53-Mutationen in Ovarial-, und Colorektaltumoren sowie in Tumoren des Kopf-Hals Bereichs. [siehe Abbildung 1] In der AML sind TP53-Mutationen mit <5% relativ selten, wohingegen diese in der AML mit komplexem Karyotyp gehäuft auftreten. (~70%) (Schoch,C. 2005) 10 Abbildung 1: Prävalenz von TP53 Mutationen in unterschiedlichen Tumoren entsprechend der internationalen Datenbank der Krebsforschung (Olivier,M. 2002) Die Datenbank für Mutationen im TP53 gibt außerdem auch Auskunft über die Häufigkeitsverteilung aller bisher bekannten TP53-Mutationen und zeigt eine Konzentrierung in sechs Codons 175, 245, 248, 249, 273 und 282, welche deshalb auch als „Hot Spot“-Regionen bezeichnet werden und über 40 % aller TP53-Mutationen ausmachen. (60 Olivier,M. 2002), (Weisz,L. 2007) Die Mutationen können zwei Hauptklassen zugeordnet werden. Die Klasse-1Mutanten, auch Kontaktmutanten genannt, führen durch die Änderung von Aminosäuren zu einer Störung der DNA Bindung, ohne jedoch die Konformation von p53 zu ändern. Klasse-2-Mutanten werden als Konformationsmutanten bezeichnet und haben durch Punktmutationen in dem Maße die ursprüngliche p53- Konformation verloren, so dass eine DNA-Bindung nicht mehr möglich ist. (Cho, Y. 1994), (Prives, C. 1994), (Roemer,K. 1999) Umso mehr Veränderungen der wt-Konformation durch die Mutation erfolgt sind, desto weniger wtp53 Eigenschaften sind noch vorhanden. (Weisz,L. 2007) Wtp53 hemmt normalerweise das Wachstum von Tumorzellen. Viele Daten der letzten Jahre konnten dem mutierten p53 unterschiedliche Funktionen in der Kanzerogenese zuweisen, dem „Loss of function – gain of function“ bzw. dem dominant negativen 11 Effekt. Ist TP53 mutiert, führt dies zum totalen Verlust der p53-Funktion (Loss of function) oder zum sogenannten Funktionsgewinn (Gain of function). Dies fördert die Ausbildung von Tumoren, z.B. durch die transkriptionelle Aktivierung von Genen, die in Zellproliferation, Zellüberleben und Angiogenese involviert sind und ein verstärktes tumorigenes Potential mit erhöhter Resistenz gegenüber Chemotherapie und Bestrahlung besitzen. Beim dominant negativen Effekt kommt es bei gleichzeitiger Expression von mutp53 und wt53 zur Inhibierung der Wildtyp- Form und ihrer Tumorsuppressorfunktion. (Weisz,L. 2007) Maus-Modelle, die die TP53 Inaktivierung untersuchten, identifizierten Funktionsgewinne für Hot-Spot Mutationen wie R175H, R248W und R273H so wie eine gesteigerte Proliferation, eine beschleunigte Tumorgenese und Leukämogenese die eine aggressivere AML zur Folge hatten. (Song,H.2007), (Donehower, LA.2009), (Zuber, J. 2009) Einige Arbeitsgruppen beschäftigten sich mit der Erhebung der TP53 Inzidenzen, sowie der Analyse chromosomaler Aberrationen bei der AML, insbesondere auch bei der AML mit komplexem Karyotyp, (Haferlach, C. 2008), (Schoch,C. 2005), (Bowen, D.2009). Bislang gibt es jedoch keine Arbeit, in der im Rahmen einer homogen behandelten Studienpopulation der P53-Mutationsstatus systematisch evaluiert und die molekularen Daten mit klinischen Charakteristika korreliert wurden. 1.8 Zielsetzung In der vorliegenden Arbeit wurden erwachsene AML-Patienten mit komplexem Karyotyp (n=234), die im Rahmen der konsekutiven, multizentrischen Behandlungsstudien AMLHD93, AMLHD98A, AMLHD98B, AMLSG07/04 und AMLSG06/04 der AML Studiengruppe (AMLSG) behandelt wurden, mit Hilfe molekulargenetischer Analysen auf Mutationen in TP53 untersucht. Ziel war es, die Inzidenzen von TP53-Mutationen an einer großen, möglichst homogenen AML-Kohorte zu bestimmen. Geprüft werden sollte auch, inwieweit TP53Mutationen mit bestimmten chromosomalen und/oder genetischen Veränderungen sowie klinischen Charakteristika assoziiert sind. Darüber hinaus sollte in dieser genetisch und klinisch sehr gut charakterisierten Patientenkohorte die prognostische Bedeutung der TP53 Mutationen innerhalb der AML mit komplexem Karyotyp evaluiert werden. 12 2. Material und Methoden 2.1 Patienten und Patientenproben Zur Analyse des TP53 wurden 234 erwachsene Patienten mit akuter myeloischer Leukämie und komplexem Karyotyp untersucht. Für die jüngeren Patienten wurde ein Altersrahmen zwischen 18 bis 60 Jahre festgelegt, in den Studien für Ältere (AMLHD98B und AML0604) war das Einschlusskriterium mit einem „Alter > 60 Jahre“ definiert. Die zur Analyse notwendige genomische Desoxyribonukleinsäure (DNA) wurde entweder aus leukämischen Knochenmark- oder leukämischen peripheren Blutzellen der Patienten mittels Ficoll-Dichtezentrifugation isoliert. Der Großteil der Patienten wurde im Rahmen von prospektiven, multizentrischen Therapiestudien behandelt [AMLHD98A (n= 34), AMLHD98B (n=29) und AMLSG 0604 (n=44), AMLSG0704 (n=67). Außerhalb klinischer Studien wurden 60 Patienten behandelt. Insgesamt hatten 72 Patienten eine de-novo-AML, 22 Patienten eine sekundäre AML (s-AML) nach einem myelodysplastischem Syndrom und 14 Patienten eine therapie-assoziierte AML (t-AML) infolge der Behandlung eines Primärmalignoms. In allen Studien wurden morphologische, zytochemische und immunphänotypische Analysen durchgeführt und anhand der Kriterien der French-American-British Cooperative Group (FAB) (Bennett, J.M. 1985) und ab 2004 anhand der WHO-Kriterien (Vardiman,J.W. 2009) beurteilt. Alle Patienten wurden über die Therapie, die jeweiligen Studienmodalitäten, die molekulare Analyse der Knochenmark- bzw. Blutproben und die Konservierung dieser Proben aufgeklärt und gaben ihr Einverständis gemäß der Deklaration von Helsinki. 2.1.1. Zytogenetische und molekulargenetische Charakterisierung Zur zytogenetischen und molekularen Diagnostik wurde zum Zeitpunkt der Diagnosestellung den Patienten heparinisierte KM-und PB-Proben (im Verhältnis Heparin-Na:KM zytogenetischen 1:10 und bzw. Heparin-Na:Blut molekularen 1:20) Diagnostik an entnommen die und zur teilnehmenden Referenzlabore gesandt. (ULM, Innere III und Hannover (Prof. Ganser)) Der Karyotyp anschliessend mittels konventioneller Chromosomenbänderung ermittelt und gemäß den Richtlinen des International System for Cytogenetic Nomenclature (ISCN) beschrieben. Zusätzlich kam eine „Fluoreszenz-in-situ-Hybridisierung“ 13 (FISH) mit einem Sondensatz für die Detektion chromosomaler Aberrationen zum Einsatz. (Frohling S. Skelin S. Liebisch C. Scholl C. Schlenk RF. Dohner H. Dohner K. Acute Myeloid Leukemia Study Group,Ulm 2002), (Rucker,F.G. 2012) Im Rahmen von weiteren Forschungsprojekten wurden der Mutationsstatus für die AML-assoziierten Marker FLT3-ITD, FLT3-TKD, NPM1 und CEBPA bestimmt. 2.2 Therapie Die Grundprinzip der Behandlung der jüngeren AML-Patienten (AMLHD98A und AML0704) aber auch der älteren AML-Patienten (AMLHD98B und AMLSG0604) beruht auf dem Prinzip der intensiven Doppelinduktion, gefolgt von einer risikoadaptierten Konsolidierungstherapie, wobei die Intensität der Therapie bei den älteren Patienten geringer war. Hierbei bestand die Doppelinduktionstherapie aus zwei Zyklen ICE (Idarubicin, Cytarabin, Etopsid), falls nach dem ersten ICEZyklus mindestens eine partielle Remission (PR; Blastenanteil im KM <50% des initialen Befundes) vorlag. Auf den ersten Induktionszyklus erhielten refraktäre Patienten einen Hochdosis-AraC-basierten zweiten Induktionszyklus. Nach Erreichen einer kompletten Remission (CR; <5% Blasten im KM) nach Doppelinduktion entsprechend erfolgte des die Risikoprofils protokollgemäße mit entweder Konsolidierungstherapie Hochdosis-AraC-basierten Konsolidierungszyklen oder einer Spätkonsolidierung mit einer autologen oder bei Existenz eines HLA-identen Familienspenders einer allogenen Stammzelltransplantation (SCT). Auch im Falle einer refraktären Erkrankung oder einem nicht-Ansprechen wurde bei den jüngeren Patienten in der Regel eine allogene Stammzelltransplantation durchgeführt. Bei den älteren Patienten bestand die Konsolidierungstherapie aus einer Chemotherapie, das Konzept der allogenen SCT wurde nur in Ausnahmefällen verfolgt. 2.3 Mutationsanalyse – methodische Grundlagen Die Analyse von Sequenzvarianten kann nicht nur zur Identifizierung menschlicher Krankheitsgene eingesetzt werden. (Onuchic,L. F. 2002) Auch finden sich Anwendungsmöglichkeiten in der Populationsgenetik und Evolutionsforschung. Mittlererweile werden von unzähligen klinischen Laboratorien weltweit basierend auf Mutationsnachweis direkte Genotypisierung von hereditären Krankheiten angeboten, um so die Basis genetischer Beratung und Diagnostik zu schaffen.Die 14 verschiedenen Techniken zur Mutationsdetektion unterscheiden sich zum Teil erheblich in Sensitivität und Spezifität. Auch richtet sich die Auswahl der geeigneten Methode nach dem zu untersuchenden Gen und dessen Struktur. Wir untersuchten alle Patienten mit der „denaturating high performance liquid chromatography“(DHPLC, denaturierende Hochdruckflüssigkeitschromatographie) und sequenzierten auffällige Sequenzen im Anschluss direkt. Um der oben beschriebenen Fragestellung nachzugehen wurden folgende Methoden mit genomischer DNA etabliert: DNA- Extraktion Amplifikation der Hot-Spot tragenden Exons 4 bis 10 des TP53 mittels PCR Aufreinigung der DNA- Abschnitte Analyse der DNA- Abschnitte mittels DHPLC direkte Sequenzierung der positiven Analysen Diese Methoden werden nun ausführlich erläutert. 15 2.4 DNA-Extraktion den mononukleären Mittels DNAzol-Lösung (Fa. Gibco BRL, Eggenstein) wurde die DNA sowohl aus Knochenmarkzellen, mononukleären Blutzellen extrahiert. als auch aus den peripheren Für die Extraktion wurde zunächst 2ml DNAzol-Lösung mit diesen Zellen gemischt, um eine Zellwandlyse zu erreichen. Danach wurde zweimal nacheinander je 1ml 100% Ethanol (Fa. RdH, Seelze) dazugegeben und der Behälter geschwenkt. Die angefertigte Lösung wurde in 2 Eppendorf-Behälter gefüllt und 15 Minuten bei 13.000 Umdrehungen pro Minute und 4°C zentrifugiert (Heraeus Biofuge Pico, Fa. Kendro Laboratory Products GmbH, Langenselbold). Nachdem der Überstand vorsichtig abgekippt wurde, wurde 95% Ethanol auf das Pellet gegeben und 10 Minuten bei 13.000 Umdrehungen pro Minute und 4°C zentrifugiert. Der Überstand wurde wieder abgekippt. Dieser letzte Schritt wurde noch einmal wiederholt und danach der Eppendorf-Behälter für 5 Minuten kopfüber zum Trocknen aufgestellt. Nachdem das Pellet getrocknet war, wurde es mit 100µl Wasser für Infusionslösungen resuspendiert und über Nacht bei 37°C geschüttelt (Thermomixer 5437, Fa. Eppendorf, Hamburg). Am nächsten Tag wurde in jedes Eppendorf-Gefäß je 5µl RNAse A (Familie Roche Diagnostics, Mannheim) gegeben. Die Behälter wurden 3 Minuten bei 13.000 Umdrehungen pro Minute zentrifugiert und danach für eine halbe Stunde auf den 37°C warmen Heizblock (QBT2, Fa. Grant Instruments Ltd., Cambridge, Großbritannien) gestellt, um so einen Verdau von RNA-Fragmenten zu erreichen. Nach einer erneuten kurzen Zentrifugation wurde die Lösung mit einer Pipette durchmischt und nochmals zentrifugiert. Nach photometrischer Konzentrationsmessung (UV-Photometer Ultrospec III, Fa. Pharmacia, Uppsala, Schweden) wurde die DNA-Lösung bei -20°C aufbewahrt. 2.5 Herstellung von DNA-Arbeitslösungen und DNA-Platten Zunächst wurden die Stocklösungen der DNA-Proben auf eine Konzentration von 25 Nanogramm DNA/µl auf einen 30 µl Ansatz verdünnt, um in einem späteren Schritt der Untersuchungsreihe bei der Auftrennung der DNA-Fragmente mittels „denaturing high performance liquid chromatography“ (DHPLC; denaturierende Hochdruckflüssigkeitschromatographie) eine ideale DNA- Konzentration der 16 Fragmente zu erhalten. Je 15 µl der DNA der 25 ng/µl Arbeitslösungen wurden in eine 96-well Platte pipettiert und als sog.Geber-Platte benannt. Zum Nachweis homo- und heterozygoter Mutationen sowie zur Erleichternung der späteren Auswertung der Chromatogramme wurden folgende Zelllinien in gleicher Konzentration analog der Patienten-DNA immer mitgeführt: BL-60, Daudi, MHH, Jurkat, Granta, Hela. Diese helfen durch ihr spezifisches Mutationsmuster be kannte Polymorphismen zu detektieren und Mutationen zu erkennen. Gemäss der International Agency for Research on Cancer (IARC) liegen für diese Zell-Linien Angaben über das TP53 Mutationsprofil vor: Hela, Granta, MHH, JVM, BL-60: Wildtyp Jurkat: Mutation in Exon 7: T256A Daudi: Mutation in Exon 8: G266E Namalwa: Mutation in Exon 7: R248Q Auch wurde eine Positivkontrolle der DNA-Arbeitslösung als Leerwert mitgeführt. Diese Geber-Platten wurden verwendet um anschließend mit Hilfe eines Pipettierroboters 1,2 µl DNA (für einen 30 µl PCR-Ansatz) in die zuvor mit 10 µl Wasser vorgelegten Platten zu pipettieren und bei – 20 °C als Nehmerplatten gelagert, sodass zu einem späteren Zeitpunkt im Rahmen der DNA- Amplifikation nur noch die Hinzugabe des Mastermixes nötig war. 2.6 DNA-Amplifikation 2.6.1. Polymerase-Kettenreaktion (PCR) 2.6.2. Auswahl der Polymerase und Oligonukleotide Nach Angaben des Herstellers des im Rahmen dieser Arbeit verwendeten DHPLC-Gerätes empfiehlt es sich eine DNA-Polymerase mit 3´- 5‘ Exonukleaseaktivität (proof reading) zu verwenden, wie beispielsweise die Discoverase Polymerase der Firma Invitrogen. Durch diese wird während der Amplifikation die Anzahl an falsch eingebauten Nukleotiden verringert und ermöglicht dadurch eine Steigerung der Amplifikationsgenauigkeit von einfachen und komplexeren DNA-Fragmenten.Zusätzlich wurde eine dNTP-Stocklösung mit einer Konzentration von 10 mM je dNTP verwendet. Hierfür wurden zu 60µl Aqua 17 destillata je 10µl dATP, dTTP, dGTP und dCTP in der Konzentration 100 mM zugegeben.Alle Lösungen wurden bei –20°C aufbewahrt. 2.6.3. Primer Ebenso wurden Gebrauchslösungen der Primer und Aqua destillata eingesetzt.[1:10 Verdünnung aus Stocklösungen (450 µl Aqua Braun + 50 µl Primerstocklösung)] Wir beschränkten uns auf die Analyse von insgesamt 6 DNA – Abschnitten: Exon 4 (411 Basenpaare) , Exon 5 (422 Basenpaare), Exon 6 (411 Basenpaare), Exon 7 (332 Basenpaare), Exon 8 und 9 (411 Basenpaare) und Exon 10 (475 Basenpaare), die als „Hot-Spot-Region“ des P53 bekannt sind, da dort die meisten Mutationen lokalisiert sind. Die genauen Sequenzen der intronspezifischen Primer sind Tabellen zu entnehmen, wobei diese in Anlehnung an Smith et al. und Gross et al. (für Exon 8/9) und an die „Hot-Spot -Region“ des P53 (Exon 4- Exon 10) ausgewählt und anhand eines anderen Kollektives mit bekannten Mutationen am DHPLC-Gerät optimiert wurden. Tabelle 1: Primerauswahl Exon bp 4 Smith 411 6 Smith 411 5 Smith 422 7 Smith 332 8/9 Gross 411 10 Smith 475 5’Primer (forward) 3’Primer (reverse) ctt cct gaa aac aac gtt ctg gag atc aca cat taa gtg gg cca cca tga gcg ctg ctc ag ccc tta gcc tcg taa gct tc cta gct cgc tag tgg gtt gc cac tcg gat aag atg ctg ag gcc tcc cct gct tgc cac ag ggg agc agt aag gag att cc cga gag tga gac ccc atc tc tgg tta tag gat tca acc gg ttc ctt act gcc tct tgc tt aga aaa cgg cat ttt gag tg 2.6.4 Reaktionsansatz 30 µl - 1,2 µl DNA (25ng/µl) - 2,4 µl dNTP (Desoxyribonukleinstriphosphate, je 10mM dNTPs) - 6 µl PCR-Puffer 1,2 µl Primer forward 1,2 µl Primer reverse 0,6 µl Hot Star Taq DNA Polymerase (2,5U) 7,4 µl Aqua destillata 18 Bei Exon 10 wurden nur 0,3µl Primer bei einem 30µl-Ansatz verwendet, Exon 6 wurde zusätzlich MgSO4 zugeführt. Die PCR-Bedingungen richteten sich nach Standardprotokollen. Initiale Denaturierung 30 Sekunden 94 °C Denaturierung 30 Sekunden 94 °C Initiale Denaturierung 30 Sekunden 94 ° C 5 Minuten 68 ° C Primerhybridisierung 1 Minute Polymerisation 1 Minute Abkühlung Unendlich Extension 68 °C 68 ° C 4°C Exon Hybridisierungstemperatur Zyklen 6 Smith 62 ° C 5 Smith 7 Smith 8/9 Granta 10 Smith 2.7 62 ° C 28 64 ° C 30 35 58 ° C 33 55 ° C 33 Gelelektrophorese der PCR-Produkte Die Überprüfung der Produktgewinnung erfolgt mittels Gelelektrophorese. Dabei werden je 5 µl des PCR-Produktes mit 2 µl Gel-Loading-Buffer (10x Blue-Juice) und 5 µl Wasser gemischt und neben 6 µl des 1kb DNA-ladders gegeben. Der 100 bp DNA-ladder ist eine Kontrolllösung mit 15 blunt-ended Fragmenten von 100 bp bis 1500 bp Länge (Vielfache von 100) und einem zusätzlichen Fragment von 2072 bp Länge. Diese Lösung dient als Anhalt dafür, wie lang die zu untersuchenden DNA-Fragmente sind. Die Gele werden mittels Aufkochen von Agarose mit TAE-Puffer hergestellt. Die elektrophoretische Auftrennung der DNA – Fragmente erfolgt dann bei bei 175 V, 500 mA und 150 Watt über 30 Minuten. Dabei wandern die amplifizierten Nukleinsäuren aufgrund ihrer negativ geladenen Phosphatgruppen im elektrischen Feld auf die positive Elektrode zu. Anschließend wird die charakteristische Bandenordnung der vervielfältigten DNA- Fragmente 19 durch 10-minütiges Einlegen des Agarose-Gels in eine Ethidiumbromid Lösung (10mg/ml) und danach unter Exposition von UV-Licht (UV-Kamera) sichtbar gemacht und mithilfe des Transilluminator 8000-Systems (Fa. Stratagene, Amsterdam, Niederlande) fotographisch dokumentiert. Abbildung 2: Aufgetragenes Gelbild gelelektrophoretisch separierter DNA-Fragmente nach der Polymerase-Kettenreaktion. Hierbei stellt sich bei allen zehn Proben in Höhe von XX der Wildtyp dar (untere Bande durch Pfeil markiert). Abkürzungen: 100 bpL.: 100 Basenpaar-Längenstandard; P1-P10: Probe 1- Probe 10; LW: Leerwert 2.8 Zumischung von Wildtyp-DNA Der amplifizierten Patienten-DNA wurde 20% Wildtyp-DNA hinzugefügt, da bei homozygoten Anlageträgern ansonsten keine Heteroduplexbildung stattfindet und diese somit nicht detektiert würden. Je nach amplifziertem Exon wurde die DNA der Zelllinie Granta oder BL-60 der PCR zugemischt, die in den jeweiligen Exons typische Polymorphismen enthalten, was die anschliessende Auswertung der Chromatogramme erleichterte. Der PCR-Ansatz wurde wie unter Punkt 2.9.4 beschrieben pipettiert, meist wurde für die Wildtyp-PCR ein 100 µl PCR-Ansatz gemacht. Wildtypzellinien für die Zumischung: Exon 4 Granta Exon 5 Granta Exon 7 Granta Exon 6 Exon 8/9 Exon 10 BL-60 BL-60 BL-60 Bei gleich starken PCR-Produkten von Patienten-PCR und Wildtyp-PCR gilt folgendes Mischungsverhältnis: 20 Zumischung 20 %: Patienten-PCR/ Wildtyp-PCR 25 µl/5 µl Patienten-PCR/ Wildtyp-PCR 30 µl/6 µl Patienten-PCR/ Wildtyp-PCR 45 µl/9 µl Patienten-PCR/ Wildtyp-PCR 50 µl/10 µl 2.9 Heteroduplex Die PCR-Produkte mit Wildtyp-DNA Zumischung wurden vor der DHPLC-Analyse einer Heteroduplex-Formierung mit Hilfe einer PCR-Maschine zugeführt. Zunächst erfolgte die Denaturierung der DNA bei 95°C und anschliessend eine langsame, schrittweise Abkühlung bis auf 45°C. Dies ermöglichte die Entstehung von Heteroduplices und Homoduplices. Diese konnten bis zum Lauf in der DHPLCAnlayse 1-2 Wochen bei 4°C aufbewahrt werden. PCR-Programm der Heteroduplex-Formierung Initiale Denaturierung Schrittweise Abkühlung in 25 Zyklen Extension Abkühlung 2 Minuten 10 Sek- 2 °C/Zyklus 95 ° C 94 ° C 30 Minuten 45 ° C Unendlich 4°C 2.10 Mutationsanalyse am Wave-Gerät Bei der denaturierenden Hochdruckflüssigkeitschromatotographie handelt es sich um ein Verfahren, das der Mutationsdetektion dient. Grundsätzlich besteht das Gerät aus einem Autosampler mit zwei 96-well-Platten, einem Ofen, einer Chromatographie-Säule und einem UV-Detektor. Bei diesem Modell (Wave TM 3500 HAT System) handelt es sich um ein Hochdurchsatz-Gerät (high-throughput, HT) und es verfügt zusätzlich über einen Fragment Kollektor. 21 2.10.1 Prinzip der Auftrennung von DNA-Fragmenten Die denaturierende Hochdruckflüssigkeitschromatographie-Technik mit Hilfe des WAVE- Systems beruht im Wesentlichen auf der Unterscheidung von Homo-und Heteroduplices von mindestens zwei DNA-Fragmenten. Durch die in der PCR mittels Erhitzen stattgefundene Denaturierung der Amplikone und dem anschließenden Abkühlen lagern sich die entstandenen DNA- Einzelstränge wieder zu Doppelsträngen aneinander. Weichen dabei die Sequenzen an einer bestimmten Stelle voneinander ab, d.h. trägt eines der beiden Fragmente eine Mutation, bilden sich Heteroduplices mit einer Basenpaarung außerhalb des Watson-Crick-Models, wie beispielsweise A-C und G-T. In gleicher Menge entstehen Homoduplices mit komplementärer Basenpaarung. Abbildung 3: Schematische Darstellung der Bildung von Homo- und Heteroduplices. Bei heterozygoten Anlageträgern sind WT und mutierte DNA in dem Verhältnis 1:1 vorhanden. Nach Denaturierung Basenpaarungen) und und Hybridisierung Heteroduplices entstehen (AC und gleich viele Homo- GT-Basenfehlpaarungen). (AT und GC- Abkürzungen: A=Nukleotid mit der Base Adenin, T=Nukleotid mit der Base Thymin, G=Nukleotid mit der Base Guanin, C=Nukleotid mit der Base Cytosin, WT=Wildtyp, DNA=Desoxyribonukleinsäure Die Heteroduplices mit ihren Basen-„Mismatches“ können nun aufgrund der veränderten physikalischen Eigenschaften mit Hilfe einer hochauflösenden Polymermatrix von den Homoduplices getrennt werden. Das Prinzip der Fragmentauftrennung basiert auf der Ionenpaar-Umkehrphasen-Chromatographie, wobei die Eigenschaften einer stationären Phase und einer mobilen Phase ausgenutzt werden. Bei der stationären Phase handelt es sich um eine Säule aus alkylierten, hydrophoben Polystyren-Divinylbenzol Partikeln, wohingegen die 22 mobile Phase aus einem Gemisch verschiedener Puffer besteht, von denen dem amphiphilen Molekül TEAA (Triethylammoniumacetat) eine Brückenfunktion zukommt. Durch die Bindung seiner hydrophoben Alkylketten an die Chromatographiesäulen können freie Ammonium-Kationen über ihr positives Oberflächenpotenzial mit den negativ geladenen Phosphatgruppen der in der mobilen Phase gelösten DNA wechselwirken. Mit Hilfe einer kontinuierlich steigenden Acetonitrilkonzentration wird der TEAA-DNA-Komplex aus seiner hyrdophoben Interaktion mit der Säule verdrängt und somit die DNA eluiert. Mit Hilfe eines UV-Detektors konnten die eluierten Homo-und Heteroduplices bei einer Wellenlänge von 260 nm erfasst werden und anschließend softwaregestützt (Navigator ™ Software) in einem Absorptions-Zeit- Chromatogramm ausgewertet werden. In der Mehrzahl der Fälle zeigt das Chromatogramm bei Heterozygoten zwei Peaks, der erste verursacht durch die thermolabileren Heteroduplices, der zweite durch die Homoduplices (Abb. 4). Zum Teil ist es aber auch nur eine zusätzliche Schulter im Homoduplex-Peak nachweisbar die auf die Anwesenheit von Heteroduplices und damit eine Sequenzvariante schliessen lässt. (Schmitt, T. 2000). Die Art und Weise, wie sich die Heteroduplex-Peaks im Chromatogramm darstellen wird vor allem durch die Schmelzeigenschaften der dem Mismatch benachbarten Basen beeinflusst. (Ke,S.H. 1993) Bei homozygoten Anlageträgern bilden sich keine Heteroduplices, sondern es entstehen vier identische Homoduplices, so dass ein homogener Peak im Chromatogramm resultiert. (Abb. 4). Durch Zugabe von amplifizierter Wildtyp-DNA in gleichen Teilen lässt sich die Mutation jedoch darstellen. 23 Abbildung 4: DHPLC Chromatogramme Die Abbildung zeigt die Ergebnisse einer DHPLC-Analyse eines 185 bp Fragments bei 61 Grad Celsius. Mutationen im heterozygoten Zustand lassen einen Heteroduplex- Peak erkennen der deutlich vor den Homoduplices eluiert. Durch Mischen mit dem Wildtp wird bei Homozygoten ein heterozygoter Zustand erreicht. Bei der Mutation handelt es sich um einen SNP (single nucleotide polymorphism) (Abb. nach (Schmitt, T. 2000) ) 2.10.2 Puffer Für den Betrieb des Wave-Systems wurden vier Pufferlösungen benötigt. Um eine hohe Sensitivität der Analysemethode zu gewährleisten, erfolgte die Pufferherstellung äußerst sorgfältig. Die einzelnen Pufferlösungen setzten sich aus folgenden Komponenten zusammen: Puffer A: 5% TEAA Puffer B: 5% TEAA; 25% Acetonitril Puffer C: 75 % Acetonitril Puffer D: 8% Acetonitril Als Lösungsmittel diente ultrafiltriertes Wasser. Das verwendete Acetonitril wurde von der Firma JT Baker, das TEAA von Transgenomic bezogen. 24 Die Puffer A und Puffer B bildeten gemeinsam die mobile Phase, in der die zu untersuchenden DNA –Fragmente gelöst wurden. Über eine vom DHPLC-Gerät gesteuerte Pumpe mit Ventilsystem wurde für jedes PCR-Fragment ein bestimmtes Verhältnis beider Puffer erzeugt und über die Zeit eines Laufes ein linear steigender Acetonitril-Gradient gebildet. Hierbei können vier Phasen der DNA-Fragment Analyse unterschieden werden: In der ersten Phase erfolgte die Probeninjektion mit einer Erhöhung um 5 % des Anteils des Puffer B. Bei der zweiten Phase wird die DNA mittels konstanter Steigerung der Konzentration des Puffer B um 2 % pro Minute von der Chromatographiesäule eluiert. Die dritte Phase besteht aus einem Reinigungsschritt der Säulenmatrix mit 100 % Puffer B. Damit werden die noch an der Chromatographiesäule bindenden DNAFragmente gelöst. In der vierten Phase wird die stationäre Phase mit der ursprünglichen Pufferkonzentration äquilibriert. Um Kontamination der Injektionseinheit zu verhindern, wurde diese zwischen jedem Analysevorgang mit der Injektionsspritzenwaschlösung gespült. Puffer D wird bei speziellen Reinigungsvorgängen eingesetzt, um eventuelle Probenrückstände in der Chromatographiesäule zu entfernen. 2.10.3 Analysevorgang Vor jeder Analyse wurde das Gerät gemäß der Anleitung gereinigt. Danach musste das Wave-System zur Überprüfung der regelrechten Funktion zwei Mutationsstandards zweimal mit unterschiedlichen Schmelztemperaturen „High melting standard“ [70°C] und „Low melting standard“ [50°C]) innerhalb der Normwerte detektieren. Zusätzlich diente ein Kalibrierungsstandard (sizing standard) zur Überprüfung des Systems. Ferner wurde vor jedem Wechsel der Analysebedingungen (z.B. der Temperatur) eine Äquilibrierung durchgeführt. Anschließend wurden die PCR-Reaktionsgefäße mit 30 µl PCR-Produkt pro Probe in den Autosampler des Wave-Systems gestellt. Die Analyse einer Probe, von der 25 Injektion bis zum Chromatogramm dauerte ungefähr 20 Minuten und wurde gestützt auf die Navigator-Software durchgeführt. Alle 96-192 Injektionen erfolge ein Reinigungsprogramm der Säulenmatrix („extended hot wash“) mit reinem Puffer D bei 70°C.Nach 1000 Injektionen erfolgte derselbe Waschschritt, jedoch bei in umgekehrter Richtung eingebauter Chromatographiesäule („Reverse hot wash“) 2.10.4 Sensitivitätsbestimmung Zur Überprüfung der Sensitivität des Wave-Assays und zur Erleichterung der Auswertung wurden standartisierte Verdünnungsserien der Zellinien Hela, Granta, Jurkat, MHH, Daudi, Namalwa, JVM und BL-60 angefertigt und anschließend gemeinsam bei jeder Analyse separat mit den Patientenproben durch das Wave- System analysiert. Gemäss der International Agency for Research on Cancer (IARC) liegen für diese Zell-Linien Angaben über das TP53 Mutationsprofil vor: Hela, Granta, MHH, JVM, BL-60: Wildtyp Jurkat: Mutation in Exon 7: T256A Daudi: Mutation in Exon 8: G266E Namalwa: Mutation in Exon 7: R248Q 2.11 Auswertung Hierzu wurden alle Proben individuell beurteilt und auf Auswertbarkeit überprüft. Proben, die gleich hoch wie, bzw. kleiner als der PCR-Leerwert mit der Zumischung- (=LW/z.B. Granta)-Peak waren, galten als nicht auswertbar. Ebenso Proben < 2 mV. Die Wildtypzelllinie wurde als Referenz ausgewählt und mit den anderen Proben verglichen. Abweichende Muster wurden anschließend zur Validierung sequenziert. 2.12 Amplifikation der selektierten DNA-Fragmente mittels PCR Zur Re-Amplifikation der in der DHPLC Analyse positiven Probe erfolgte eine erneute PCR nach folgendem PCR-Ansatz für 50 µl: - 10 µl 5× PCR Puffer (MgSO4) 26 - 4 µl dNTP (10 mM) - 2 µl p53 Primer (reverse) Die 2 µl 2 ml 1 µl 27 µl PCR-Bedingungen p53 Primer (forward) Patientenprobe (25 ng/µl) Discoverase (Hot Star Taq Polymerase) Aqua dest. und die Gelelektrophorese zur Überprüfung Produktgewinnung waren mit dem oben genannten Protokoll identisch. der 2.12.1 Aufreinigung der PCR-Produkte Zur Aufreinigung der PCR-Produkte wurde der QIAquick PCR-Purification-Kit (Fa. Qiagen, Hilden) verwendet, das sich zur Aufreinigung von Einzel- oder Doppelstrang-PCR-Produkten der Größe 100bp bis 10kb eignet. Es enthält QIAquick Spin Colums (Säulen für die Zentrifugation mit 800µl Volumen), Collection tubes (Auffangtubes für die Zentrifugation mit 2ml Volumen) und zwei Puffer, Buffer PB und Buffer PE (Konzentrat). Zu 55ml des zuletzt genannten wurde vor Gebrauch 220ml 100% Ethanol (Fa. RdH, Seelze) gegeben. Das Set wurde bei Raumtemperatur gelagert. Außerdem wurden noch Ethanol 100% (Fa. RdH, Seelze) und Aqua ad iniectabilia (Fa. Braun, Melsungen) verwendet. Primerreste, ungebundene Nukleotide, Enzyme und Salze wurden durch die Aufreinigung mittels einer Silikagel-Membran der QIAquick Spin Colum entfernt. Danach wurde die an die Membran gebundene DNA mit Aqua ad iniectabilia eluiert. Entsprechend der Anzahl der PCR-Proben wurden die Collection tubes mit den QIAquick Spin Colums aufgestellt. Die PCR-Proben wurden jeweils mit 200µl PB-Puffer gemischt und auf die Membranen der QIAquick Spin Colums gegeben. Danach wurden die Tubes 1 Minute lang bei 13.000 Umdrehungen pro Minute zentrifugiert. Die Tubes mit dem Durchfluss wurden verworfen und die QIAquick Spin Colums in neue Tubes gestellt. Anschließend wurden 750µl PE-Puffer auf die Membranen gegeben und wieder 1 Minute bei 13.000 Umdrehungen pro Minute zentrifugiert, wobei der Durchfluss wieder verworfen und neue Tubes aufgestellt wurden. Dieser Zentrifugationsschritt unter Verwerfung des Eluats wurde noch einmal wiederholt. Danach wurden die QIAquick Spin Colums jeweils in eine Eppendorf-Tube der Größe 1,5ml mit abgeschnittenem Deckel gestellt. 27 In das Zentrum der Membranen der QIAquick Spin Colums wurden nun 50µl Aqua ad iniectabilia pipettiert und 1 Minute bei 13.000 Umdrehungen pro Minute zentrifugiert. Der gewonnene Durchfluss wurde jeweils in eine neue EppendorfTube der Größe 1,5ml gegeben und bei –20°C gelagert. 2.12.2 Cycle Sequencing Reaction (CSR) Die von (Sanger,F. 1977) entwickelte Sequenzierungsmethode bildet die Grundlage der automatisierten „Cycle Sequencing Reaction“. Bei dieser vor der eigentlichen Sequenzierung stattfindenden linearen PCR, d.h. es wird nur ein Primer dem Reaktionsgemisch hinzugefügt, wird dann dem Zufallsprinzip entweder Desoxynukleotidtriphosphate (dNTP’s) oder fluoreszenzmarkierte Didesoxynukleotidtriphosphate (ddNTP’s) von der DNA- Polymerase eingebaut. Verwendet das Enzym eines der vier ddNTP’s, kommt es zum Stop der Synthese des komplementären DNA-Stranges. Auf diese Weise entsteht von dem zu untersuchenden DNA- Templat eine Vielzahl in der Länge variierender DNA-Fragmente, die sich jeweils um ein Nukleotid unterscheiden. Anschließend werden diese DNA-Fragmente elektrophoretisch mit Hilfe eines automatischen Sequenziergerätes der Länge nach aufgetrennt, detektiert und computergestützt analysiert. Für die CSR wurden das ABI BigDye Terminator Cycle Sequencing Kit und pro Reaktionsansatz einer der Primer verwendet. Dabei enthielt das BigDye-Gemisch neben den mit vier verschiedenen Fluoreszenzfarbstoffen markierten ddNTP (Agrün, T-rot, G-schwarz und C-blau) auch modizifierte dNTP, die thermostabile AmpliTaq-DNA-Polymerase, MgCl2 und Tris-HCL-Puffer. Jeder PCR-Ansatz für die CSR von insgesamt 15 µl bestand aus 9,7 µl dHPLC Wasser 2 µl BigDye 2 µl Sequenzierpuffer 0,3 µl Primer 2 µl gereinigtes PCR-Produkt Die CSR wurde mit Hilfe eines PCR-Gerätes mit folgendem Programm durchgeführt: Nach einem initialen Denaturierungsschritt bei 95°C für 2 Minuten 28 folgten 45 Zyklen mit Denaturierung 94°C für 15 Sekunden, „Annealing“ bei 54°C für 3 Minuten und die Elongation bei 60°C für 3 Minuten. Bis zur Weiterverarbeitung wurden die PCR-Produkte bei 4°C im Kühlschrank aufbewahrt. 2.12.3 Aufreinigung der DNA mittels DyeEx Spin Kit 30 Mit dem DyeEx™ 96-Kit wurden die in der Sequenzreaktion nicht eingebauten ddNTPs entfernt. Dazu wurden zunächst die Folien auf der Ober -und Unterseite der Dye Ex 96 Platte entfernt, auf die Waste collection Platte gestellt und bei 2350 Umdrehungen pro Minute 3 Minuten lang zentrifugiert. Der Durchfluss wurde verworfen und die Dye Ex 96 Platte auf eine 12x8-well Strips Platte mit einem passenden Adapter gestellt. Anschließend wurde das gesamte CSR-Produkt jeweils in die Mitte des Gels pipettiert und bei 2350 Umdrehungen pro Minute 3 Minuten lang zentrifugiert. Der Durchfluss wurde jeweils in ein 0,5mlReaktionsgefäß gegeben und bis zur Sequenzierung bei –20°C gelagert 2.12.4 Sequenzierung Ließ das Chromatogramm der DHPLC-Analyse eine Mutation vermuten, so wurde eine Sequenzierung der Patienten-DNA durchgeführt. Die Sequenzierung mit Hilfe des automatischen Sequenziergeräts ermöglicht die Bestimmung einer Basenabfolge durch elektrophoretische Auftrennung, wobei verschieden lange fluoreszenzmarkierte DNA-Fragmente der Größe nach sortiert werden. Nach einer primären Denaturierung bei 95°C für 2 Minuten folgte das Eintauchen der mit einem polymeren Gel gefüllten Kapillare und der direkt dahinter liegenden Kathode in das Probengefäß. Durch Anlegen einer elektrischen Spannung zwischen Kathode und Anode für ca. 18 Sekunden erfolgte die sogenannte elektrokinetische Injektion, aufgrund derer sich die Ionen im elektrischen Feld am Kapillareinlass konzentrierten. Anschließend wurde das Polymer in der Kapillare aufgetrennt. Am Kapillarende befand sich ein Argon-Ionen-Laser der die nacheinander passierenden mit je einem der Fluoreszenzfarbstoffe markierten DNA-Fragmente zu Emission anregte. Das emittierte Spektrum wurde über eine CCD-Kamera (Charge-Coupled Device Kamera) detektiert. Hierbei lag das Fluoreszenzmaximum für jeden der vier Farbstoffe bei einer anderen Wellenlänge. Danach wurden die Rohdaten mit Hilfe eines Computerprogramms analysiert und in eine graphische Darstellung der DNA-Sequenz umgewandelt. Für die exakte 29 Analyse der Länge und der Basenabfolge des TP53 wurde jede Proben- Sequenz mit einer Referenzsequenz verglichen. Die Rohdaten, die während des Runs gespeichert wurden, wurden durch das Software-Programm Sequencing Analysis, Version 3.4 (Fa. PE Applied Biosystems, Weiterstadt, Deutschland) analysiert und ausgedruckt. Dann wurde die Basenabfolge der Sequenzen mit der einer WTSequenz verglichen. Punktmutationen erforderten eine besondere Sorgfalt hinsichtlich Signalverlustes von Peaks und Ersatz des Verlustes durch ein zweites darüber liegendes Signal. 2.12.5 Statistische Auswertung Es wurden paarweise Vergleiche von Patientenmerkmalen (Kovariante, unabhängige Variable) durchgeführt, um mit Hilfe des Mann-Whitney Tests stetige Variablen bzw. mit Hilfe des exakten Tests nach Fisher kategoriale Variablen zu identifizieren. Als klinische Endpunkte dieser Untersuchung wurden das Erreichen einer kompletten Remission nach der Induktionstherapie, das rezidivfreie- (RFS) und das Gesamtüberleben (OS) definiert. Dabei war für das RFS der Zeitraum bis zum Eintritt eines Rezidives oder des Todes während der kompletten Remission (CR), für das OS der Tod entscheidend. Alle Endpunkte wurden nach den empfohlenen Kriterien angewendet (Cheson et al. 2003). Überlebenskurven zur Abschätzung der medianen Überlebenszeit wurden nach der Methode von Kaplan und Meier erstellt. Die statistischen Analysen wurden mittels GraphPad Prism 4 durchgeführt. Statistische Signifikanz wurde bei einem p-Wert kleiner 0,05 erreicht. Die Dokumentation der klinischen Daten sowie die statistischen Analysen wurden in der Studienzentrale der Klinik für Innere Medizin III durchgeführt. 2.12.6 Reagenzien, Chemikalien und Geräte (siehe Anhang) 30 3. Ergebnisse 3.1 Inzidenz von TP53-Mutationen Die zentrale Region des TP53 enthält die DNA Bindungsdomäne und ist das Ziel von 90% der TP53 Mutationen, die in Tumoren gefunden wurden (sog. „Hot-Spotregion“). Daher untersuchten wir bei 234 Patienten mit komplexem Karyotyp das TP53 in den Regionen Exon 4 bis Exon 10, die die Hot-Spot Region repräsentieren. Hierzu wurde die denaturierende Hochdruck- flüssigkeitschromatographie (=DHPLC) angewendet. Im positiven Fall erfolgte anschliessend eine konventionelle Sequenzierung nach Sanger. Insgesamt konnten in 120 Patienten 144 Mutationen (exon 4 [e4] n=7; exon 5 [e5] n= 43; exon 6 [e6] n=23; exon 7 [e7] n= 34; exon 8 [e8] n= 35; exon 10 [e10] n= 2) nachgewiesen werden (Inzidenz: 120/234=51,2%), mit einem Maximum von 3 Mutationen pro Fall. 114 Patienten hatten keine Mutation. 3.2 TP53 Mutationstypen Biochemische Analysen zeigten, dass Mutationen von TP53 sehr unterschiedliche Einflüsse auf die Restaktivität des Proteins, DNA- Bindungsaktivitäten oder Transkription haben können. (Soussi,T. 2000) Die DNA- Bindungsdomäne von P53 ist stark degeneriert und die Wildtyp- oder mutierte Version des P53 haben verschiedene Affinitäten. Das mutierte Protein zeigt durch Inaktivierung auch einen dominanten Effekt auf die Funktion des Wildtypes, wodurch die Bedeutung einer einzelnen Allel-Mutation hervorgerufen wird. (Resnick,M.A. 2003) Obwohl die Kanzerogenese bei den meisten Tumorsuppressorgenen einen Verlust beider Allele erfordert, kann die Mutation eines einzelnen Allels des TP53 schon zu einem kompletten Funktionsverlust des Proteins führen. Die neueste Version der UMD P53 Database beinhaltet 31,000 Mutationen von 29,200 Patienten (einige Patienten haben mehrere Mutationen). TP53 Mutationen, die gehäuft in verschiedenen Patienten vorkommen werden als sogenannte „Hot-Spot“- Mutationen bezeichnet. (Soussi,T. 2000) Die Datenbank gibt Auskunft über die Nomenklatur, die Häufigkeit der einzelnen Mutationstypen, Informationen über den Grad der Restaktivität des Proteins und versucht die verschiedenen Mutationstypen zu klassifizieren und klinische Analysen zu erleichtern. 31 Von den 144 Mutationen der untersuchten Kohorte waren 112 Missense Mutationen (78%), 17 frame-shift (12%), 7 nonsense (5%) und 8 Mutationen befanden sich in intronischen Splice-Regionen (5%). Zusätzlich konnten 166 Polymorphismen (71%), 113 davon in intronischen, 53 in exonischen DNA- Abschnitten detektiert werden. Jeweils drei Mutationen lagen in zwei Patienten (e5, e6, e8) und (e4, e5, e7) sowie jeweils zwei Mutationen in zwanzig Patienten vor. ([e5, e6 ] n=5; [e5,e8] n=3 ; [e5, e7] n=3; [e7, e7] n= 2 ;[e7, e8] n=2 ; [e6, e8] n=1; [e5, e5] n=1 ; [e4, e6]n=1; [e6, e7] n= 1, [e6, e7] n=1). 5% 12% 5% 78% Missense Nonsense Frameshift splice Abbildung 5: Prozentuale Verteilung der Mutationstypen im TP53 3.2.1 Missense Mutationen Bei den Missense Mutationen kommt es durch den Austausch einer einzelnen Base zu einer veränderten Aminosäuresequenz im Protein und damit zu einer veränderten Funktion. 112 der insgesamt 144 Mutationen (78 %) waren dadurch gekennzeichnet und bilden somit die häufigste Mutationsart. Eine Analyse dieser Punktmutationen zeigte, dass es sich in 34 Fällen (30%) um G> A Transitionen, in 20 Fällen (17%) um A>G Transitionen und in 9 Fällen (8%) um T>A (8%) Transversionen, bzw. 6 Fällen (5%) um C>G Transversionen handelt. Eine 32 Transition ist durch einen Basenaustausch innerhalb der gleichen NukleotidbasenGruppe gekennzeichnet. Hierbei kommt es zu einem Austausch zwischen zwei Purinbasen (A<>G) und den beiden Pyrimidinbasen (C<>T). Eine Transversion hingegen ist ein Austausch zwischen den beiden Nukleotidbasen-Gruppen, d.h. eine Purinbase (A oder G) wird durch eine Pyrimidinbase (C oder T) und umgekehrt ausgetauscht Tabelle 2: Tabellarische Aufzeichnung aller Missense-Mutationen im TP53 Bp: Basenpaar; WT-Codon: Wildtyp-Codon; mut.Codon: mutiertes Codon; AA: Aminoacid ; n.a. = nicht verfügbar. Effect-group: Mutations-Klassifikation basierend auf der 3D-Protein Struktur und dem Mutationstyp; 1: missense in DNA-binding loops, 2: other missense; 0: keine Mutation. Abkürzungen der Basen und Aminosäuren nach der internationalen Nomenklatur: Ala: Alanin; Arg: Arginin; Asn: Asparagin; Asp: Asparaginsäure; Cys: Cystein; Gln: Glutamin; Glu: Glutaminsäure; Gly: Glycin; His: Histidin; Leu: Leucin; Lys: Lysin; Phe: Phenylalanin; Pro: Prolin; Ser: Serin; Tyr: Tyrosin; A: Adenin; G: Guanin; C: Cytosin; T: Thymin 33 34 35 Y126N L130V K132R FS:del37(130) M133K C135S FS:del1 (147) FS:del13(152) inFS:del6(157) T155N FS:del1(51) R158C FS:del1(51) Y163C K164E K164N FS:del4(96) Q165X H168P H168R FS:del1(172) FS:del1(110) R175H TP53 - Verlust C176S I195T G245C G245C R248W R248Q R248W R248Q R196P R196P M246V R196X R196X R196X R248W R248Q V203E FS:del2(209) H179R H179R Exon 5 N239D S240I S241F S241Y G245C G245S G187S G187S FS:del1(188) R175H C176S Missense M237I M237I R175H R175H R175H kein VerlustTP53 FS:del1(107) N235S FS:del8(172) F113V Exon 4 R158H Frameshift Nonsense Y234C Y234C K132R T211N R248W R248Q R248W R213L R213X S215R R249S L265P G266R F270L V272L V272M V272M R273H R273C R273H R273L R273C R273C R273H V274A C275Y C275Y C275Y V218A C275Y Y220C Exon 6 R283P Exon 7 Exon 8,9 FS:del1(265) Q136X C141Y R213Q V143M V143M FS:del5(145) Y220C Y220C Y220C Y234C C238Y N239D G245S inFS:Ins3(161) inFS:del9(253) R158H L252I C176S G245S L257V P177R L257P H179D P278R L260F R337C R342X Exon 10 R267P F270L V272L C275Y P278A R280S R280G D281N R282W R283C E286A E286G Abbildung 6: Abbildung von 136 TP53 Mutationen von Exon 4 – Exon 10: Hemizygote Mutationen befinden sich im unteren Bereich des Schaubildes, heterozygote, und/oder homozygote Mutationen im oberen Bereich. Exon 4-Exon 10 ist hier abgebildet: die Missense Mutationen (schwarz), Nonsense Mutationen (rot), und Insertionen/Deletionen (in/del) (grün) sind in ihrer ungefähren Lage abgebildet. Insertionen und Deletionen bedeuten häufig eine Verschiebung des Leserasters (FS= Frameshift), z.B. FS: del1(51) ist eine Frameshift Mutation durch die Deletion einer Base im Codon 51. Abbildung 7: Beispiel einer Missense Mutation: Heterozygote Missense Mutation im Codon 272 in Exon 8 36 Abbildung 8: Chromatogramm; Mutation P: Val 272 Leu (GTG>TTG), Heterozygot bei 59,0°C; Abkürzung der Aminosäuren und Basen Val: Valin; Leu: Leucin; G: Guanin; T: Thymin Abbildung 9: Chromatogramm; Mutation P: Val 272 Leu (GTG>TTG),Heterozygot bei 62,0 °C, Abkürzung der Aminosäuren und Basen Val: Valin; Leu: Leucin; G: Guanin; T: Thymin 37 3.2.1.1 Mutationsmuster der Missense-Mutationen im TP53 T:A>G:C T:A>C:G T:A>A:T C:G>T:A C:G>G:C C:G>A:T 0 10 20 30 Häufigkeitsverteilung 40 50 Abbildung 10: Mutationsmuster der Missense-Mutationen im TP53 Die Abbildung zeigt eine Häufigkeitsverteilung der jeweiligen mutierten Basen aller MissenseMutationen und bestätigt das vermehrte Auftreten von C:G>T:A Transitionen (43%), wie sich das auch in Lunge-, Ovarial-, Kolorektal-, Melanomen, und Brusttumoren zeigte. (Pfeifer,G.P. 2009) Abkürzungen der Basen : A: Adenin, C: Cytosin; G: Guanin; T: Thymin 3.2.2 Nonsense Mutationen Da ein einzelner Basenaustausch auch zu einem Stop-Codon (TGA bzw. TAA, TAG) führen kann, welches zum völligen Abbruch der Aminosäuresynthese an dieser Stelle führt nennt man diese Mutationen Nonsense-Mutationen. Die Aktivität des P53 wird als quasi null angenommen. Im vorliegenden Patientenkolletiv konnten wir diese Art der Mutation bei 7 Patienten finden. Hierbei war das Codon 196 im Exon 6 bei drei Patienten durch den Austausch (C>T) betroffen. Einzelne Mutationen gab es im Codon 136, 165, 213 auf Exon 5 und 6 und eine Mutation im Exon 10 im Codon 342. Tabelle 3: tabellarische Aufzählung aller Nonsense-Mutationen im TP53 Bp: Basenpaar; WT-Codon: Wildtyp-Codon; mut.Codon: mutiertes Codon; AA: Aminoacid; WT: Wildtyp; Homo: Homozygot, Hetero: Heterozygot; Effect-group: Mutations-Klassifikation basierend auf der 3D-Protein Struktur und dem Mutationstyp; 4: Frame Shift + Splice + nonsense; Abkürzungen der Basen und Aminosäuren: A: Adenin, C: Cytosin, G: Guanin, T: Thymin, Arg: Arginin; Gln: Glutamin 38 Exon Codon Exon 5 136 Exon 6 196 Exon 5 Exon 6 Exon 6 Exon 6 Exon 10 3.2.3 165 196 196 213 342 Be- WT mut WT_ mut_ Protein Homo TAA Gln Stop Q 136 X Arg Stop R 196 X schreibung Codon Codon C>T CAG TAG C>T C>T C>T C>T C>T C>T CAA CGA CGA CGA CGA CGA TGA TGA TGA TGA TGA AA Gln Arg Arg Arg Arg AA Stop Stop Stop Stop Stop Q 165 X R 196 X R 196 X R 213 X R 342 X Biallelic zygous aberration 0 0 0 0 0 0 1 0 1 1 1 1 1 1 Effect group 4 4 4 4 4 4 4 Frame-Shift Zudem fanden sich 17 Frame-Shift Mutationen, die jeweils durch eine Deletion oder Insertion zu Verschiebung des Leserasters führten, dabei lag in 14 Fällen eine Frame-shift Mutation vor. (siehe Abbildung). Drei Mutationen verursachten durch den Verlust eines Tripletts zwar den Ausfall einer Aminosäure jedoch keiner Verschiebung des Leserasters, weswegen es als In-Frame Mutation bezeichnet wird. [exon 4: n= 5; exon 5: n=8; exon 6: n=2 ; exon 7: n=1 ; exon 8: n=1 ] Tabelle 4: Tabellarische Aufzählung aller Frame-Shift Mutationen im TP53 Bp: Basenpaar; WT-Codon: Wildtyp-Codon; mut.Codon: mutiertes Codon; AA: Aminoacid; WT: Wildtyp; Homo: Homozygot; Effect-group: Mutations-Klassifikation basierend auf der 3D-Protein Struktur und dem Mutationstyp; 3: inFrame, Deletion, Insertion; 4: Frame Shift + Splice + nonsense; Abkürzungen der Basen und Aminosäuren A: Adenin, C: Cytosin, G: Guanin, T: Thymin, Arg: Arginin; Gln: Glutamin; Asn: Asparagin; Arg: Arginin, Glu: Glutaminsäure,Leu: Leucin; Ser: Serin; Thr: Threonin; Tyr: Tyrosin Exon Exon 4 Codon 51 Be- 110 del 1 96 130-142 del 37 172 del 8 Exon 5 145 Exon 5 Exon 5 del 4 Ins 3 157-159 Exon 5 del 1 161 Exon 5 172 AAC Glu Asn Tyr Thr TCC Exon 4 Exon 5 mut_ TCT del 1 del 4 Exon 4 WT_ Codon 96,97 107 mut Codon Exon 4 Exon 4 WT schreibung GAA TAC ACG TCT TCC CGT TGT inFS 0 0 3 NA FS 1 1 4 Ala inFS 0 1 3 NA FS 0 0 4 NA FS 0 1 4 Leu GTT GCG Val Ala NA NA NA NA FS Arg Lys FS 0 1 4 The Thr inFS 0 0 3 GTT NA AAA del 9 ACC ACA del 1 Val Ser Leu AGA 265 Ser CTG del 2 Exon 8 4 NA NA del 1 4 NA del 5 188 0 4 NA 209 253-256 0 1 4 Exon 6 Exon 7 1 0 1 del 13 Exon 6 FS 1 Effect group 1 GCC del 1 FS FS Biallelic FS 152-156 147 Ser Homo zygous aberration Leu Exon 5 Exon 5 pretation Arg GTC del 1 Ser Inter- AA CTC del 6 CTC AA GTT CTG CTG TTG NA Na NA Val Val Leu NA 39 Trp NA NA FS FS FS FS FS 0 1 1 0 0 0 1 1 1 1 1 0 4 4 4 4 4 4 Abbildung 11: Beispiel 1: Frame-Shift Mutation, Deletion 1, Codon 107 Abbildung 12: Beispiel 2: Frame Shift Mutation, Deletion 8bp , Codon 172 3.2.4 Intronische Splice-Mutationen 8 Mutationen fanden sich in intronischen Splice-Regionen, die hauptsächlich die Exone 5 und 6 betrafen. Die Validierung entstammt der International Agency for Research on Cancer (IARC), die Sequenz der Version X54156 der Gen-Bank. Dabei wies ein Patient eine Mutation sowohl im Intron 4 als auch Intron 8 auf. Tabelle 5: tabellarische Aufzählung aller Mutationen in Intronic-Splice Regionen Bp: Basenpaar; WT:Wildtyp; Mut: Mutiert; AA: Aminoacid, NA: Not available; Abkürzungen der Basen nach der internationel Nomenklatur: A: Adenin, C: Cytosin, G: Guanin, T: Thymin Intron Bp Be- WT Mut WT_ mut_ Inter- Intron 4 bp: 13.054 G>A NA NA NA NA intron 4 splice Intron 5 bp. 12.300 G>T NA NA NA NA intron 4 splice schreibung Codon Codon AA Intron 4 bp: 13.054 G>A NA NA NA AA NA pretation intron 4 splice Intron 5 bp: 13.318 A>G NA NA NA NA intron 5 splice Intron 5 bp: 13.239 G>A NA NA NA NA intron 5 splice Intron 5 bp: 13.239 G>A Intron 8 bp: 14.589 G>C Intron 8 bp: 14.589 G>C NA NA NA NA NA NA NA NA NA 40 NA NA NA intron 5 splice intron 8 splice intron 8 splice 3.3 TP53 Sequenzvarianten Varianten in der TP53 DNA -Sequenz, die auch in gesunden menschlichen Populationen vorkommen, werden als Polymorphismen oder EinzelnukleotidPolymorphismus (engl. SNP =Single Nucleotide Polymorphism) berücksichtigt und in einem Datensatz, der IARC TP53 Mutation Database, zusammengefasst. (Olivier,M. 2002) Häuft kommt es zu sogenannten Missense Mutationen mit Austausch einer Aminosäure [z.B. P72R, P278A, P278R], Insertionen oder SilentMutationen, bei denen es durch einen einzelnen Basenaustausch zwar zu einem veränderten Triplett nicht aber zu einem Austausch einer Aminosäure kommt [z.B. P34P, P36P, S149S, R213R]. Die meisten der TP53 Sequenzvarianten befinden sich in Introns, außerhalb der Splice-Regionen. Insgesamt konnten mehr als 200 SNPs im TP53 identifiziert werden. Die funktionellenen Konsequenzen sind aber noch weitestgehend unbekannt und nur Wenige wurden bislang ausführlich untersucht. Theoretisch könnten sie durch die verstärkte Mutabilität der geänderten DNA-Sequenz einen Effekt auf die P53 Funktion haben, gesteigerte Splicing-Tendenzen, instabile Transkripte, Translationen oder gewebespezifische Veränderungen nach sich ziehen. (Lozano,G. 1991) In unserem Patientenkollektiv fanden sich insgesamt 53 Sequenzvariationen im Bereich der Exons 4-8. Darunter 40 missense-Mutationen und 13 Silentmutationen und 113 Sequenzvariationen im Bereich der Introns 3-10: Exon 4 (n=42); Exon 5 (n=1); Exon 6 (n=8); Exon 8 (n=2); gesamt: n=53 (22%) Tabelle 6: Tabellarische Aufzählung aller Sequenzvariationen in exonischen Bereichen des TP53 Bp: Basenpaar; WT-Codon: Wildtyp-Codon; mut.Codon: mutiertes Codon; AA: Aminoacid; WT: Wildtyp; Homo: Homozygot; Hetero: Heterozygot; Effect-group: Mutations-Klassifikation basierend auf der 3D-Protein Struktur und dem Mutationstyp; 0: silent+intron; 1: missense in DNA-binding loops; 2: other missense; Abkürzungen der Basen und Aminosäuren: A: Adenin, C: Cytosin, G: Guanin, T: Thymin; Ala: Alanin, Arg: Arginin; Pro: Prolin; Ser: Serin 41 Exon Codon BP 4-exon 72 bp:12.139 4-exon 36 bp:12.032 4-exon bp:12.026 34 5-exon 149 8-exon 278 6-exon 8-exon 213 278 bp:12.435 bp:13.399 bp:14.501 bp:14.502 Be- WT Mut WT_ mut_ Protein P G>C CGC CCC schreibung Codon Codon AA Arg AA Pro P72R C>G CCC CCG Pro Pro P34P C>A TCC TCA Ser Ser S149S G>A CCG CCA Pro Pro A>G CGA CGG Arg Arg C>G CCT CGT Pro Arg C>G CCT GCT Pro Ala P36P R213R P278A P278R Inter- Effect Häufigkeit pretation group missense 38 0 3 silent 0 silent 0 silent silent missense missense n 2 0 1 1 1 1 8 1 1 3-intron (n=5); 4-intron (n=20); 6-intron (n=28); 7-intron (n=55); 9-intron (n=3); 10intron (n=2), gesamt: n=113 (48%) Tabelle 7: Tabellarische Aufzählung aller Sequenzvariationen in intronischen Regionen des TP53 Bp: Basenpaar; WT-Codon: Wildtyp-Codon; mut.Codon: mutiertes Codon; AA: Aminoacid; WT: Wildtyp; NA: Not available; Ins: Insertion; Effect-group: Mutations-Klassifikation basierend auf der 3D-Protein Struktur und dem Mutationstyp; 0: silent+intron; Abkürzungen der Basen: A: Adenin, C: Cytosin, G: Guanin, T: Thymin Intron BP BeWT schreibungCodon mut Codon WT_ AA mut_ AA NA NA NA NA NA NA NA 3-intron bp: 11.992 C> A NA 4-intron bp: 12.300 NA NA NA NA NA NA NA NA NA NA 3-intron bp: 11.966 4-intron bp: 12.317 Ins C>A NA A>G NA 4-intron bp: 12.964 A>G 6-intron bp: 13.494 G>A 6-intron bp: 13.463 NA NA NA NA NA NA NA NA NA 6-intron bp: 13.964 G>C NA NA NA NA 7-intron bp: 14.201 T>G NA NA NA NA 10-intron bp: 17.708 A>T 7-intron bp: 14.181 9-intron bp: 14.766 3.4 C>T T>C NA NA NA NA NA NA NA NA NA NA NA NA InterEffect Häufigkeit pretation group n intronic 0 4 intronic 0 1 intronic 0 1 intronic 0 1 intronic 0 18 intronic 0 1 intronic 0 20 intronic 0 7 intronic 0 28 intronic 0 27 intronic 0 3 intronic 0 2 Exonverteilung der Mutationen Basierend auf Studien, in denen die gesamte TP53-Gensequenz untersucht wurde, gruppieren sich 86 % der Mutationen zwischen Codon 125 (=Exon 5) und 300 (=Exon 8), was der DNA – Bindungsregion entspricht. Die meisten davon sind Missense Mutationen. Außerhalb dieser Regionen dominieren Nonsense- und Frame-Shift Mutationen. (Olivier,M. 2010) Auch in der vorliegenden Gruppe befanden sich 110 von 112 (98%) der Missense-Mutationen in diesem Bereich, 6 von 7 (86 %) der Nonsense Mutationen und 12 von 17 (71 %) der Frame Shift Mutationen. 42 Die nachfolgende Abbildung gibt eine Übersicht der Verteilung der Missense Mutationen auf die Codon 113 bis 337. 28 von 122 (= 24%) fanden sich in den Codons 175 (n=5), 245 (n=6), 248 (n=9), 273 (n=7) und 282 (n=1), die als sogenannten Hot-Spot-Regionen gelten, da sie im Vergleich mit allen somatischen TP53-Mutationen gehäuft sind. 10 9 8 Häufigkeit 7 6 5 4 3 2 1 113 130 133 141 155 163 168 176 179 195 203 213 218 234 237 239 241 246 249 257 265 267 272 274 278 281 283 337 0 Codon Nummer Abbildung 13: Häufigkeitsverteilung der Mutationen in Bezug auf die Codons Diese Abbildung stellt die Häufigkeitverteilung der Mutationen auf die jeweiligen Exone dar. Hier Exon 4 (e4), Exon 5 (e5), Exon 6 (e6), Exon 7 (e7), Exon 8 (e8) und Exon 10 (e10). (n= Anzahl der Mutationen) [e4] n=7; [e5] n=43; [e6] n=23; [e7] n=34; [e8] n=35; [e10] n=2 43 50 Anzahl der Mutationen 45 40 35 Exon 4 25 Exon 6 Exon 5 30 Exon 7 20 Exon 8 15 Exon 10 10 5 0 Exon 4 Exon 5 Exon 6 Exon 7 Exon 8 Exon 10 Abbildung 14: Häufigkeitsverteilung der Mutationen auf die Exone 3.5 Exon- und Intronverteilung der Polymorphismen Anzahl der Polymorphismen 60 50 40 30 20 10 0 Intron Exon 4 Intron Exon 5 Exon 6 Intron Exon 8 Intron Intron 3 4 7 9 10 Abbildung 15: Häufigkeitsverteilung der Polymorphismen Das Vorkommen der Polymorphismen konzentrierte sich im exonischen Bereich auf die Exone 4 (n=42), Exon 5 (n=1), Exon 6 (n=8) und Exon 8 (n=2) und im 44 intronischen Bereich auf Intron 3 (n=5), Intron 4 (n=20), Intron 6 (n=28), Intron 7 (n=55), Intron 9 (n=3) und Intron 10 (n=2). 3.6 Korrelation des TP53-Status mit klinischen Daten Tabelle 8: Klinische Charakterstik basierend auf dem TP53 Mutationsstatus TP53 Wildtyp TP53 mutiert 33 (51%) / 32 (49%) 45 (50%) / 45 (50%) Leukozyten (10 /L, Median) 11.7 6.4 Hämoglobin (g/L, Median) 9.1 8.8 Blasten im peripheren Blut (%, Median) 45 411 27 430 .09 31 (48%) 22 (34%) 24 (27%) 49 (54%) .01 Gesamtüberleben (Monate, Median) 10.0 4.0 <.0001 Rezidivfreies Überleben (Monate, Median) 10.3 5.7 .003 Klinische Daten Geschlecht (Mann/Frau) Alter (Jahre, Median) 6 Blutplättchen (10 /L, Median) 6 Blasten im Knochenmark (%, Median) LDH Wert (U/L, Median) Therapie-Ansprechen Komplette Remission nach Induktion Refrakträre Erkrankung nach induktion Überleben Ereignisfreies Überleben (Monate, Median) n=65 56 47 73 1.3 n=90 p-Wert 63 .005 41 .27 65 1.1 .99 .09 .21 .09 .43 .01 .002 Zur Beantwortung der Fragestellung inwieweit der TP53 Mutationsstatus mit bestimmten klinischen Merkmalen assoziiert ist führten wir eine Korrelation der wichtigsten klinischen Charakteristika basierend auf dem TP53-Mutationsstatus durch. Da nicht von allen 234 Patienten klinische Daten vorlagen wurden die Analysen von den 155 Patienten durchgeführt, von denen ausreichend klinische Daten vorlagen. Hier zeigte sich, dass Patienten mit mutierten TP53 Status älter waren (Median 63 Jahre vs. 56 Jahre), im Trend weniger Leukozyten (Median 6,4 % vs. 11,7 %) sowie weniger Blasten im Knochenmark (Median 65 % vs. Median 73 %) und im peripheren Blut (Median 27% vs. 45 %) hatten. Das Ansprechen auf die Therapie ergab signifikante Unterschiede zwischen der mutierten und der Wildtyp Form des TP53. Eine komplette Remission (CR) nach Induktion konnte bei 48 % der Patienten mit Wildtyp-Form des TP53 erreicht werden, wohingegen nur 27 % der TP53-mutierten AML Patienten eine CR erreichten. Nach Induktion waren 34 % der Patienten mit TP53-Wildtyp refraktär; die Rate an refraktärer Erkrankung war bei Patienten mit TP53 Mutation mit 54 % signifikant höher. 45 3.7 Korrelation des TP53-Status mit genomischen Veränderungen Abbildung 16: Korrelation des TP53 Status mit genomischen Veränderungen Die Korrelation des TP53 Status ergibt, dass die mutierte Form (TP53mut= rot) sowohl eine erhöhte Anzahl an Verlusten, Zugewinnen, als auch Amplifikationen und Aberrationen aufweist. (TP53WT: Wildtyp des TP53= schwarz) 46 Abbildung 17: Korrelation des TP53 Status mit Verlust bzw. Zugewinn von genomischen Material Patienten mit einer AML mit TP53 Mutationen wiesen im Vergleich zur AML mit Wildtyp TP53 eine signifikant höhere Rate an umschriebenen genetischen Veränderungen auf: Verlust von 5q (80 % vs. 37 %), Verlust von 7q (60% vs. 45%), Verlust von 17p (56% vs. 29%), Verlust von 16q (37% vs. 15%), Verlust von 18q (35% vs. 17%), Verlust von 12p (32% vs. 22 %), Zugewinn von 8q (28% vs. 29%), Zugewinn von 11q (33% vs. 16%), Zugewinn von 21q (19% vs. 12%), Zugewinn von 1p (21% vs. 9%), Amplifikation 11q (16% vs. 0%), Amplifikation 21q (13% vs. 4 %), Amplifikation 8q (2% vs. 5 %). Diese Ergebnisse legen nahe, dass TP53 Mutationen mit einer erhöhten genetischen Instabilität assoziiert sind. 47 3.8 Überlebensanalysen Abbildung 18: Überlebensanalysen der Patienten mit Mutation im TP53 im Vergleich zu Patienten ohne Mutation innerhalb eines komplexen Karyotyps bei Patienten mit Akuter myeloischer Leukämie. OS= Overall Survival, EFS= Event free Survival, RFS= Relapse free Survival 48 Für die Evaluation der prognostischen Bedeutung einer TP53-Mutation innerhalb eines komplexen Karyotyps wurden Korrelationen mit dem klinischen Verlauf für die Endpunkte Gesamtüberleben, ereignisfreies Überleben und rezidivfreies Überleben durchgeführt. Für die alle drei Endpunkte zeigte sich ein signifikanter Unterschied zwischen Patienten mit TP53- Wildtyp und Patienten mit TP53Mutation. Für alle drei Endpunkte war das Vorhandensein einer TP53-Mutation mit einem signifikant schlechteren Verlauf assoziiert. Das mittlere Gesamtüberleben betrug 6 Monate, das mittlere ereignisfreie Überleben lag bei ca. 3,5 Monaten und das mittlere rezidivfreie Überleben bei 6 Monaten für Patienten mit TP53-Mutation. 49 4. Diskussion 4.1 Das Tumorsuppressorgen Das TP53-Gen ist ein Tumorsuppressorgen und liegt auf dem kurzen Arm von Chromosom 17. Als „Wächter des Genoms“ ist es an vielen verschiedenen zellulären Prozessen beteiligt. (Isobe, M. 1986), (Levine, A.J. 1997) So reguliert es als Transkriptionsfaktor unter anderem nach DNA-Schädigung und Hypoxie die Expression von Genen, die an der Kontrolle des Zellzyklus, an der Induktion der Apoptose oder an der DNA-Reparatur beteiligt sind. (Levine, A.J. 1997) Der Verlust der p53-Funktion führt zur genetischen Instabilität, einer gestörten Apoptose und unkontrollierten Zellproliferation. (Livingstone, L.R. 1992), (Yin, Y. 1992) In der AML ist TP53 hauptsächlich durch Deletionen und Punktmutationen inaktiviert, die sich fast ausschließlich in den Exonen 4-8 befinden. (Fenaux, P. 1991) Es gibt eine deutliche Assoziation von 17p-Deletionen und TP53 Mutationen was zum Verlust beider TP53 Allele führt. Ebenso zeigt sich eine enge Korrelation der TP53 Mutationen mit dem Alter, chromosomalen Aberrationen an Chromosom 5 und/oder 7 (Deletionen oder Verlust) und dem komplexem Karyotyp. (Fenaux, P. 1991), (Christiansen, D.H. 2001) Patienten mit TP53 Mutationen sind häufiger resistent gegenüber Chemotherapien und haben eine sehr kurze Überlebenszeit. (Wattel, E. 1994) TP53 liegt in vielen Tumorarten in hoher Frequenz meist durch Punktmutationen mutiert vor. Durch die Anhäufung der Mutationen in den sog. Hot-Spot Regionen ist TP53 relativ leicht zu analysieren. (Soussi, T. 2000) TP53Mutationen sind bei der akuten myeloischen Leukämie mit 2-3 % relativ selten, jedoch liegt der Anteil von TP53-Mutationen bei der akuten myeloischen Leukämie innerhalb der Gruppe mit komplexem Karyotyp weitaus höher und ist mit einer schlechten Gesamtüberlebensrate assoziiert. In dieser Arbeit ermittelten wir die Inzidenzen von TP53-Mutationen an einer großen, genetisch gut charakterisierten AML-Kohorte mit komplexem Karyotyp. Ziel war es, die prognostische Bedeutung von TP53-Mutationen innerhalb der AML mit komplexem Karyotyp zu evaluieren, insbesondere zu zeigen in wie weit die schlechte Prognose dieser zytogenetischen Subgruppe auf das Vorhandensein einer TP53 Mutation zurückzuführen ist. Darüber hinaus führten wir Korrelationen von TP53 Mutationen mit klinischen und weiteren genetischen 50 Aberrationen durch um zu prüfen inwieweit TP53-Mutationen mit bestimmten chromosomalen Veränderungen und klinischen Charakteristika assoziiert sind. Insgesamt konnten 144 TP53 Mutationen in 120 von 234 AML Patienten mit komplexem Karyotyp nachgewiesen werden (51,2 %). 22 Patienten zeigten mehr als eine Mutation. Von diesen 144 Mutationen der untersuchten Kohorte waren 112 Missense Mutationen (78 %), 17 frame-shift (12%), 7 nonsense (5%) und 8 Mutationen befanden sich in intronischen Splice-Regionen (5%). Sieben Mutationen fanden sich in Exon 4, 43 in Exon 5, 23 in Exon 6, 34 in Exon 7, 35 in Exon 8 und zwei Mutationen in Exon 10. (vgl. 3.4) Zusätzlich konnten 166 Polymorphismen, 113 davon in intronischen, 53 in exonischen DNA- Abschnitten detektiert werden. 4.2 Das Mutationsprofil des TP53 Die IARC (International Agency for Research on Cancer) TP53 Datenbank gibt genaue Angaben über die Prävalenz von Mutationen im TP53-Gen in menschlichen Tumoren. Basierend auf Studien, die die gesamte TP53- Gensequenz zahlreicher Tumoren untersuchen, gruppieren sich 86 % der Mutationen (n=24210) zwischen Codon 125 (Exon 5) und 300 (Exon 8). Ein Abschnitt, der die DNA–Bindungsregion repräsentiert. Bei den meisten Mutationen handelt es sich um Missense Mutationen (87,9%). Außerhalb dieser Regionen dominieren Nonsense- und Frame-Shift Mutationen. Innerhalb der Punktmutationen haben mit ca. 25 % den grössten Anteil C:G>T:A Substitutionen an CpG Dinukleotiden (Cytosin-Phosphat-Guanin). Obwohl in vielen Tumorarten diese Transitionen dominieren, erscheint dennoch ein sehr komplexes Bild, wenn man sich auf seltenere Tumorarten konzentriert. Die Mutationsmuster zwischen zwei verschiedenen Krebsarten oder auch innerhalb von zwei Patientenpopulationen innerhalb einer Krebsart können sehr stark variieren. (Olivier,M. 2010) In der hier untersuchten Kohorte, die sich nur auf die Patienten mit AML und komplexen Karyotyp beschränkte, lagen 110 von 112 (98%) der MissenseMutationen, 6 von 7 (86 %) der Nonsense Mutationen und 12 von 17 (71 %) der Frame Shift Mutationen innerhalb Codon 125 -300. Mutations-Gruppe dominierten deutlich 51 Innerhalb der Missense- Transitionen, mit 30% die G>A Transitionen, gefolgt von A>G Transitionen (17%). Transversionen machten einen kleineren Anteil aus. (T>A (8%) und C>G (5%)). Einige epidemiologische Studien haben versucht ein tumorspezifisches Mutationsmuster zu identifizieren, um potentielle Mutagene und Karzinogene zu erforschen. In verschiedenen Krebsarten sollen diese zu einer Art epidemiologischen Fingerabdruck führen. Dazu wurden detaillierte Aufzeichnungen in verschiedenen Organismen und Experimenten in vivo und in vitro gestartet um eine Korrelation der Mutationsentstehung und der Exposition gegenüber Mutagenen zu erläutern. Hierzu zählen zum Beispiel das gehäufte Vorkommen von CC>TT Transitionen im Zusammenhang mit UV-Strahlung und Hautkrebs (Giglia-Mari, G. 2003) oder auch eine vermehrte G>T Transversion in Lungenkrebs bei Rauchern verglichen mit Nicht-Rauchern. (Ding,L. 2008) Für die hier am häufigsten gefundene Transition mit G>A oder A>G sind bislang keine Zusammenhänge bekannt. Auch über einzelne Transversionen wie T>A und C>G liegen keine Daten vor. Demnach werden weitere Studien zu TP53 Mutationen dazu betragen das Zusammenwirkungen verschiedener Faktoren auf TP53 Mutationsmuster zu verstehen, insbesondere auch im Vergleich unterschiedlicher Patientenkohorten. Auch die Tatsache, dass TP53 Mutationen in ihrer biologischen Wirkung sehr unterschiedlich sind und dass sie in verschiedenen Stadien der Tumorentwicklung vorkommen können, ist es unmöglich einen einfachen Zusammenhang abzuleiten, der sich einheitlich auf den klinischen Kontext übertragen lässt. (Olivier,M. 2010) 4.3 Mögliche Bedeutung von TP53 Polymorphismen Es wird angenommen, dass das menschliche Genom aus ungefähr drei Billionen Basenpaaren besteht, wovon die sogenannten single-nucleotide-polymorphisms (SNP) einen Anteil von mindestens einer Million Basenpaaren ausmachen. Diese Polymorphismen sind als eine genetische Variante definiert, die in der Allgemeinpopulation mit einer Frequenz >1 % vorhanden ist. (Sachidanandam, R. 2001) Mehr als 200 Sequenzvarianten im TP53 wurden identifiziert. Allerdings sind die meisten dieser Sequenzvariationen wahrscheinlich ohne biologische Wirksamkeit. Ausstehend ist, inwieweit diese Polymorphismen funktionell über eine veränderte p53 Funktion auf ein erhöhtes Tumorrisiko einwirken könnten. Da 52 die Effekte der Polymorphismen sehr subtil und entsprechend dem genetischen Hintergrund stark variieren können, ist es herausfordernd einen Effekt auf die Tumorentstehung nachzuweisen und es werden grosse Patientenkohorten benötigt um ein grösseres und verständlicheres Bild der Tumorentwicklung zu zeichnen. (96 Whibley et al., 2009) Eine Sequenzvariatione im TP53-Gen wurde in der Literatur besonders kontrovers sowohl bezüglich ihrer prognostischen Relevanz als auch der möglichen Beteiligung an der Tumorgenese diskutiert. Der Aminosäure-Polymorphismus P72R (215G>C) (refSNP ID: rs1042522) in Exon 4, der durch Austausch der Base Cytosin durch Guanin zum Austausch der Aminosäure Prolin durch Arginin führt und die Fähigkeit des P53-Proteins zur Induktion der Apoptose in vitro verändern soll. (Dumont et al., 2003) Auch fanden sich Unterschiede in der Fähigkeit bestimmte Komponenten der TranslationsMaschinerie zu binden, die Transkription zu aktivieren oder die Transformation von Stammzellen zu unterdrücken. (Thomas, M. 1999) Auch in der hier untersuchten Patientenkohorte waren 38 der Polymorphismen im exonischen Bereich im Codon 72. insgesamt 53 (71%) Eine andere Studie zeigte, dass das Protein mit der Aminosäure Arginin empfindlicher auf das Onkogen E6 des Humanen Papillomvirus 16 reagiert, was zu Degradation des P53 Proteins führt. Homozygote Träger für Arg72 zeigten ein deutlich erhöhtes Risiko für HPV-assoziierte Zervix-Karzinome. (Storey,A. 1998) Jedoch konnten Metaanalysen und populationsbasierenden Studien diese Erkenntnis nicht bestätigen (Klug, S.J. 2001), (Koushik, A. 2004) und es bleibt weiterhin offen inwieweit die bekannten Polymorphismen auf die Entstehung von Tumoren oder auch einer AML einwirken. 4.4 Genetische Instabilität , Assoziation mit chromosomalen Aberrationen Die Komplexität der Karyotypen, die in der AML mit komplexem Karyotyp beobachtet werden führen zu der Annahme einer zugrundeliegenden genetischen Instabilität. Und obwohl die Karyotypen und deren Aberrationen sehr komplex sind, konnte in vielen Studien ein deutliches Muster der beteiligten Chromosomen erkannt werden. Verschiedene Mechanismen, die zu einem Materialverlust der 53 Chromosomen führen scheinen eine Rolle zu spielen, die vom betroffenen Chromosom abhängig sind. (Schoch,C. 2002) Studien in Hefe haben Gene identifiziert, die, wenn alteriert, zu chromosomaler Instabilität führen. Diese Gene sind involviert in Chromosomenkondensation und Zusammenhalt von Schwesterchromatiden. Die Formation und Dynamik der Mikrotubuli dienen als Kontrollpunkte in der Überwachung des Zellzyklus. Mehrere Gene, die diese Kontrollpunkte regulieren, sind in die Tumorentstehung verwickelt, wie zum Beispiel ATM, BRCA1, RAD51 und TP53. (Lengauer,Christoph 1998) Ein Teil dieser hier durchgeführten Arbeit ist in die Publikation von Rücker et al. eingeflossen (Rücker,F.G. 2012), in der die Frequenz von TP53 Mutationen und die Korrelation mit anderen genetischen Veränderungen und klinischen Parametern in der AML mit komplexem Karyotyp untersucht wurde. Auch wurde die Assoziation zwischen LOH (Loss of heterozygosity) und TP53 Mutationen aber auch die Assoziation von 17p-Deletion und P53 Mutationen untersucht und die Assoziation von TP53 Mutationen mit bestimmten chromosomalen Veränderungen, insbesondere Chromosom 5 und Chromosom 7. Hier zeigte sich ein signifikanter Zusammenhang zwischen dem TP53-Status und Amplifikationen, Deletionen und Zugewinnen genomischen Materials, insbesondere der Chromosomen 5 und 7. Eine AML mit TP53 Alteration wies im Vergleich zu AML mit TP53 Wildtyp eine signifikant höhere Rate an umschriebenen genetischen Veränderungen auf: Verlust von 5q (80 % vs. 37 %), Verlust von 7q (60% vs. 45%), Verlust von 17p (56% vs. 29%), Verlust von 16q (37% vs. 15%), Verlust von 18q (35% vs. 17%), Verlust von 12p (32% vs. 22 %), Zugewinn von 8q (28% vs. 29%), Zugewinn von 11q (33% vs. 16%), Zugewinn von 21q (19% vs. 12%), Zugewinn von 1p (21% vs. 9%), Amplifikation 11q (16% vs. 0%), Amplifikation 21q (13% vs. 4 %), Amplifikation 8q (2% vs. 5 %). Diese Ergebnisse zeigen sehr deutlich, dass TP53 Mutationen mit einer erhöhten genetischen Instabilität assoziiert sind und vor allem die Chromosomenabschnitte 5q, 7q und 17p zu betreffen scheinen. Auch in der Arbeitsgruppe von Schoch et al. wurden 125 Patienten mit AML mit komplexem Karyotyp (zwischen 3-30 chromosomalen Aberrationen pro Fall) in Hinblick auf genetische Abnormalitäten analysiert. Der TP53 Mutationsstatus wurde hierbei jedoch nicht untersucht. Insgesamt wurde auch hier beobachtet, 54 dass ein Verlust von Chromosomenmaterial häufiger war als ein Zugewinn, und dass die am häufigsten deletierten Chromosomen die Bereiche 5q, 7q und 17p betraf. 103 (82%) der Patienten zeigten eine Deletion in 5q, 57 (46%) in 7q und 66 (53%) einen 17p-Verlust. 24 % der Fälle zeigten eine Kombination von 5q, 7q und 17p. Zusätzlich war in 26 % eine 5q-Deletion begleitet von einem 17p Verlust und in 18 % von einem 7q Verlust. Ein 5q Verlust ohne 7q oder 17p Verlust fand sich nur in 15%. Ein 7q Verlust ohne einen 5q oder 17p Verlust in 4%, ein 17p Verlust ohne einen 5q oder 7q Verlust in 3%. (Schoch,C. 2002) In einer weiteren Studie von Schoch et. al. (Schoch,C. 2005) wurde die Rolle des TP53 Gens in der AML mit komplexem Karyotyp untersucht. Dabei konnte in 43 von 49 Fällen (88%) eine TP53 Mutation identifiziert werden. Davon waren 27/30 (90%) Fällen mit Deletion und damit Verlust eines TP53 Allels und 16/19 (84%) ohne Deletion eines TP53 Allels. In einer erweiterten Patientenkohorte wurde der zytogenetische Status bei 565 AML Fällen mit komplexem Karyotyp genauer bestimmt und nach definierten Kriterien in „typische“ und „untypische“ Muster von Chromosomenaberrationen unterteilt. Zu den „typischen“ Chromosomenaberrationen zählte der Verlust von 5q, 7q oder 17p und ein Verlust einer zusätzlichen Region 18q21q22, 12p13 oder 16q22q24 oder ein Gewinn von 11q23q25, 1p33p36, 8q22q24 oder 21q11q22. Dabei fanden sich 102 „atypische“ Formen des komplexen Karyotyps und 465 „typische“ Formen des komplexen Karyotypes. In einer Studie von Haferlach und Kollegen (Haferlach, C. 2008) wurde die Inzidenz von TP53-Mutationen und Deletionen in 107 Patienten mit einer AML mit komplexen Karyotyp analysiert. Von 57 Fällen mit einem Verlust des TP53 Allels konnte in 50 Fällen eine TP53 Mutation im verbliebenen Allel detektiert werden. Auch zeigte sich in 33 von 50 Fällen mit zwei intakten TP53- Allelen eine TP53Mutation, so dass sich insgesamt eine Inzidenz von 78% der TP53 Mutationen ergab. In einer zweiten Patientenkohorte wurde der TP53 Mutationsstatus bei 235 AML Patienten bestimmt, davon 42 mit einer AML und komplexem Karyptyp. Dabei konnten in 33 von 235 Fällen (14%) eine TP53-Mutation nachgewiesen werden. (214 mit einer de-novo AML, 13 t-AML und 8 s-AML) Schliesslich wurden die Fälle der AML mit komplexem Karyotyp in zwei Subgruppen unterteilt. Eine Gruppe mit „typischen“ Deletionen in den Bereichen 5q31, 7q31, 17p13 und in eine Gruppe mit „untypischen“ Deletionen. Die Inzidenz von TP53 Mutationen innerhalb der „typischen“ Gruppe war mit 76 % (20/26) deutlich höher als in der 55 „untypischen“ Gruppe mit 56.2 % (9/16). Über das Alter der Patientenkohorte, dem Zusammenhang mit klinischen Parametern oder einer möglichen therapeutischen Vorbehandlung wurden keine Angaben gemacht. Auch in dieser Kohorte wurde bestätigt, dass die Inzidenz von TP53 Mutationen in einer AML mit komplexem Karyotyp (29/42, 69%) deutlich höher ist, als in Patienten mit einer AML ohne komplexen Karyotyp (4/193, 2.1%). TP53 Mutationen finden sich daneben gehäuft in einer genetischen Subgruppe, die ein bestimmtes Muster von ChromosomenDeletionen aufweist (5q, 7q und 17p). Christansen et al untersuchte 77 Patienten mit t-AML und t-MDS und fand eine Assoziation von TP53-Mutationen mit einer Deletion 5q und einem komplexen Karyotyp, jedoch keine Assoziation mit einer Deletion von 7q. (Christiansen,D.H. 2001) 4.5 Klinische und prognostische Bedeutung von TP53 Mutationen bei der AML mit komplexem Karyotyp TP53 ist das Gen, das in der AML mit komplexem Karyotyp am häufigsten alteriert ist und mit einem höheren Alter, genomischer Komplexität und schlechter Prognose assoziiert ist. Ein grosser Teil dieser hier durchgeführten Arbeit ist in die Publikation von Rücker et al. eingeflossen, in der die Frequenz von TP53 Mutationen/Deletionen und die Korrelation mit anderen genetischen Veränderungen und klinischen Parametern in der AML mit komplexem Karyotyp analysiert wurde. (Rucker,F.G. 2012) Da in der hier vorliegenden Arbeit nicht von allen 234 untersuchten Patienten ausreichend klinische Daten vorlagen, wurden die Analysen auf 155 Patienten beschränkt. Die untersuchte Patientenkohorte ergab, dass Patienten mit TP53 Alterationen Status älter waren (Median 63 Jahre vs. 56 Jahre), im Trend weniger Leukozyten (Median 6,4 % vs. 11,7 %) sowie weniger Blasten im Knochenmark (Median 65 % vs. Median 73 %), und im peripheren Blut (Median 27% vs. 45 %) hatten. Das Ansprechen auf die Therapie ergab signifikante Unterschiede zwischen der mutierten und der Wildtyp Form des TP53. Das Ansprechen auf die Induktionstherapie war jeweils für die Vollremission 27% und 48% (P=.01), für die refraktäre Erkrankung 54% und 34 % (P=.06), für den frühen Tod 21% und 15 % (P=.52) bei Patienten mit TP53 Alterationen und ohne TP53 Alteration. 56 Andere Prädiktoren, die für ein schlechtes Ansprechen auf die Induktionstherapie sprachen, waren das Alter und genetische Verluste, die vor allem Chromosomen 5q, 7q und 16 q betrafen. Die Laktatdehydrogenase und Leukozytenanzahl im Serum beeinflusste nicht das Erreichen der Vollremission. Auch in den Überlebensanalysen ergab sich für die Endpunkte Gesamtüberleben (OS), ereignisfreies (EFS) und rezidivfreies Überleben (RFS) ein signifikanter Unterschied zwischen Patienten mit TP53- Wildtyp und Patienten mit TP53Alteration, die alle mit einem signifikant schlechteren Verlauf assoziiert waren. Bei Patienten mit TP53 Wildtyp betrug das mittlere Gesamtüberleben 10 Monate (bei TP53 Alterationen 4 Monate), das ereignisfreie Überleben 1,3 Monate (bei TP53 Alterationen 1,1 Monate) und das rezidivfreie Überleben 10,3 Monate (bei TP53 Alterationen 5,7 Monate. Mehrere Studien konnten belegen, dass die TP53 Mutationen der stärkste und wichtigste Indikator für das Überleben und die Prognose bei der AML darstellen. So zeigte sich auch bei Stirewalt, dass die TP53-Mutationen nicht mit der peripheren Blastenzahl und der Gesamtleukozytenzahl korrelierten, allerdings gab es eine signifikante Assoziation mit zytogenetischen Aberrationen, „unfavorable cytogenetic risk“ und einem schlechteren Überleben. Zusätzliche Analysen von 109 Patienten mit bekanntem Karyotyp ergaben, dass 6 (55%) der 11 Patienten mit TP53-Mutationen Abnormalitäten sowohl im Chromosom 5 als auch Chromosom 7 hatten. (Stirewalt,D.L.2001) Bowen (Bowen, D.2009) untersuchte die Inzidenz und die prognostischen Auswirkungen von TP53-Mutationen in einer Kohorte von 166 Patienten mit zytogenetischen Aberrationen der Chromosomen 5, 7 oder 17 oder mit komplexem Karyotyp (hier 5 oder mehr Aberrationen), die alle gemäss dem Studienprotokoll des UK Medical Research Council behandelt wurden und eine intensive Chemotherapie erhielten. Insgesamt zeigten 109 der untersuchten Patienten einen komplexen Karyotyp wovon 58 Fälle (53%) eine Mutation im TP53-Gen aufwiesen. Auch in dieser Studie war das Vorliegen einer TP53Mutation mit einer geringeren Vollremissions- (28%) sowie Überlebensrate im Vergleich zu Patienten mit TP53 Wildtyp assoziiert. Schoch et al. (2005) untersuchte die Rolle des TP53 Gens in der AML mit komplexem Karyotyp und setzte sie in Bezug zu klinischen Parametern. Eine 57 Patientenkohorte von 202 Fällen mit komplexem Karyotyp, bei denen klinische Daten vorlagen wurde anhand der Muster bestimmter Chromosomenaberrationen in eine „typische“ (n=117) und „untypische“ (n=25) Gruppe unterteilt und mit klinischen Parametern verglichen. Es zeigte sich ein signifikanter Zusammenhang zwischen dem Gesamtüberleben und der Aberrationsmuster sowie dem Alter aber nicht mit dem Geschlecht sowie der Anzahl der Leukozyten. Die Patienten aus der „typischen“ Patientengruppe waren mit einem mittleren Alter von 64 etwas älter als die Patienten aus der „atypischen“ Patientengruppe mit 62 Jahren und zeigten ein signifikant kürzeres Gesamtüberleben (5.4 vs. 10.2 Monate). 4.6 Methode In der Wahl der Strategien und Methoden für die Identifikation von Mutationen in TP53 erscheint die denaturierende Hochdruckflüssigkeitschromatographie (DHPLC) sehr vielversprechend in Bezug auf ihre Sensitivität und dem Automatisierungspotential. Insgesamt wäre es mittels konventioneller PCR und anschliessender Sequenzierungsmethode sehr aufwendig, grössere Patientenkohorten zu untersuchen. Deshalb bietet sich eine sogenannte prescreening- Untersuchung an, die durch die DHPLC-Methode exzellent gegeben ist, d.h. es ist ein reines Screening-Verfahren, das einen hohen Durchsatz von Proben in einem kurzen Zeitraum erlaubt und alle Proben, die letztendlich ein auffälliges Profil aufweisen, werden dann mittels Sequenzierung auf Vorliegen einer Mutation validiert. Das ist ein effizienter und kostengünstiger Ansatz, der zudem den sensitiven Patientenkohorten erlaubt. 4.7 Nachweis von TP53-Mutationen in großen Ausblick Die Identifizierung molekularer Marker bei der AML hat in den letzten Jahren gezeigt, dass diese Leukämieform eine genetisch äußerst komplexe und heterogene Erkrankung darstellt. Basierend auf der Kenntnis der genetischen Veränderungen kann die AML weiter klassifiziert und eine differenziertere Prognoseeinschätzung durchgeführt werden. Darüber hinaus konnten tiefere Einblicke in das Verständnis der Pathogenese myeloider Erkrankungen gegeben werden, vor allem durch Einsicht in die Interaktion verschiedener Mutationstypen und dem differenzierterem Wissen über einzelne Subtypen. Durch das Verstehen 58 der molekularen Pathogenese könnte es in Zukunft möglich sein, für definierte Subgruppen der AML gezielt Therapien zu entwickeln, die direkt in den Prozess der Leukämogenese eingreifen. Einen ersten Ansatz lieferte zum Beispiel die Beschreibung von PRIMA-1 (p53-dependent reactivation and induction of massive apoptosis) und RITA (reactivation of p53 and induction of tumour cell apoptosis), die zu einer Wiederherstellung der p53-Funktion führen. (Nahi,H. 2008) Durch die Entwicklung und Verabreichung dieser optimierten Therapien mit zielgerichteten Medikamenten und molekularen Angriffspunkten innerhalb der Signaltransduktionswege und Transplantationsstrategien kann man sich in Subgruppen mit derzeit schlechter Prognose eine deutliche Verbesserung der klinischen Resultate erhoffen. (Haferlach,T. 2007), (Haferlach,T. 2008) So spielen TP53-Mutationen bei der AML mit komplexem Karyotyp eine wichtige Rolle, da sie mit einer sehr hohen Inzidenz auftreten und mit sehr komplexen und prognostisch ungünstigen chromosomalen Aberrationen assoziiert sind. Darüber hinaus sind sie mit einem sehr schlechten Gesamtüberleben korreliert und weisen somit eine äusserst schlechte Prognose auf. Im Verlauf zu den TP53-WildtypPatienten ist der Verlauf signifikant schlechter weshalb TP53- Mutationen innerhalb der AML mit komplexen Karyotyp eine Subgruppe von Patienten identifiziert, die eine besonders schlechte Prognose aufweisen. Besonders für diese Patienten sind neue Therapiekonzepte notwendig. 59 5. Zusammenfassung Das TP53 Gen ist ein Transkriptionsfaktor, der die Reparatur der Desoxyribonukleinsäure (DNA) bzw. Apoptose reguliert und dadurch wesentlich zur Aufrechterhaltung der genomischen Stabilität beiträgt. Mutationen finden sich bis zu 50 % in humanen Tumoren, unter anderem von Darm, Leber, Brust und Lunge. Verglichen dazu ist die Inzidenz an TP53 Mutationen in der akut myeloischen Leukämie (AML) mit 2-3 % sehr gering, auffallend jedoch eine bis zu 60 % - Inzidenz bei AML Patienten mit komplexem Karyotyp, die eine besonders ungünstige Prognose und ein schlechtes Ansprechen auf Chemotherapie aufweisen. In dieser Arbeit sollte das TP53 Gen hinsichtlich Inzidenz, Mutationsmuster, Sequenzvarationen, sowie der klinischen Bedeutung in 234 erwachsenen AMLPatienten aus multizentrischen prospektiven Therapiestudien untersucht werden. Hierfür wurden die Patientenproben nach einer Amplifikation auf DNA -Ebene und einer DHPLC (denaturing high performance liquid chromatography) basierten Screening Methode analysiert und Proben mit pathologischen DHPLC-Profilen mittels Sequenzanalyse validiert. Wir fokusierten uns auf die sog. Hot-Spot Region von TP53, repräsentiert durch die Exone 4 bis 10, die ca. 86 % der Mutationen beinhalten. Insgesamt zeigten sich 144 Mutationen in 120 Patienten (51,2 %) mit einem Maximum von 3 Mutationen pro Fall. 114 Patienten hatten keine Mutation. (48,8 %) Von den 144 Mutationen waren 112 Missense Mutationen (78 %), 17 Frame Shift (12%), 7 Nonsense (5%) und 8 Mutationen befanden sich in intronischen Splice-Regionen (5%). Darüber hinaus fanden sich 166 Polymorphismen (71%), 113 davon in intronischen-, 53 in exonischen DNA-Abschnitten. Multiple Mutationen fanden wir mit jeweils drei Mutationen in zwei Patienten (Exon5, Exon 6, Exon 8 und Exon 4, Exon 5, Exon 7) sowie jeweils zwei Mutationen in zwanzig Patienten. Man weiss nur wenig über die Pathogenese der AML mit komplexem Karyotyp, jedoch legt die hohe Frequenz von TP53 Alterationen und bestimmte biallelische Alterationen nahe, dass das Protein P53 eine wichtige Rolle in der Entstehung von Leukämien haben muss. Diese Mutationen im TP53 scheinen mit bestimmten 60 klinischen Charakteristika aber auch dem klinischen Verlauf der AML-Patienten mit komplexen Karyotyp assoziiert zu sein. Die untersuchte Patientenkohorte, von denen ausreichend klinische Daten vorlagen, ergab, dass Patienten mit mutierten TP53 Status älter waren (Median 63 Jahre vs. 56 Jahre), im Trend weniger Leukozyten (Median 6,4 % vs. 11,7 %) sowie weniger Blasten im Knochenmark (Median 65 % vs. Median 73 %), und im peripheren Blut (Median 27% vs. 45 %) hatten. Das Ansprechen auf die Therapie ergab signifikante Unterschiede zwischen der mutierten und der Wildtyp Form des TP53. Das Ansprechen auf die Induktionstherapie war jeweils für die Vollremission 27 % und 48 % (P=.01), für die refraktäre Erkrankung 54% und 34 % (P=.06), für den frühen Tod 21% und 15 % (P=.52) bei Patienten mit TP53 Mutationen und ohne TP53 Mutationen. Andere Prädiktoren, die für eine schlechte Response auf die Induktionstherapie sprachen, waren das Alter und genetische Verluste, die vor allem Chromosomen 5q, 7q und 16 q betrafen. Auch in den Überlebensanalysen ergab sich für die Endpunkte Gesamtüberleben, Ereignisfreies- und Rezidivfreies Überleben ein signifikanter Unterschied zwischen Patienten mit TP53- Wildtyp und Patienten mit TP53Mutation, die alle mit einem signifikant schlechteren Verlauf assoziiert waren. Das mittlere Gesamtüberleben betrug 6 Monate, das mittlere ereignisfreie Überleben lag bei ca. 3,5 Monaten und das mittlere rezidivfreie Überleben bei 6 Monaten für Patienten mit TP53-Mutation. Zukünftige Aufgabe wird es sein, durch die Entwicklung und den Einsatz verfeinerter Methoden den Einblick in die Mechanismen der Leukämieentstehung zu erweitern und basierend auf diesen Erkenntnissen neue zielgerichtete Therapien zu entwickeln. Das Vorliegen einer TP53 Mutation bei AML Patienten mit komplexen Karyotyp erlaubt die Identifizierung von Patienten mit einer besonders ungünstigen Prognose und trägt somit zur Verfeinerung der risiko-basierten Stratifizierung der AML Patienten bei. Die Ergebnisse dieser Arbeit waren eine wesentliche Grundlage für eine umfassene Studie zur Charakterisierung der AML mit komplexem Karyotyp. Erst durch die Generierung dieser Daten konnten wichtige Korrelationen mit klinischen Daten dieser Patienten durchgeführt werden. Anhand dieser Ergebnisse ergeben sich zahlreiche neue Fragestellungen, die es in naher Zukunft zu beantworten gilt, insbesondere die Frage welche Therapie für AML Patienten mit TP53 Mutation den größten Erfolg verspricht. 61 6. Literaturverzeichnis 1. Andersen M K, Johansson B, Larsen S O, Pedersen-Bjergaard J: Chromosomal abnormalities in secondary MDS and AML. Relationship to drugs and radiation with specific emphasis on the balanced rearrangements. Haematologica, 83: 483-488 (1998) 2. Auerbach A D, Allen R G: Leukemia and preleukemia in Fanconi anemia patients. A review of the literature and report of the International Fanconi Anemia Registry. Cancer Genetics & Cytogenetics, 51: 1-121 (1991) 3. Bennett J M, Catovsky D, Daniel M T, Flandrin G, Galton D A, Gralnick H R, Sultan C: Proposed revised criteria for the classification of acute myeloid leukemia. A report of the French-American-British Cooperative Group. Annals of internal Medicine, 103: 620-625 (1985) 4. Bowen D, Groves M J, Burnett A K, Patel Y, Allen C, Green C, Gale R E, Hills R, Linch D C: TP53 gene mutation is frequent in patients with acute myeloid leukemia and complex karyotype, and is associated with very poor prognosis. Leukemia, 23: 203-206 (2009) 5. Byrd JC. Mrozek K. Dodge RK. Carroll AJ. Edwards CG. Arthur DC. Pettenati MJ. Patil SR. Rao KW. Watson MS. Koduru PR. Moore JO. Stone RM. Mayer RJ. Feldman EJ. Davey FR. Schiffer CA. Larson RA. Bloomfield CD. Cancer and Leukemia Group B (CALGB 8461): Pretreatment cytogenetic abnormalities are predictive of induction success, cumulative incidence of relapse, and overall survival in adult patients with de novo acute myeloid leukemia: results from Cancer and Leukemia Group B (CALGB 8461). Blood, 100: 4325-4336 (2002) 6. Castro P D, Liang J C, Nagarajan L: Deletions of chromosome 5q13.3 and 17p loci cooperate in myeloid neoplasms. Blood, 95: 2138-2143 (2000) 7. Cho Y, Gorina S, Jeffrey P D, Pavletich N P: Crystal structure of a p53 tumor suppressor-DNA complex: understanding tumorigenic mutations. Science, 265: 346-355 (1994) 8. Cheson et al. 2003, Cheson BD, Bennett JM, Kopecky KJ, Büchner T, Willman CL, Estey EH, Schiffer CA, Doehner H, Tallman MS, Lister TA, LoCoco F, Willemze R, Biondi A, Hiddemann W, Larson RA, Löwenberg B, Sanz MA, Head DR, Ohno R, Bloomfield CD. Revised recommendations of the International Working Group for Diagnosis, Standardization of Response Criteria, Treatment Outcomes, and Reporting Standards for Therapeutic Trials in Acute Myeloid Leukemia. Journal of Clinical Oncology, 21: 4642-4649 (2003) 9. Christiansen D H, Andersen M K, Pedersen-Bjergaard J: Mutations with loss of heterozygosity of p53 are common in therapy-related myelodysplasia and acute myeloid leukemia after exposure to alkylating agents and significantly associated with deletion or loss of 5q, a complex 62 karyotype, and a poor prognosis. Journal of Clinical Oncology, 19: 14051413 (2001) 10. Curtis R E, Boice J D,Jr, Stovall M, Bernstein L, Greenberg R S, Flannery J T, Schwartz A G, Weyer P, Moloney W C, Hoover R N: Risk of leukemia after chemotherapy and radiation treatment for breast cancer. New England Journal of Medicine, 326: 1745-1751 (1992) 11. Curtis R E, Hoover R N, Kleinerman R A, Harvey E B: Second cancer following cancer of the female genital system in Connecticut, 1935-82. National Cancer Institute Monographs, 68: 113-137(1985) 12. Dash A, Gilliland D G: Molecular genetics of acute myeloid leukaemia. Bailliere's Best Practice in Clinical Haematology, 14: 49-64 (2001) 13. Ding L, Getz G, Wheeler D A, Mardis E R, McLellan M D, Cibulskis K, Sougnez C, Greulich H, Muzny D M, Morgan M B, Fulton L, Fulton R S, Zhang Q, Wendl M C, Lawrence M S, Larson D E, Chen K, Dooling D J, Sabo A, Hawes A C, Shen H, Jhangiani S N, Lewis L R, Hall O, Zhu Y, Mathew T, Ren Y, Yao J, Scherer S E, Clerc K, Metcalf G A, Ng B, Milosavljevic A, Gonzalez-Garay M L, Osborne J R, Meyer R, Shi X, Tang Y, Koboldt D C, Lin L, Abbott R, Miner T L, Pohl C, Fewell G, Haipek C, Schmidt H, Dunford-Shore B H, Kraja A, Crosby S D, Sawyer C S, Vickery T, Sander S, Robinson J, Winckler W, Baldwin J, Chirieac L R, Dutt A, Fennell T, Hanna M, Johnson B E, Onofrio R C, Thomas R K, Tonon G, Weir B A, Zhao X, Ziaugra L, Zody M C, Giordano T, Orringer M B, Roth J A, Spitz M R, Wistuba I I, Ozenberger B, Good P J, Chang A C, Beer D G, Watson M A, Ladanyi M, Broderick S, Yoshizawa A, Travis W D, Pao W, Province M A, Weinstock G M, Varmus H E, Gabriel S B, Lander E S, Gibbs R A, Meyerson M, Wilson R K: Somatic mutations affect key pathways in lung adenocarcinoma. Nature, 455: 1069-1075 (2008) 14. Dohner K, Schlenk R F, Habdank M, Scholl C, Rucker F G, Corbacioglu A, Bullinger L, Frohling S, Dohner H: Mutant nucleophosmin (NPM1) predicts favorable prognosis in younger adults with acute myeloid leukemia and normal cytogenetics: interaction with other gene mutations. Blood, 106: 3740-3746 (2005) 15. Donehower LA, Lozano G. 20 years studying p53 functions in genetically engineered mice. Nature reviews Cancer, 9: 831-841 (2009) 16. Dumont P, Leu J I, Della Pietra A C, 3rd, George D L, Murphy M: The codon 72 polymorphic variants of p53 have markedly different apoptotic potential. Nature Genetics, 33: 357-365 (2003) 17. el-Deiry W S: Regulation of p53 downstream genes. Seminars in Cancer Biology, 8: 345-357 (1998) 18. el-Deiry W S, Kern S E, Pietenpol J A, Kinzler K W, Vogelstein B: Definition of a consensus binding site for p53. Nature Genetics, 1: 45-49 (1992) 63 19. el-Deiry W S, Tokino T, Velculescu V E, Levy D B, Parsons R, Trent J M, Lin D, Mercer W E, Kinzler K W, Vogelstein B: WAF1, a potential mediator of p53 tumor suppression. Cell, 75: 817-825 (1993) 20. Espinosa J M, Verdun R E, Emerson B M: p53 functions through stressand promoter-specific recruitment of transcription initiation components before and after DNA damage. Molecular Cell, 12: 1015-1027 (2003) 21. Estey E, Dohner H: Acute myeloid leukaemia. Lancet, 368: 1894-1907 (2006) 22. Fenaux P, Jonveaux P, Quiquandon I, Lai J L, Pignon J M, LoucheuxLefebvre M H, Bauters F, Berger R, Kerckaert J P: P53 gene mutations in acute myeloid leukemia with 17p monosomy. Blood, 78: 1652-1657 (1991) 23. Frohling S. Skelin S. Liebisch C. Scholl C. Schlenk RF. Dohner H. Dohner K. Acute Myeloid Leukemia Study Group,Ulm: Comparison of cytogenetic and molecular cytogenetic detection of chromosome abnormalities in 240 consecutive adult patients with acute myeloid leukemia. Journal of Clinical Oncology, 20: 2480-2485 (2002) 24. Frohling S, Schlenk R F, Kayser S, Morhardt M, Benner A, Dohner K, Dohner H, German-Austrian AML Study G: Cytogenetics and age are major determinants of outcome in intensively treated acute myeloid leukemia patients older than 60 years: results from AMLSG trial AML HD98-B. Blood, 108: 3280-3288 (2006) 25. Frohling S, Schlenk R F, Stolze I, Bihlmayr J, Benner A, Kreitmeier S, Tobis K, Dohner H, Dohner K: CEBPA mutations in younger adults with acute myeloid leukemia and normal cytogenetics: prognostic relevance and analysis of cooperating mutations. Journal of Clinical Oncology, 22: 624633 (2004) 26. Frohling S, Scholl C, Gilliland D G, Levine R L: Genetics of myeloid malignancies: pathogenetic and clinical implications. Journal of Clinical Oncology, 23: 6285-6295 (2005) 27. Giglia-Mari G, Sarasin A: TP53 mutations in human skin cancers. Human Mutation, 21: 217-228 (2003) 28. Gilliland D G, Griffin J D: The roles of FLT3 in hematopoiesis and leukemia. Blood, 100: 1532-1542 (2002) 29. Gilliland D G, Jordan C T, Felix C A: The molecular basis of leukemia. Hematology, 80-97(2004) 30. Grimwade D, Walker H, Oliver F, Wheatley K, Harrison C, Harrison G, Rees J, Hann I, Stevens R, Burnett A, Goldstone A: The importance of diagnostic cytogenetics on outcome in AML: analysis of 1,612 patients entered into the 64 MRC AML 10 trial. The Medical Research Council Adult and Children's Leukaemia Working Parties. Blood, 92: 2322-2333 (1998) 31. Haferlach C, Dicker F, Herholz H, Schnittger S, Kern W, Haferlach T: Mutations of the TP53 gene in acute myeloid leukemia are strongly associated with a complex aberrant karyotype. Leukemia, 22: 1539-1541 (2008) 32. Haferlach T: Molecular genetic pathways as therapeutic targets in acute myeloid leukemia. Hematology, 400-411(2008) 33. Haferlach T, Bacher U, Haferlach C, Kern W, Schnittger S: Insight into the molecular pathogenesis of myeloid malignancies. Current Opinion Hematology, 14: 90-97 (2007) 34. Haferlach T, Bacher U, Kern W, Schnittger S, Haferlach C: Diagnostic pathways in acute leukemias: a proposal for a multimodal approach. Annals Hematology, 86: 311-327 (2007) 35. Hainaut P, Hernandez T, Robinson A, Rodriguez-Tome P, Flores T, Hollstein M, Harris C C, Montesano R: IARC Database of p53 gene mutations in human tumors and cell lines: updated compilation, revised formats and new visualisation tools. Nucleic Acids Research, 26: 205-213 (1998) 36. Harms K, Nozell S, Chen X: The common and distinct target genes of the p53 family transcription factors. Cellular & Molecular Life Sciences, 61: 822842 (2004) 37. Hasle H, Clemmensen I H, Mikkelsen M: Risks of leukaemia and solid tumours in individuals with Down's syndrome. Lancet, 355: 165-169 (2000) 38. Hayes R B, Yin S N, Dosemeci M, Li G L, Wacholder S, Travis L B, Li C Y, Rothman N, Hoover R N, Linet M S: Benzene and the dose-related incidence of hematologic neoplasms in China. Chinese Academy of Preventive Medicine National Cancer Institute Benzene Study Group. Journal of the National Cancer Institute, 89: 1065-1071 (1997) 39. Hupp T R, Meek D W, Midgley C A, Lane D P: Activation of the cryptic DNA binding function of mutant forms of p53. Nucleic Acids Research, 21: 31673174 (1993) 40. Isobe M, Emanuel B S, Givol D, Oren M, Croce C M: Localization of gene for human p53 tumour antigen to band 17p13. Nature, 320: 84-85 (1986) 41. Ke S H, Wartell R M: Influence of nearest neighbor sequence on the stability of base pair mismatches in long DNA; determination by temperature-gradient gel electrophoresis. Nucleic Acids Research, 21: 5137-5143 (1993) 65 42. Kern W, Voskova D, Schoch C, Schnittger S, Hiddemann W, Haferlach T: Prognostic impact of early response to induction therapy as assessed by multiparameter flow cytometry in acute myeloid leukemia. Haematologica, 89: 528-540 (2004) 43. Kirsch D G, Kastan M B: Tumor-suppressor p53: implications for tumor development and prognosis. Journal of Clinical Oncology, 16: 3158-3168 (1998) 44. Klug S J, Wilmotte R, Santos C, Almonte M, Herrero R, Guerrero I, Caceres E, Peixoto-Guimaraes D, Lenoir G, Hainaut P, Walboomers J M, Munoz N: TP53 polymorphism, HPV infection, and risk of cervical cancer. Cancer Epidemiology, Biomarkers & Prevention, 10: 1009-1012 (2001) 45. Koushik A, Platt R W, Franco E L: p53 codon 72 polymorphism and cervical neoplasia: a meta-analysis review. Cancer Epidemiology, Biomarkers & Prevention, 13: 11-22 (2004) 46. Lane D P: Cancer. p53, guardian of the genome. Nature, 358: 15-16 (1992) 47. Lane D P, Crawford L V: T antigen is bound to a host protein in SV40transformed cells. Nature, 278: 261-263 (1979) 48. Lengauer C, Kinzler K W, Vogelstein B: Genetic instabilities in human cancers. Nature, 396: 643-649 (1998) 49. Leone G, Mele L, Pulsoni A, Equitani F, Pagano L: The incidence of secondary leukemias. Haematologica, 84: 937-945 (1999) 50. Levine A J: p53, the cellular gatekeeper for growth and division. Cell, 88: 323-331 (1997) 51. Liu G, Chen X: DNA polymerase eta, the product of the xeroderma pigmentosum variant gene and a target of p53, modulates the DNA damage checkpoint and p53 activation. Molecular & Cellular Biology, 26: 1398-1413 (2006) 52. Livingstone L R, White A, Sprouse J, Livanos E, Jacks T, Tlsty T D: Altered cell cycle arrest and gene amplification potential accompany loss of wildtype p53. Cell, 70: 923-935 (1992) 53. Lowenberg B: Prognostic factors in acute myeloid leukaemia. Bailliere's Best Practice in Clinical Haematology, 14: 65-75 (2001) 54. Lowenberg B, Downing J R, Burnett A: Medical Progress: Acute Myeloid Leukemia. New England Journal of Medicine, 341: 1051-1062 (1999) 55. Lozano G, Levine A J: Tissue-specific expression of p53 in transgenic mice is regulated by intron sequences. Molecular Carcinogenetics, 4: 3-91 (1991) 66 56. Miyashita T, Reed J C: Tumor suppressor p53 is a direct transcriptional activator of the human bax gene. Cell, 80: 293-299 (1995) 57. Nahi H, Selivanova G, Lehmann S, Mollgard L, Bengtzen S, Concha H, Svensson A, Wiman K G, Merup M, Paul C: Mutated and non-mutated TP53 as targets in the treatment of leukaemia. British Journal Haematology, 141: 445-453 (2008) 58. Oda E, Ohki R, Murasawa H, Nemoto J, Shibue T, Yamashita T, Tokino T, Taniguchi T, Tanaka N: Noxa, a BH3-only member of the Bcl-2 family and candidate mediator of p53-induced apoptosis. Science, 288: 1053-1058 (2000) 59. Ohki R, Nemoto J, Murasawa H, Oda E, Inazawa J, Tanaka N, Taniguchi T: Reprimo, a new candidate mediator of the p53-mediated cell cycle arrest at the G2 phase. The Journal of Biology of Chemistry, 275: 22627-22630 (2000) 60. Olivier M, Eeles R, Hollstein M, Khan M A, Harris C C, Hainaut P: The IARC TP53 database: new online mutation analysis and recommendations to users. Human Mutation, 19: 607-614 (2002) 61. Olivier M, Hollstein M, Hainaut P: TP53 mutations in human cancers: origins, consequences, and clinical use. Cold Spring Harbor perspectives in biology, 2: a001008 (2010) 62. Onuchic L F, Furu L, Nagasawa Y, Hou X, Eggermann T, Ren Z, Bergmann C, Senderek J, Esquivel E, Zeltner R, Rudnik-Schoneborn S, Mrug M, Sweeney W, Avner E D, Zerres K, Guay-Woodford L M, Somlo S, Germino G G: PKHD1, the polycystic kidney and hepatic disease 1 gene, encodes a novel large protein containing multiple immunoglobulin-like plexintranscription-factor domains and parallel beta-helix 1 repeats. American Journal of Human Genetics, 70: 1305-1317 (2002) 63. Owen-Schaub L B, Zhang W, Cusack J C, Angelo L S, Santee S M, Fujiwara T, Roth J A, Deisseroth A B, Zhang W W, Kruzel E: Wild-type human p53 and a temperature-sensitive mutant induce Fas/APO-1 expression. Molecular & Cellular Biology, 15: 3032-3040 (1995) 64. Pabst T, Mueller B U, Zhang P, Radomska H S, Narravula S, Schnittger S, Behre G, Hiddemann W, Tenen D G: Dominant-negative mutations of CEBPA, encoding CCAAT/enhancer binding protein-alpha (C/EBPalpha), in acute myeloid leukemia. Nature Genetics, 27: 263-270 (2001) 65. Pedersen-Bjergaard J, Rowley J D: The balanced and the unbalanced chromosome aberrations of acute myeloid leukemia may develop in different ways and may contribute differently to malignant transformation. Blood, 83: 2780-278 (1994) 67 66. Pfeifer G P, Besaratinia A: Mutational spectra of human cancer. Human Genetics, 125: 493-506 (2009) 67. Prives C, Bargonetti J, Farmer G, Ferrari E, Friedlander P, Wang Y, Jayaraman L, Pavletich N, Hubscher U: DNA-binding properties of the p53 tumor suppressor protein. Cold Spring Harbour Symposia on Quantitave Biology, 59: 207-213 (1994) 68. Resnick M A, Inga A: Functional mutants of the sequence-specific transcription factor p53 and implications for master genes of diversity. Proceedings of the National Academy of Sciences of the United States of America, 100: 9934-9939 (2003) 69. Roemer K: Mutant p53: gain-of-function oncoproteins and wild-type p53 inactivators. Biological Chemistry, 380: 879-887 (1999) 70. Roemer K, Mueller-Lantzsch N: p53 transactivation domain mutant Q22, S23 is impaired for repression of promoters and mediation of apoptosis. Oncogene, 12: 2069-2079 (1996) 71. Rücker F G, Bullinger L, Schwaenen C, Lipka D B, Wessendorf S, Frohling S, Bentz M, Miller S, Scholl C, Schlenk R F, Radlwimmer B, Kestler H A, Pollack J R, Lichter P, Dohner K, Dohner H: Disclosure of candidate genes in acute myeloid leukemia with complex karyotypes using microarray-based molecular characterization. Journal of Clinical Oncology, 24: 3887-3894 (2006) 72. Rücker FG, Schlenk RF, Bullinger L, Kayser S, Teleanu V, Kett H, Habdank M, Kugler CM, Holzmann K, Gaidzik VI, Paschka P, Held G, von LilienfeldToal M, Lübbert M, Fröhling S, Zenz T, Krauter J, Schlegelberger B, Ganser A, Lichter P, Döhner K, Döhner H. TP53 alterations in acute myeloid leukemia with complex karyotype correlate with specific copy number alterations, monosomal karyotype, and dismal outcome. Blood, 119: 21142121.(2012) 73. Sachidanandam R, Weissman D, Schmidt S C, Kakol J M, Stein L D, Marth G, Sherry S, Mullikin J C, Mortimore B J, Willey D L, Hunt S E, Cole C G, Coggill P C, Rice C M, Ning Z, Rogers J, Bentley D R, Kwok P Y, Mardis E R, Yeh R T, Schultz B, Coja. A map of human genome sequence variation containing 1.42 million single nucleotide polymorphisms. Nature, 409: 92833 (2001) 74. Song H., Hollstein M, Xu Y. p53 gain-of-function cancer mutants induce genetic instability by inactivating ATM. Nature Cell Biology 9: 573-580 (2007) 75. Sakamuro D, Sabbatini P, White E, Prendergast G C: The polyproline region of p53 is required to activate apoptosis but not growth arrest. Oncogene, 15: 887-898 (1997) 68 76. Sanger F, Nicklen S, Coulson A R: DNA sequencing with chain-terminating inhibitors. Proceedings of the National Academy of Sciences of the United States of America, 74: 5463-5467 (1977) 77. Schessl C, Rawat V P, Cusan M, Deshpande A, Kohl T M, Rosten P M, Spiekermann K, Humphries R K, Schnittger S, Kern W, Hiddemann W, Quintanilla-Martinez L, Bohlander S K, Feuring-Buske M, Buske C: The AML1-ETO fusion gene and the FLT3 length mutation collaborate in inducing acute leukemia in mice. The Journal of Clinical Investigation, 115: 2159-2168 (2005) 78. Schlenk R F, Dohner K, Krauter J, Frohling S, Corbacioglu A, Bullinger L, Habdank M, Spath D, Morgan M, Benner A, Schlegelberger B, Heil G, Ganser A, Dohner H, German-Austrian Acute Myeloid Leukemia Study,Group: Mutations and treatment outcome in cytogenetically normal acute myeloid leukemia. New England Journal of Medicine, 358: 19091918(2008) 79. Schmitt T, Misi L: Single Nucleotide Polymorphism (SNP), Insertion & Deletion Detection on the Wave Nucleic Acid Fragment Analysis System. Transgenomic: 1-4 (2000) 80. Schoch C, Haferlach T, Bursch S, Gerstner D, Schnittger S, Dugas M, Kern W, Loffler H, Hiddemann W: Loss of genetic material is more common than gain in acute myeloid leukemia with complex aberrant karyotype: a detailed analysis of 125 cases using conventional chromosome analysis and fluorescence in situ hybridization including 24-color FISH. Genes Chromosomes Cancer, 35: 20-29 (2002) 81. Schoch C, Kern W, Kohlmann A, Hiddemann W, Schnittger S, Haferlach T: Acute myeloid leukemia with a complex aberrant karyotype is a distinct biological entity characterized by genomic imbalances and a specific gene expression profile. Genes Chromosomes Cancer, 43: 227-238 (2005) 82. Schoch C, Kern W, Krawitz P, Dugas M, Schnittger S, Haferlach T, Hiddemann W: Dependence of age-specific incidence of acute myeloid leukemia on karyotype. Blood, 98: 3500 (2001) 83. Schoch C, Haferlach T, Haase D, Fonatsch C, Loffler H, Schlegelberger B, Staib P, Sauerland M C, Heinecke A, Buchner T, Hiddemann W, for the German AML Cooperative Study Group: Patients with de novo acute myeloid leukaemia and complex karyotype aberrations show a poor prognosis despite intensive treatment: a study of 90 patients. British Journal of Haematology, 112: 118-126 (2001) 84. Slovak M L, Kopecky K J, Cassileth P A, Harrington D H, Theil K S, Mohamed A, Paietta E, Willman C L, Head D R, Rowe J M, Forman S J, Appelbaum F R: Karyotypic analysis predicts outcome of preremission and postremission therapy in adult acute myeloid leukemia: a Southwest Oncology Group/Eastern Cooperative Oncology Group Study. Blood, 96: 4075-4083 (2000) 69 85. Soussi T, Caron de Fromentel C, May P: Structural aspects of the p53 protein in relation to gene evolution. Oncogene, 5: 945-952 (1990) 86. Soussi T, Dehouche K, Beroud C: p53 website and analysis of p53 gene mutations in human cancer: forging a link between epidemiology and carcinogenesis. Human Mutations, 15: 105-113 (2000) 87. Stirewalt D L, Kopecky K J, Meshinchi S, Appelbaum F R, Slovak M L, Willman C L, Radich J P: FLT3, RAS, and TP53 mutations in elderly patients with acute myeloid leukemia. Blood, 97: 3589-3595 (2001) 88. Storey A, Thomas M, Kalita A, Harwood C, Gardiol D, Mantovani F, Breuer J, Leigh I M, Matlashewski G, Banks L: Role of a p53 polymorphism in the development of human papillomavirus-associated cancer. Nature, 393: 229234 (1998) 89. Swerdlow, SH, Campo, E, Harris, NL et al: WHO Classification of Tumours of Haematopoietic and Lymphoid Tissues. Lyon, France, International Agency for Research Press, 4: 1-439 (2008) 90. Thomas M, Kalita A, Labrecque S, Pim D, Banks L, Matlashewski G: Two polymorphic variants of wild-type p53 differ biochemically and biologically. Molecular & Cellular Biology, 19: 1092-1100 (1999) 91. Unger T, Nau M M, Segal S, Minna J D: p53: a transdominant regulator of transcription whose function is ablated by mutations occurring in human cancer. The EMBO Journal , 11: 1383-1390 (1992) 92. Vogelstein B, Lane D, Levine A J: Surfing the p53 network. Nature, 408: 307-310 (2000) 93. Wattel E, Preudhomme C, Hecquet B, Vanrumbeke M, Quesnel B, Dervite I, Morel P, Fenaux P: p53 mutations are associated with resistance to chemotherapy and short survival in hematologic malignancies. Blood, 84: 3148-3157 (1994) 94. Wei C L, Wu Q, Vega V B, Chiu K P, Ng P, Zhang T, Shahab A, Yong H C, Fu Y, Weng Z, Liu J, Zhao X D, Chew J L, Lee Y L, Kuznetsov V A, Sung W K, Miller L D, Lim B, Liu E T, Yu Q, Ng H H, Ruan Y: A global map of p53 transcription-factor binding sites in the human genome. Cell, 124: 207-219 (2006) 95. Weisz L, Oren M, Rotter V: Transcription regulation by mutant p53. Oncogene, 26: 2202-2211 (2007) 96. Whibley C, Pharoah PD, Hollstein M.: p53 polymorphisms: cancer implications. National Review Cancer, 9:95-107 (2009) 70 97. Zuber J, Radtke I, Pardee TS, et al. Mouse models of human AML accurately predict chemotherapy response. Genes &Development, 23: 877889 (2009) 71 7. Anhang Reagenzien 10× Optimase-PCR-Puffer Fa. Qiagen 10× PCR-Puffer Fa. Qiagen, Hilden ABI Prism 3100 Genetic Analyzer 3130xl Fa. Applied Biosystems, Foster city, USA ABI Big Dye Terminator Cycle Sequencing Kit Agarose Fa. Applied Biosystems, Foster City, USA Sigma Aldrich Chemie GmbH Aqua destillata Fa. Braun, Melsungen Dye Ex Spin Kit Fa. Qiagen, Hilden Ethidiumbromid Fa. Appli Chem GeneAmp PCR System 2700 High melting standard (Wave High-Range Mutation Control Standard): Fa. Applied Biosystems, Foster city, USA HPLC-Wasser Injektionsspritzenwaschlösung (syringe washing solution) Low melting standard (Wave Low-Range Mutation Control Standard) Merck Darmstadt Fa. MD. Transgenomic Inc, Omaha, NE, USA MgSO4 (25mM) Fa.MD Transgenomic Inc, Omaha, NE, USA DNA-ladder, 100bp-Längenstandard (500µl/ml) Elektrophorese-Kammer Gel-Loading-Buffer (10×Blue-Juice) Hot Star Taq Polymerase (5U/µl MgCl2 (25mM) Fa. New England Biolaps MBT Brand, Gießen Fa. Invitrogen, Karlsruhe Fa. MD Transgenomic Inc, Omaha, NE, USA Fa. Qiagen, Hilden Fa. MD Transgenomic Inc, Omaha, NE, USA Fa. Qiagen, Hilden Mikrowelle Moulinex München Optimase-Polymerase: (2,5 U/µl) Fa. Optimase Transgenomic, Omaha, NE,USA Nukleotide: dATP, dCTP, dGTP, dTTP (25mM) Pipetten (20µl, 100µl, 200µl, 1000µl) Fa. Roche, Mannheim Abimed, Langenfeld Pipettenspitzen mit Filter (steril) Gilson, Middleton, USA Puffer A Fa. MD Transgenomic Inc, Omaha, NE, USA Puffer D (Wave Optimized Solution D Fa. MD. Transgenomic Inc, Omaha, NE, USA QIAquick PCR Purifikation Kit Fa. Qiagen, Hilden Primer Puffer B Q Solution Sequenzierpuffer (5×) Sizing standard (Wave DNA Sizing Control) MWG Biotech Ebersberg Fa. MD Transgenomic Inc, Omaha, NE, USA Fa. Qiagen, Hilden Fa. Applied Biosystems, Foster city, USA Software des Wave TM 3500HT System Fa. MD. Transgenomic Inc, Omaha, NE, USA Navigator-Software: Fa. MD Transgenomic Inc, Omaha, NE, USAW Thermocycler Basic 96 SC-TCSQB SensoQuest, Göttingen TAE-Puffer Waage Wave TM 3500HT System USB, Cleveland, USA Precisa 220 M Fa. MD Transgenomic Inc, Omaha, NE, USA 72 Abbildungsverzeichnis Abbildung 1: Abbildung 2: Abbildung 3: Abbildung 4: Abbildung 5: Abbildung 6: Abbildung 7: Abbildung 8: Abbildung 9: Prävalenz von TP53 Mutationen in unterschiedlichen Tumoren entsprechend der internationalen Datenbank der Krebsforschung .................................................................................................... 11 Aufgetragenes Gelbild gelelektrophoretisch separierter DNAFragmente nach der Polymerase-Kettenreaktion. ...................... 20 Schematische Darstellung der Bildung von Homo- und Heteroduplices. ........................................................................... 22 DHPLC Chromatogramme .......................................................... 24 Prozentuale Verteilung der Mutationstypen im TP53 .................. 32 Abbildung von 136 TP53 Mutationen von Exon 4 – Exon 10: ..... 36 Beispiel einer Missense Mutation: Heterozygote Missense Mutation im Codon 272 in Exon 8 ............................................... 36 Chromatogramm; Mutation P: Val 272 Leu (GTG>TTG), Heterozygot bei 59,0°C ............................................................... 37 Chromatogramm; Mutation P: Val 272 Leu (GTG>TTG)............. 37 Abbildung 10: Mutationsmuster der Missense-Mutationen im TP53 .................. 38 Abbildung 11: Beispiel 1: Frame-Shift Mutation, Deletion 1, Codon 107........... 40 Abbildung 12: Beispiel 2: Frame Shift Mutation, Deletion 8bp , Codon 172 ....... 40 Abbildung 13: Häufigkeitsverteilung der Mutationen in Bezug auf die Codons .. 43 Abbildung 14: Häufigkeitsverteilung der Mutationen auf die Exone ................... 44 Abbildung 15: Häufigkeitsverteilung der Polymorphismen ................................. 44 Abbildung 16: Korrelation des TP53 Status mit genomischen Veränderungen.. 46 Abbildung 17: Korrelation des TP53 Status mit Verlust bzw. Zugewinn von genomischen Material ................................................................. 47 Abbildung 18: Überlebensanalysen der Patienten mit Mutation im TP53 im Vergleich zu Patienten ohne Mutation innerhalb eines komplexen Karyotyps bei Patienten mit Akuter myeloischer Leukämie. ....... 48 73 8. Lebenslauf Der Lebenslauf wurde aus Gründen des Datenschutzes entfernt. 74 Der Lebenslauf wurde aus Gründen des Datenschutzes entfernt. 75 Teile dieser Dissertation wurden bereits in folgendem Zeitschriftenartikel veröffentlicht: Rücker FG, Schlenk RF, Bullinger L, Kayser S, Teleanu V, Kett H, Habdank M, Kugler CM, Holzmann K, Gaidzik VI, Paschka P, Held G, von Lilienfeld-Toal M, Lübbert M, Fröhling S, Zenz T, Krauter J, Schlegelberger B, Ganser A, Lichter P, Döhner K, Döhner H. TP53 alterations in acute myeloid leukemia with complex karyotype correlate with specific copy number alterations, monosomal karyotype, and dismal outcome. Blood, 119: 2114-2121.(2012) 76