Nils Johnsson liefert Bilder für das spätere "Proteinkino"
Werbung

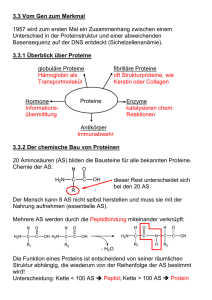
Powered by Seiten-Adresse: https://www.gesundheitsindustriebw.de/de/fachbeitrag/aktuell/nils-johnsson-liefert-bilderfuer-das-spaetere-proteinkino/ Nils Johnsson liefert Bilder für das spätere "Proteinkino" Nils Johnsson betreibt Kommunikationsforschung am lebenden Objekt. Der 48-jährige Biochemiker untersucht, wie Proteine untereinander den komplizierten Prozess der Zytokinese, der Teilung des Zellplasmas, bewerkstelligen. Dank einer eigens entwickelten Methode kann Johnsson messen, wenn Proteine miteinander in Wechselwirkung stehen. Seit kurzem ist der Wissenschaftler wieder dort gelandet, wo seine akademische Laufbahn begann, in Baden-Württemberg. In Tübingen begann Johnsson sein Studium, ehe er am MaxPlanck-Institut (MPI) für biophysikalische Chemie in Göttingen bei Prof. Klaus Weber diplomierte und promovierte. Dort, erzählt Johnsson, schnitt er im Schlachthof Därme auf, kratzte Zellen ab und reinigte Protein auf für seine Doktorarbeit. Schon damals wollte der junge Wissenschaftler Proteine in ihrer natürlichen Umgebung, in der Zelle, untersuchen. Das führte ihn zum Hefezellenforscher Alexander Varshavsky an das MIT und dann zum California Institute of Technology. Einer der ersten „Mieter“ im neuen Forschungszentrum Prof. Dr. Nils Johnsson (Foto: Pytlik) 1995 kehrte er nach Deutschland zurück ans MPI für Züchtungsforschung, wo er seine 1 Arbeiten fortführte - die Charakterisierung von Proteinen in der lebenden Zelle (Hefe) - und seine dann patentierte Methode zu Ende entwickelte. Nach sechs Jahren wechselte er an das Institut für Genetik und Toxikologie am Karlsruher Forschungszentrum. 2005 ging er dann als W2-Professor ans Zentrum für Molekularbiologie der Entzündung der Uni Münster. Vor kurzem ist Johnsson mit seiner Familie nach Ulm gezogen, wo er am neuen lebenswissenschaftlichen Forschungszentrum das Institut für Molekulare Genetik und Zellbiologie leitet und schon eine Zusammenarbeit mit seinem „Nachbarn“ Lenhard Rudolph begonnen hat. „Chemie im kleinsten Raum“ Als Grundlagenforscher kennt Johnsson zwar die Mühen des Sisyphus. Doch sein Forschungsgegenstand - die Proteine - begeistert ihn nach wie vor. Ihn fasziniert, wie diese molekularen Maschinen auf kleinstem Raum arbeiten, mit welcher Präzision und unter welch erschwerten Bedingungen. „In der Zellbiologie ist das Chemie im kleinsten Raum, im flüssigen Medium. Da staunt jeder Elektroniker, wenn der sieht, wie mit mobilen Elementen robuste und flexible Schaltkreise aufgebaut sind“. Im Visier: Proteine an den Schaltstellen Eine frisch abgespaltene Tochterzelle. Die Kappe (blau) der Zelle wird von dem Protein gebildet, das während der Zellteilung in die zwischen Mutter- und Tochterzelle neu gebildete Zellwand eingebaut wurde. Der rote und grüne Abschnitt der Zelle markieren die Synthese des Proteins während früherer Phasen des Zellwachstums. (Foto: Johnsson) In Johnssons Blickpunkt steht die Zytokinese, die Teilung des Zellplasmas. Der Vorgang selbst, der aus mehreren Prozessen besteht, sei für sich genommen schon komplex. Johnsson will erforschen, wie diese Abläufe miteinander verschaltet sind, welche Proteine an diesen Schaltstellen sitzen. Aktuell erstellt seine Arbeitsgruppe eine Art Karte der an der Zytokinese beteiligten Proteine , um dann diejenigen Mitglieder dieser Proteinkarte zu identifizieren, die an 2 Schaltstellen sitzen. Deren Interaktion wird nicht nur genetisch, sondern auch biochemisch und zellbiologisch untersucht. Wie koordinieren Proteine diese komplexen Prozesse? Während der Zellteilung müssen die beiden Zellen darauf achten, dass die Plasmamembran nicht reißt. Das besorgen so genannte Sensoren, die diesen Vorgang stets beobachten und vermutlich Haltsignale zum Zytokineseapparat senden, wenn der Vorgang aus dem Ruder zu laufen droht. „Da gibt es Sensoren und dahinter geschaltete Signalkaskaden, die mit anderen Mitgliedern der Zytokinese sprechen müssen, um diesen Vorgang zu koordinieren.“ Diese über Proteine an diesen Schaltstellen vermittelte Koordination ist nach Johnssons Worten noch nicht richtig verstanden. Dabei bedient sich Nils Johnsson einer Art genetischer Methode, die die Interaktion zwischen den Genprodukten misst. Mit dem Verfahren, das sich die Technik des so genannten SplitUbiqutins zunutze macht, lassen sich sehr viele Interaktionen gleichzeitig und auch dort, wo sie stattfinden, messen. Das hat Vorteile für die Forscher: „Wir können Proteine , von denen man weiß, dass sie miteinander agieren sollten, untersuchen, aber auch nach neuen Interaktionspartnern fahnden.“ Das Split Ubiquitin Verfahren: Die Proteine X1 und X2 wurden an die N- (Nub) und C-terminale Hälfte (Cub) des Ubiquitins (Ub) gekoppelt. Sobald X1 und X2 aneinander binden, wird aus den beiden Hälften ein intaktes UbiquitinMolekül gebildet, woraufhin der Reporter abgespaltet wird. Das freigesetzte Reporterprotein dient als Signal für die erfolgte Proteininteraktion. (Abbildung: Johnsson) Starres Bild des dynamischen Netzwerks Dahinter steckt die Überzeugung, dass alle zellulären Prozesse von Proteinen und den Interaktionen zwischen ihnen gesteuert werden. Die von Johnsson angewandte Methode ist eine Art Einstiegspunkt, liefert zunächst nicht mehr als ein starres Bild von einem dynamischen und sehr flexiblen Netzwerk. Denn die Methode liefert nur qualitative, keine quantitativen Daten, sagt nichts über die Bindungsstärke beider Proteine und auch nichts über den Zeitpunkt und die Dauer der Wechselwirkung während des Zellzyklus. 3 Diese Hefen auf der Wachstumsplatte synthetisieren jeweils eine unterschiedliche Kombination von Nub und Cub Fusionsporteinen. Sobald eine Hefezelle ein Paar interagierender Proteine synthetisiert, sichert der freigesetzte Reporter das Überleben der Zellen auf einem speziellen Selektionsmedium. 96 verschiedene Proteinpaare werden so gleichzeitig auf ihre Wechselwirkungen getestet. Hier zeigen drei eine deutliche Interaktion. Die untere Abbildung zeigt das Wachstum der Hefen auf einem Medium ohne Proteininteraktionsmessung. (Foto: Johnsson) Basis für weitere Untersuchungen Die Informationen dienen als Grundlage für weitere, zum Beispiel proteinbiochemische Untersuchungen, mit der die Affinitäten der beteiligten Akteure gemessen werden, mit dem Ziel herauszufinden, welche Aufgabe ihnen in der Zytokinese zufällt. In einem weiteren Schritt versucht Johnsson die Allele der miteinander agierenden Gene zu finden, die verhindern, dass diese beiden Proteine miteinander agieren. Dann lässt sich messen, welche Auswirkung diese fehlende Interaktion auf den Prozess hat, und aus diesem Umkehrschluss versuchen die Ulmer Forscher mechanistisch herauszufinden, wie dieser Prozess tatsächlich abläuft. „Sie kommen mit einer Perspektive nicht aus“ Für das molekulare Verständnis der komplexen Proteinmechanismen in Raum und Zeit muss nach Johnssons Worten das Arsenal biomolekularer Bildgebungsverfahren und anderer Methoden zusammengeschaltet werden. Man muss sich das wohl wie die Arbeit eines riesigen Puzzles vorstellen, wenn Johnsson davon spricht, dass unterschiedliche Methoden unterschiedliche Ergebnisse bringen, die sich nicht ausschließen, sondern sich wahrscheinlich ergänzen. 4 Hefezellen sind der perfekte Organismus Johnsson erforscht diese grundlegenden zellulären Vorgänge in Hefezellen, wohl wissend, dass dieser Organismus eher im Schatten des öffentlichen Interesses steht. Das ficht den Biologen nicht an, denn Saccharomyces cerevisiae ist der „perfekte Organismus zum Ausprobieren“. Nirgendwo gehe das so schnell und effizient, weil die Hefezelle sich schnell regeneriere, viele ihrer Prozesse denen humaner ähneln und sie sich genetisch ausgezeichnet manipulieren lasse. Das Hefe-Proteom, so Johnssons Plädoyer, sei das bestbeschriebene aller Organismen. „Die Hefe ist immer einen Schritt voraus“, will heißen, das Maß der Unsicherheit ist vergleichsweise gering, weil das Genom relativ einfach sei. Einige Hauptverdächtige sind schon bekannt Johnssons Arbeitsgruppe untersucht rund 400 Proteine , das ist ein knappes Zehntel des HefeProteoms. Mit diesem Ansatz liegen die Ulmer mittendrin, zwischen dem proteomweiten Ansatz und dem ganz weniger Proteine . Obwohl die Zytokinese-Proteinfamilie immer neue Mitglieder erhält und damit auch die Anzahl der zu bearbeitenden Proteine wächst („Das ist ein bisschen Sisyphus-Arbeit“), haben Johnsson und Mitarbeiter schon einige Hauptverdächtige aufgespürt, wie beispielsweise das CDC 24 Protein oder Zellstress-Sensoren. „Es gibt fast schon zu viele interessante Proteine , als dass sie eine Arbeitsgruppe bearbeiten könnte“, bedauert Johnsson. Schwieriger Umgang mit der Datenmenge Damit rührt der Endvierziger an ein Problem, mit dem er und seine Kollegen kämpfen: mit der unheimliche Datenmenge. Die Erstellung von Daten sei wichtig, aber fast noch schwieriger und genauso wichtig sei der vorsichtige Umgang mit den Daten, sagt Johnsson, spricht von artifiziellen Interaktionen und weißen Flecken auf der (Protein-)Karte, die man als solche nicht erkennt. „Das ist ein iterativer Prozess“. Mit seiner Arbeitsgruppe will Johnsson Licht ins Dunkel der Zytokinese bringen und hofft auf zahlreiche Folgeuntersuchungen. Da die Zellteilung ein universaler Prozess ist, erhoffen sich Johnsson und Kollegen Aufschlüsse über diese Prozesse in höheren Zellen. So wird auch der Einfluss einer gestörten Zellteilung mit einigen Krebsformen in Verbindung gebracht. Kleine Erfolge motivieren Dass der Erkenntnisfortschritt ein Eichhörnchen ist, wissen Grundlagenforscher wie Johnsson nur zu gut. Ihn motiviert, wenn erste Erfolge sichtbar werden, wenn sich Hypothesen bestätigen. „Das hält einen aufrecht“. Indessen wissen gerade Zellbiologen wie Johnsson um die ungeheure Komplexizität des zellulären Geschehens. Ihm fallen da schnell die Lipide der Zellmembran ein, die Proteincluster organisieren. In Ulm lebt sich Johnsson gerade ein. Nach einer „relativ turbulenten Zeit“, wie er es selbst 5 nennt, bereitet er Vorlesungen und Praktika vor, baut seine Arbeitsgruppe weiter aus und hat sich fest vorgenommen, in dem lebenswissenschaftlichen Forschungsgebäude die Zusammenarbeit zu suchen. Dass Hefezellbiologen wie er Methoden fürs „Proteinkino“ entwickeln, und damit über Werkzeuge verfügen, die auch andere Forscher interessieren, weiß Johnsson. Und dass Kooperation immer dann gelingt, wenn nur einige Türe dazwischen liegen, weiß der erfahrene Forscher auch. wp - 08.04.08 © BIOPRO Baden-Württemberg GmbH Fachbeitrag 11.03.2008 BioRegionUlm 6