Fragen zu Kapitel 2
Werbung

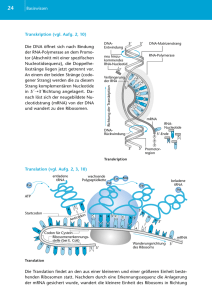
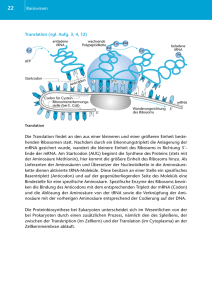
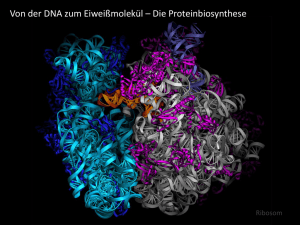
Molekulare Biologie der Zelle Kapitel 6A: Transkription (RNA-Synthese) Fragen zu Kapitel 6A: Transkription (RNA-Synthese) 1. Wie reguliert eine Zelle die Konzentration eines bestimmten Proteins? Hauptsächlich über die Syntheserate der entsprechenden mRNA. Transkription reguliert, Translation nicht reguliert 2. Strukturelle Unterschiede zwischen DNA und RNA. Rückrat: DNA 2’-Deoxyribose, RNA „richtige“ Ribose-Einheit mit 2’-OH-Gruppe Basenpaare: DNA A-T, RNA A-U 3. Die Initiation der Transkription beim Bakterium (S.11-16). Gen-Anfang auf der DNA, ist durch den Promotor gekennzeichnet; 2 charakteristische Sequenz-Blöcke werden durch den -Faktor der RNA-Polymerase erkannt öffnet den Doppelstrang 10 BP weiter beginnt Transkription 4. Die bakterielle RNA-Polymerase in voller Aktion (S.8). ganzer DNA-Strang läuft in die RNA-Polymerase hinein und hinten wieder unverändert hinaus, in der Mitte nur kleinräumig geöffnet aus separaten Kanal gleitet neuer RNA-Einzelstrang aus Enzym heraus im Zentrum der RNA-Polymerase wird die RNA an ihrem 3’-Ende stetig verlängert nur die frisch angehängten Basen bleiben kurz mit dem DNA-Vorlagestrang gepaart (~10BP) 5. Der Abschluss der Transkription beim Bakterium (S.11-12). Sequenz sehr stabile Haarnadelschleife im Austrittkanal Nase von Polymerase wieder hochgeklappt Konformationsänderung des Enzyms und Bremsung Abschluss-Sequenz wo Basen jeweils nur zwei H-Brücken mit Vorlagestrang Dissoziation der fertigen RNA aus der RNA-Polymerase 6. Welche RNA-Polymerasen haben Eukaryonten, was leisten sie jeweils? RNA-Polymerase I : rRNA-Moleküle, die größten aller RNA-Moleküle RNA-Polymerase II: hauptsächlich mRNA (mittelgroße) RNA-Polymerase III: die kleinsten RNA-Sorten (tRNA und andere kleine RNAs) 7. Die Initiation der Transkription von mRNA beim Eukaryonten: a) Wie heißt der wichtigste Sequenzblock des Promotors? b) Was tut TFIID mit TBP? a) TATA-Box b) TFIID bindet an den Promotor, wobei die Untereinheit TBP an die TATA-Box bindet . Auch INR und DPE werden vom TFIID miterkannt. 8. Welche Aktionen werden von TFIIH ausgeführt? Welches Reagenz ist dazu nötig? spaltet DNA Doppelhelix unter ATP-Verbrauch phosphoryliert mittels ATP den C-Terminalen Schwanz der RNA-Polymerase II an mehreren Stellen nötig: ATP 9. Was tun die generellen Transkriptionsfaktoren, was die speziellen Transkriptionsfaktoren, wie wirken sie zusammen? generellen bewirken das der Promotor zu arbeiten beginnt speziellen dienen der Regulation der Gen-Expression (Ein- und Ausschalten der Transkription) spezielle Transkriptionsfaktoren binden an Gen-regulatorischen Sequenzen oberhalb des Promotors Mediator-Protein lockt dann die TFII-Komponente + RNA-Polymerase II + Histon-Acetylase + Chromatin-Remodeling-Koplex zum Promotor 10. Welche Proteine kommen der RNA-Polymerase II zu Hilfe, damit sie auch entlang der Perlschnur-DNA transkribieren kann? Chromatin-Remodeling-Komplex + Histion-Acetylase Bernhard Polzinger WS 2004/2005 Seite 1/4 Molekulare Biologie der Zelle Kapitel 6A: Transkription (RNA-Synthese) lockern die DNA von den Nucleosomen frei schlotternde DNA-Schleifen um Nucleosomen 11. Warum tritt bei der Transkription ein Windungsproblem auf? a) bei Prokaryonten b) bei Eukaryonten Transkription verursacht stete Öffnung der DNA-Doppelhelix und wieder verschließen DNADoppelhelix ist eine Schraube Rotation um Längsachse zwei Möglichkeiten: RNAPolymerase dreht sich oder Doppelstrang a) bei Prokaryonten: RNA-Polymerase dreht sich nicht da sie viel massiver als der lange, filigrane DNA-Doppelstrang; b) bei Eukaryonten: wegen der Nucleosomen nicht möglich. 12. Welche Unterschiede gibt es zwischen Pro-/Eukaryonten hinsichtlich a) Ort der Transkription in der Zelle b) Prozessierung des Transkripts c) Codierung von zusammengesetzten Proteinen (Untereinheiten, Domänen) Prokaryonten a) im Cytosol b) da nur ein Kompartiment beginnt die Proteinsynthese noch bevor die mRNA ganz fertig ist c) oft mehrere Proteine auf einer einzigen mRNA meist Untereinheiten eines multimären Proteins; damit Proteine in konstantem molarem Verhältnis Eukaryonten a) im Zellkern b) Transkription und Translation räumlich und zeitlich getrennt; am 5’-Ende Schutzkappe drauf aus primären Transkript müssen Introns herausgespliced werden 3’-Ende mit Poly-ASchwanz verlängert (mRNA) c) eine reife mRNA codiert immer nur für ein Protein; Proteinbestand fast immer aus mehreren Domänen; Untereinheiten( Domänen) fusionieren, [„exon-shuffling“] 13. Struktur (in Worten beschrieben reicht) und Rolle des Cap? Prokaryonten: drei Phsphate Eukaryonten: drei Phosphate + Guanosin Schutzkappe am 5’-Ende der mRNA damit es nicht sofort verdaut wird 14. Welche Sequenz-Blöcke muss ein typisches Intron besitzen? Welche funktionelle Rolle spielt der mittlere Block? Consensus-Sequenzen: Anfangssequenz, Lassosequenz, Endsequenz Lassosequenz: mit Adenosin-Baustein, 2’-OH des Lasso-Adenins attackiert die andere Spleißstelle , sodass das 3’-OH des Exons freikommt. 15. Wie kann bei der Prozessierung der mRNA zwischen Exons und Introns unterschieden werden? (vor allem, wenn die charakteristischen Flankierungssequenzen stärker von den Consensus-Sequenzen abweichen). Anfangssequenz, Exons haben sehr charakteristische Länge (~200BP), Introns unterschiedlich lang, Exons haben andere Sequenz-Charakteristika (reich an Serin und Argenin, SR-Proteine dekorieren) 16. Welche funktionelle Rolle spielen die RNA-Komponenten und welche die ProteinKomponenten der snRNP (small nuclear Ribo Nucleo Protein)? alle katalytischen Schritte werden von RNA-Molekülen geleistet, die Proteinkomponenten wirken nur peripher mit, viele der Proteine verbrauchen ATP, viele Umlagerungen energetisch voranzutreiben 17. Erklären Sie das Prinzip des Selbstspleißens. in Mitochondrien, Chloroplasten; Intron faltet sich so dass des 3’-Ende des linken Exons mit dem 5’Ende des rechten Exons zusammenkommt; dazu ist eine ganz definierte Intronsequenz nötig Bernhard Polzinger WS 2004/2005 Seite 2/4 Molekulare Biologie der Zelle Kapitel 6A: Transkription (RNA-Synthese) 18. Wie wird die Transkription von mRNA beim Eukaryonten beendet? AAUAA signalisiert das Ende der Transkription. Spaltungsfaktoren schneiden 10-30 BP neben AAUAAA den RNA-Transkript an einer CA-Sequenz 200AMP-Reste an 3’-OH angehängt (ohne Vorlagestrang) 19. Wie kann die Kernpore eine reife mRNA von einer unfertigen bzw. von herausgeschnittenen Introns unterscheiden? Cap bindet das CBP (cap-binding-protein) Exons noch mit SR-Proteinen besetzt Poly-A-Schwanz mit Poly-A-Bindeproteinen besetzt unklar welches der Proteine auf der reifen mRNA das Signal für den Export darstellt die snRNP assoziierten Introns können Kern nicht verlassen und werden abgebaut Proteinbestückung entscheidet also über Export oder Abbau 20. Was geschieht im Nucleolus? Chromosomen ragen in Nucleolus hinein Transkription von rRNA räumlich konzentriert rRNA wird hergestellt, chemisch modifiziert und geschnitten 21. Was geschieht mit dem Nucleolus während der Mitose? schrumpft und löst sich auf 22. Was geschieht mit einer korrekt gefalteten rRNA, was mit einer inkorrekt gefalteten tRNA? nur korrekt gefaltete RNA-Moleküle können von Enzymen erkannt und gespleißt werden (ohne Hilfe von RNA), Spleißmechanismus wirkt also als Qualitätskontrolle, ob die tRNA richtig gefaltet ist , inkorrekte können nicht geschnitten werden und nicht in Funktion treten, weil Enzym nur solche annimmt 23. Welchen Zweck hat die Modifikation der tRNA-Basen a) im Allgemeinen? b) im Anticodon? a) viele dieser Modifikationen helfen mit, um besonders stabile intramolekulare Basenpaarungen, sehr kompakte Faltungen der tRNA zu bewirken b) 61 verschiedene Codons der mRNA müssen durch wenige tRNA-Sorten interpretiert werden 24. Wie sehen die Tripletts einer Aminosäure aus, welche von 4 verschiedenen Tripletts codiert wird? 3 Basen eine Aminosäure Basentriplett der mRNA: Codon 3 Positionen, 4 Optionen 43 = 64 mögliche Basentripletts davon 3 Stop-Codons früher 2-Basencode mit weniger Aminosäuren immer die ersten beiden Basen sind konstant und nur die dritte kann variieren 25. Wie wird der Leseraster auf der mRNA festgelegt? Was passiert bei der Deletion von genau einer Base mitten in einem Protein-Gen? x … durch Start-Codon definiert (ist jenes AUG welches dem 5’-Ende der mRNA (Cap) am nächsten ist. x … Leseraster verschiebt sich um eine Base nachfolgende Sequenz bekommt eine völlig andere Bedeutung 26. Welche Struktur hat eine tRNA? Wo ist das Anticodon? Wo hängt die Aminosäure? x … Kleeblattform (da Seitenlappen eingeklappt) räumlich L-Form x … an der Schleife am Ende (also gegenüber der Aminosäure) sitzt das Anticodon x … am 3’-Ende 27. Was ist eine Aminoacyl-tRNA-Transferase? Wie viele verschiedene Arten dieses Transferase-Typen hat eine Zelle? ein Enzym, welches einen Aminoacylrest auf eine tRNA überträgt 20 für 20 Aminosäuren Bernhard Polzinger WS 2004/2005 Seite 3/4 Molekulare Biologie der Zelle Kapitel 6A: Transkription (RNA-Synthese) 28. Wie ist der biochemische Mechanismus der Kopplung einer Aminosäure an eine tRNA? (Es geht hier nicht um die Zuordnung der richtigen Aminosäure zur richtigen tRNA). Frei Aminosäure wird durch ein Enzym mit ATP zur Reaktion gebracht. Dabei wird ein ganzer AMP-Rest auf die Aminosäure angehängt. Bindung zwischen der C=O Gruppe der Aminosäure und dem Phosphatrest ist eine Anhydrid-Bindung, AMP ↔ Abgangsgruppe, wenn die AnhydridBindung von der 3’-OH-Gruppe der tRNA attackiert und Bildung von Ester zwischen Aminosäure ↔ tRNA 29. Bakterien haben 61 Codons auf der mRNA, aber nur 31 Anticodons in den 31 tRNASorten. Wie können die 31 Anticodons die 61 Codons eindeutig ablesen? Einige Anticodons können an der dritten Stelle des mRNA-Codons mehr als nur ein Codon erkennen (wobbling). 3.Base um 180°drehen. 30. Bakterien haben 31 verschiedene tRNA-Sorten aber nur 20 Aminosäuren. Nach welchem Prinzip wird eine Aminosäure auf all jene tRNA-Untertypen gehängt, welche für diese eine Aminosäure codieren? Für das Kopplungsenzym sind diese Untertypen gleich, nur das Anticodon ist für diese Untertypen verschieden. 31. Was passiert, wenn eine falsche Aminosäure an eine bestimmte tRNA gehängt wurde? Aminosäure an die tRNA gekoppelt tRNA presst den Aminoazyl-Rest richtig fest in die Bindungstasche. Falsche Aminosäure sterischer Stress Enzym wechselt in Editiermodus schneidet ab Bernhard Polzinger WS 2004/2005 Seite 4/4