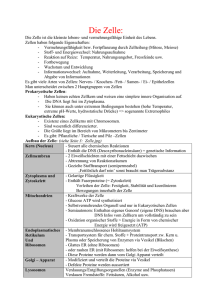
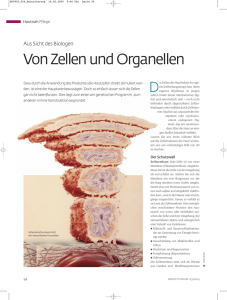
Cell Biology
Werbung

Cell Biology Kovarik, Martens (LvNr. 300037) SS12 Skriptum und Fragenausarbeitung GNU General Public License Kovarik – Foliensatz 1 Zellteilung: Phasen der Interphase: G1: Nach der Zellteilung wächst die Zelle und produziert Material für DNA-Replikation S-Phase: in der S-Phase findet DNA-Replikation statt G2: in der G2-Phase überprüft die Zelle die Vollständigkeit und Richtigkeit der DNA-Replikation. G2 ist auch die Phase des weiteren Zellwachstums. G0: falls die Zelle in der G1-Phase keine Signale für Zellteilung bekommt, geht sie in die Ruhephase (die sog. G0-Phase) über. Erst bei promitotischen Signalen kehrt sie in den Zellzyklus zurück. Mitose: Prophase Prometaphase Metaphase Anaphase Telophase Cytokinese Centrosom, Centriolen Centrosom: das “microtubule organizing center” in den meisten Zellen ist das Centrosom. Dem Centrosom entspringen Mikrotubuli der mitotischen Spindel. Das Centrosom ist für die Entstehung der bipolaren Spindel notwendig, d.h. es muss vor der Mitose repliziert werden. Mit dem Centrosom sind sog. Centriolen assoziiert, die für das richtige Positionieren und die Duplikation des Centrosoms notwendig sind. mitotische Spindel Die mitotische Spindel besteht aus Centrosomen Kinetochoren (Ort des Kontaktes zwischen Mikrotubuli und Chromosom) Kinetochor-Mikrotubuli polaren Mikrotubuli astralen Mikrotubuli Cell Biology SS12 (Kovarik, Martens) Seite 1 von 65 Christian Husch Centrosom, Kinetochor Centromer: Region des Chromosoms, die an Kinetochor bindet Kinetochor: großer Proteinkomplex, der die Centromer-Region das Chromosoms mit Kinetochor-Mikrotubuli verbindet mitotische Spindel in der Metaphase Die Länge der Mikrotubuli in der Metaphase bleibt gleich: die Polymerisation am Plus-Ende ist gleich schnell wie der Abbau am Minus-Ende. mitotische Spindel in der Metaphase und Anaphase Kinetochor-Mikrotubuli werden in der Anaphase kürzer (durch überwiegende Depolymerisation an beiden Enden) mitotische Spindel in der Anaphase Trennung der Schwesterchromatide: Anaphase A: Kinetochor-Mikrotubuli werden kürzer und bewegen die Chromosomen zu den Spindelpolen. Anaphase B: polare Mikrotubuli werden länger weil anti-parallele Mikrotubuli aneinander vorbei gleiten und schieben die Centrosomen voneinander weg. Die Spindel verlängert sich. Mechanismus der durch das Kinetochor erzeugten pol-gerichteten Kraft: Schrittweise Entwicklung der Kinetochor-Mikrotubuli-Interaktionen in eukaryotischen Zellen. Die Abbildung zeigt KinetochorMikrotubuli (KT-MT) Interaktionen während Prometaphase (Schritte 1-3), Metaphase (Schritt 4) und Anaphase (Schritt 5). Schritt 1: Das Kinetochor (KT) interagiert zunächst mit der seitlichen Oberfläche eines einzigen Mirkotubuli (MT) (seitlicher Ansatz), welches sich von einem Spindel-Pol (Spindel-Pol-MT) (rechts) erstreckt. Dieses Verfahren wird oft durch die Wechselwirkung zwischen einem KT-abgeleiteten MT und einer Spindel-Pol-MT (links) erleichtert. Es ist noch nicht bekannt, ob beide oder ob nur eines der Schwester-KTs sich an einem MT während der seitlichen Anlagerung anhaftet. Schritt 2: Sobald an einem Spindel-Pol-MT gebunden wurde, wird das KT entlang der MT-Mantelfläche in Richtung eines Spindel-Pols transportiert (gleiten, links). Anschließend wird, zumindest in Hefe, das KT am Ende eines Spindel-Pol-MT (End-Befestigung) angebunden und transportiert, wenn die MT schrumpft (End-anziehen, rechts). Cell Biology SS12 (Kovarik, Martens) Seite 2 von 65 Christian Husch Schritt 3: Nach dem KT-Transport in Richtung eines Spindel-Pols, könnten beide Schwester KTs an MTs binden. Wenn beide Schwester-KTs an am gleichen Spindel-Pol eines MT binden, muss die KT-MT-Bindung umgeformt werden, bis die richtige Bi-Orientierung etabliert ist. Schritt 4: Der Umbau der KT-MT-Anlage stoppt, sobald Spannung über Schwester KTs nach Errichtung der BiOrientierung erzeugt wurde. Die Zahl der MTs, deren Plus-Enden mit einer einzigen KT befestigt sind, steigt, je mehr Spannung auf Metazoen-KTs aufgebracht wird, während. nur ein einziges MT an jedes Schwester-KT in der Bäckerhefe bindet. Schritt 5: Nachdem alle Schwester-KTs an der Spindel bi-orientiert sind, wird die Kohäsion zwischen den Schwesterchromatiden entfernt, wodurch Schwesternchromatid-Segregation zu entgegengesetzten Spindelpolen während der Anaphase beginnt. In der Bäckerhefe geschieht die Trennung der Schwesterchromatiden entlang eines Chromosomenarms vom dem Zentromer zum Telomer. Die End-on KT-MT-Anlage wird solange beibehalten, bis die MTs depolymerisieren und Schwester-KTs zu den gegenüberliegenden Polen, in der Anaphase A, gefolgt vom Pol-zu-Pol-Abstand , welcher in der Anaphase B vergrößert wird. Wie Aurora-Kinase B/Ipl1 die Fehlerkorrektur der Kinetochor-Mikrotubuli-Befestigung fördert: Die Aurora-Kinase B/Ipl1 erleichtert SchwesterKT Bi-Orientierung durch Förderung des Umsatzes von KT-MT-Anlage in einer Zug-abhängigen Weise; dieser Prozess, so wird angenommen, findet wie folgt statt: Während ?Syntelic?-Anlage keine Spannung über Schwester KTs erzeugt (links), phosphoryliert die Kinase Aurora B/Ipl1 KT-Komponenten wie Ndc80 und DAM1, wodurch der Umsatz der KTMT-Bindung gefördert wird. Wenn biOrientierung als Ergebnis dieses Umsatzes (rechts) hergestellt ist, wird Spannung auf Schwester-KTs ausgeübt, wodurch Delokalisierung der KT-Substrate von Aurora B/Ipl1hervorgerufen wird, die zu ihrer de-Phosphorylierung führt. Das wiederum macht die KT-MT-Befestigung stabiler und stoppt den Umsatz dieser Bindung. Somit wird SchwesterKT bi-Orientierung stabil aufrechterhalten und die Fehlerkorrektur für die KT-MT-Anlage abgeschlossen. Cytokinese bei höheren Eukaryonten Während der Cytokinese entsteht in der Äquatorebene der Spindel ein Aktinring (sog. “contractile ring”), der durch die Aktivität von Myosin enger wird. Die Kontraktion wird fortgesetzt bis die vollständige Trennung der Zellmembran erreicht wird. Zellteilungskontrolle Die Zellteilung ist das Schlüsselereignis des Lebens und sie muss daher absolut präzise ablaufen. In der Zelle gibt es viele Mechanismen, die die Zellteilung steuren und den korrekten Ablauf überprüfen. Zum Zeitpunkt “start” entscheidet sich die Zelle (irreversibel) in den Zyklus zu gehen. Die einzelnen Phasen des Zellzyklus werden an bestimmnten Stellen (“checkpoints”) überprüft. Cell Biology SS12 (Kovarik, Martens) Seite 3 von 65 Christian Husch Die Ereignisse der eukaryotischen Zellteilung sind unter dem Mikroskop erkennbar. Die gut sichtbaren Vorgänge der Kernteilung (Mitose) und Zellteilung (Cytokinese), die sogenannte M-Phase, nehmen in der Regel nur einen kleinen Bruchteil des Zellzyklus ein. Die andere, viel längere Phase, wird Interphase genannt. Die fünf Stadien der Mitose sind in der Grafik veranschaulicht. Eine abrupte Änderung des biochemischen Zustands der Zelle tritt beim Übergang von Metaphase zu Anaphase auf. Eine Zelle kann in der Metaphase verweilen vor diesem Übergang. Aber ist der Punkt einmal überschritten worden, zieht die Zelle es bis zum Ende der Mitose und Zellteilung in der Interphase durch. Die DNA-Replikation tritt nur in der Interphase auf!! Der Teil der Interphase, wo DNA repliziert wird, wird als S-Phase bezeichnet (nicht gezeigt). Die Phasen des Zellzyklus: Die Zelle wächst kontinuierlich in Interphase, die aus drei Phasen besteht: DNA-Replikation geschieht in der S-Phase, G1 ist die Lücke zwischen der M-Phase und der S-Phase, während G2 der Spalt zwischen der S-Phase und M-Phase ist. In der M-Phase, teilen sich der Zellkern und dann das Zytoplasma auf. In proliferierenden Säugetierzellen kann die Interphase mehr als 90% des Zellzyklus ausmachen (zB Interphase 23 h, M-Phase 1 h). Das wichtigste Merkmal aller lebenden Organismen: Vermehrung der Zellen durch Duplikation des genetischen Materials und Zellteilung Zellzyklus: wiederholte Runden von Duplikation und Zellteilung Einzeller (z.B. Bakterien und Hefe): jede Zellteilung führt zur Entstehung eines neuen Organismus Mehrzeller: viele komplexe Sequenzen von Zellteilungen sind notwendig für die Entstehung eines neuen Individuums Zellzyklus unterliegt vielfältigen Kontrollmechanismen = Zellzykluskontrolle Warum Zellzyklus-Kontrolle? DNA-Replikation muß beendet sein, bevor die Chromosomen segregieren. Chromosomensegregation muß beendet sein, bevor sich die Zelle teilt. Konsequenzen einer defekten Zellzyklus-Kontrolle: Krebs, degenerative Erkrankungen, defektes Immunsystem Defekt der Zellzyklus-Kontrolle; Beispiel anhand einer Mutation des Myostatin-Gens: Eine Myostatin-Mutation hat Auswirkungen auf die Muskelmasse. Die Mutation führt zu einer dramatischen Zunahme des Muskelgewebes (zB Belgian Blue Stier). Der Stier wurde von Viehzüchtern produziert und es wurde erst vor kurzem festgestellt, dass er eine Mutation im Myostatin-Gen hat. Mäuse mit dem gleichen Gendefekt haben auch bemerkenswert große Muskeln Wie findet man Faktoren, die den Zell-Zyklus kontrollieren? 1) Genetischer Ansatz Klar unterscheidbare Zellzyklus-Phasen einfache genetische Manipulation(Einleiten von Mutationen, um Funktionen von Genen zu verändern) eine einfache Mutation erzeugt einen Phenoty (haploider Genotyp notwendig) Nützliche Systeme: Saccharomyces cerevisiae (Bäckerhefe) Schizosaccharomyces pombe (gespaltete Hefe) 2) Biochemischer Ansatz: In vitro-system muß verfügbar sein Nützlich: Xenopus 3) Biochemie und Zellbiologie: Große Zellen (für biologische Studien) in großer Anzahl (Für biochemische Studien) müssen verfügbar sein. Cell Biology SS12 (Kovarik, Martens) Seite 4 von 65 Christian Husch Nützliche Systeme:: Säugetierzellen-Kultur; benutzt werden immortalisierte/transformierte Zellen Beispiel anhand der Bäckerhefe, welche in einer gewissen Zellzyklus-Phase verweilte: Die Morphologie der knospenden Zellen der Bäckerhefe wurde durch eine CDC-Mutation verhindert. In einer normalen Population proliferierender Hefezellen, variieren die Knospen in der Größe entsprechend des Zellzyklus-Stadiums. In Cdc15-Mutanten die bei eingeschränkter Temperatur gewachsen sind, beendeten zwar die Anaphase, aber konnten nicht den Austritt aus der Mitose und Zytokinese abschließen. Infolgedessen verblieben sie alle mit großen Knospen, die charakteristisch für die späte M-Phase sind. Kovarik – Foliensatz 2: Lebenszyklus von S. cerevisiae: Drei Phasen des Lebenszyklus von Saccharomyces cerevisiae, die polarisiertes Zellwachstum aufweisen: Die Zellen, die in einem reichen Medium gewachsen sind, sind rund oder oval und haben definierte Muster. Wenn sie einem LowNährmedium ausgesetzt werden, bilden sich längliche Zellen und knospen am distalen Ende, um Pseudohyphen zu bilden. Haploide Zellen, die Zellen des anderen Zelltyps ausgesetzt waren, verbleiben in der G1Phase und haben somit einen Rückstand gegenüber den anderen Zelltypen. Diploide Bildung kann durch axiale Muster gefördert werden. Mutter-Zellen wechseln ihren Paarungstyp in der späten G1-Phase. Knospung an den proximalen Seiten positioniert Zellen in unmittelbarer Nähe zueinander, um die Bildung der diploiden Form zu erleichtern. Cell Biology SS12 (Kovarik, Martens) Seite 5 von 65 Christian Husch Polarisierte mRNA, Lokalisiert in der Knospenspitze in Hefe. (A) Der molekulare Mechanismus der ASH1 mRNA Lokalisation wird durch Genetik und Biochemie bestimmt. (B) Fluoreszenz-in situ-Hybridisierung (FISH) wurde zur Lokalisierung der ASH1 mRNA (rot) in dieser teilenden Hefezelle verwendet. Die mRNA ist auf die obere Spitze der Tochterzelle (hier noch eine große Knospe) beschränkt. ASH1 Protein, das von der lokalisierten mRNA transkribiert wird, ist auch auf die Tochterzelle beschränkt. Die Mutterzelle transkribiert eine mRNA von ash1. Das Protein ash1 fungiert als Repressor des Maitingtyp-Switching. Während des Buddings wird die mRNA in den Bud transportiert. Dies geschieht auf Grund einer Sequenz am 3’-Ende. Diese Sequenz wird von She2 erkannt und anschließend bindet She3 und MyosinV. Letzteres ist ein Motorprotein. Ash1 ist somit nur noch in der Tochterzelle vorhanden und inhibiert die HO-Endonuclease. Diese ist nun in der Mutter aktiv und bewirkt, dass der MAT-Lokus herausgeschnitten wird und durch den anderen ersetzt wird. Beide MAT Typen (a und α) sind als unabhängig Kopie an anderer Stelle erhalten. Daher kann nur die Mutterzelle den Matingtyp ändern. Schlüsselereignisse sind wichtig für die Zellschicksal-Bestimmung in Hefe. In postanaphase-Zellen, ASH1 mRNA, welche einen Repressor für Paarungstyp-Wechsel kodiert, ist an die Spitze der Knospe lokalisiert. Das Protein produziert bevorzugt Verbindungen mit dem Tochterkern und unterdrückt dadurch HO-Transkription. HO-Endonuklease ist entscheidend für Paarungstyp-Wechsel in Hefe. Der Signalweg in der Hefe-Paarungs-FaktorAntwort.: Der extrazelluläre Paarungs-Faktor bindet an einen G-Protein-gekoppelten Rezeptor in der Plasmamembran. Die Aktivierung des Rezeptors löst Dissoziation der GTP-gebundenen Ga-Untereinheit aus einem heterotrimeren G-Protein aus. Dies wiederum aktiviert das Rho-Familie GTP-bindende Cell Biology SS12 (Kovarik, Martens) Seite 6 von 65 Christian Husch Protein, Cdc42, das Hefe-Homolog der Säuger-GTPase, deren Aktivierung zur Bildung von Filopodien führt. Wie in Säugerzellen, aktiviert Cdc42 ein Protein (Bee1, das Homologe von WASp), das den ARP-Komplex aktiviert, was zur lokalen Aktin-Kernbildung an der Seite der Mating-Faktor-Bindung führt. Die lokale Aktin-Kernbildung und das Filamentwachstum führt zu polarisiertem Wachstum und Erlangung einer Shmoo-Form. Darüber hinaus löst die Rezeptoraktivierung andere Antworten durch eine MAP-Kinase-Kaskade aus zur Vorbereitung der Paarung der haploiden Zelle. Das Verhalten eines temperaturempfindlichen cdc-Mutanten: Bei der permissiven (niedrigen) Temperatur, teilen sich die Zellen normal, und werden in allen Phasen des Zyklus vorgefunden. Beim Erwärmen auf die restriktive (hohe) Temperatur, bei der das mutierte Gen-Produkt abnormal funktioniert, setzen die mutierten Zellen den Zellzyklus fort, bis sie an einen spezifischen Punkt ankommen, den sie nicht abschließen können. (zB Beginn der S-Phase). Da die CDC-Mutanten noch weiter wachsen, werden sie ungewöhnlich groß. Im Gegensatz, bei nicht cdc-Mutanten, wenn Defizite bestehen in einem Prozess, der notwendig ist für den weiteren Zellzyklus für Biosynthese und Wachstum (z. B. ATP-Produktion), kann der Zellzyklus jederzeit an jeder Stelle angehalten werden - je nachdem, wann die biochemischen Reserven erschöpft sind. Komponenten der Zellzyklus-Maschinerie: Cdk and cyclin CdK – cyclin dependent kinase cdks sind nur aktiv, wenn Cycline an ihnen gebunden sind und sie somit aktivieren. Durch die Bindung kommt es zur Konformationsänderung und es wird eine ATP-Bindestelle frei. Ohne CyclinBindung befindet sich cdk im inaktiven Zustand und ist durch eine Region des Proteins, der T-Schleife, blockiert. Durch Bindung des Cyclin wird die T-Schlaufe aus der aktiven Stelle verdrängt und es kommt zu einer partiellen Aktivierung von Cdk2. Die Phosphorylierung eines Threonin-Restes an der T-Schleife des Cdk2 durch CAK (cdk-aktivierende Kinase) führt zur Aktivierung des Enzyms durch Wechsel der Form der T-Schleife, wodurch sich die Fähigkeit des Enzyms verbessert, an die Proteinsubstrate zu binden. Dadurch ist der cdk-cyclin-Komplex vollständig aktiv. Inhibierung von Cdk: Die Regulation der Cdk-Aktivität erfolgt durch inhibitorische Phosphorylierung. Der aktive Cyclin-CDK-Komplex wird deaktiviert, wenn die Kinase Wee1 zwei eng aneinanderliegende Stellen über dem aktiven Zentrum phosphoryliert. Die Entfernung dieser Phosphate durch die Phosphatase Cdc25 führt zu einer Aktivierung des Cyclin-Cdk-Komplexes. Der Einfachheit halber ist nur ein inhibitorisches Phosphat gezeigt. Das aktivierende Phosphat wird durch CAK hinzugefügt. Checkpoints kontrollieren den Zellzyklus-Prozess: Die essentiellen Prozesse der Zelle (DNA-Replikation, Mitose, Zytokinese) werden durch ein Zellzyklus-Kontrollsystem überwacht. Dieser fungiert als Controller, der wichtige Prozesse auslöst, sobald spezifische Phasen/Ereignisse erreicht werden. Check-Point beim Zell-Zyklus: Ein Checkpoint ist ein jener Punkt im Zellzyklus, bei dem der Zyklus solange angehalten wird, bis alles nötigen Vorgange korrekt ausgeführt worden sind. Beispiel: Spindel-Checkpoint In der Metaphase ordnen sich die Chromosomen in der Äquatorialebene an. Der Spindelapparat setzt an den Chromosomen an. Normalerweise binden die Kinetorchor-Mikrotubuli an die Kinetochore. Findet diese Bindung nicht statt, so kann statt dessen Mad2 an die Kinetochore binden. Infolge dessen findet eine Konformationsänderung statt und Mad2 bindet nun cdc20. Damit kann cdc20 den APC-Komplex nicht aktivieren. Somit kann Securin nicht von der Separase getrennt werden und die Schwersternchromatiden werden nicht getrennt (Kohesin bleibt erhalten) Cell Biology SS12 (Kovarik, Martens) Seite 7 von 65 Christian Husch Kovarik – Foliensatz 3: DNA-damage checkpoint Wie DNA-Schäden den Zellzyklus in der G1-Phase anhalten: Wenn DNA beschädigt ist, werden Proteinkinasen, die p53 phosphorylieren, aktiviert. Mdm2 bindet normal an p53 und fördert seine Ubiquitinierung und Zerstörung in Proteasomen. Die Phosphorylierung von p53 blockiert die Bindung an Mdm2, das Resultat ist eine Akkumulation von p53 in großer Zahl und stimuliert die Transkription von Genen, welche das CKI-Protein p21 codieren. Das p21 bindet und inaktiviert G1/S-Cdk und S-CDK-Komplexe, und hält die Zelle in der G1-Phase. In einigen Fällen induziert die DNA-Schädigung auch entweder die Phosphorylierung von Mdm2 oder eine Abnahme der Mdm2 Produktion, die einen Anstieg der p53 bewirkt. Das Ubiquitin-Proteasom-System: (a) Ubiquitin wird durch ein Ubiquitin-aktivierendes Enzym (E1) aktiviert und auf das Substrat-Proteine übertragen durch Ubiquitinkonjugierenden Enzyme (E2) und Ubiquitin-Ligasen (E3), was entweder zu (b) Befestigung eines einzigen Ubiquitin-Moleküls (MonoUbiquitinierung), oder Anbringung von mehreren Ubiquitin-Einheiten bis zu mehreren Substrat Lysin-Reste auf dem gleichen Protein (MultiUbiquitinierung) oder Synthese von Ubiquitin-Ketten (Poly-Ubiquitinierung) führt. (c) Viele Poly-Ubiquitin markierten Proteine werden anschließend abgebaut durch das 26S Proteasom, das aus dem katalytischen 20S-Komplex und regulatorischen 19S-Partikeln besteht. Abgebaute Substrate werden entweder zum Proteasom durch lösliche UbiquitinRezeptoren geliefert oder erkannt durch die intrinsische Ubiquitin-bindende Aktivität des 19S-Partikels. Beim 19S Proteasom ist die Ubiquitin-Kette zerlegt, und das Substrat wird entfaltet, bevor es in den Hohlraum des 20S-Untereinheit, wo Proteolyse erfolgt, eindringen kann. Schließlich verlassen proteolytische Fragmente das Proteasom in bisher schlecht verstandener Art und Weise. (d) Ubiquitinierung kann daher direkt ProteinFunktion regulieren, in einer Proteolyse-unabhängigen Art und Weise, über Mono-, Multi-oder PolyUbiquitinierte Proteine. Die Steuerung der Proteolyse durch SCF und APC während des Zellzyklus. (A) Die Phosphorylierung eines Zielproteins, wie die CKI gezeigt, ermöglicht dem Protein, von SCF erkannt zu werden, welches konstitutiv aktiv ist. Mit Hilfe von zwei zusätzlichen Proteinen, E1 und E2, dient SCF als Ubiquitin-Ligase, die mehrere UbiquitinMoleküle auf das CKI-Protein überträgt. Das ubiquitinierte CKI-Protein wird dann sofort erkannt und abgebaut in einem Proteasom. (B) M-Cyclin Ubiquitinierung wird von APC durchgeführt, welches in der späten Phase der Mitose aktiviert wird durch die Zugabe einer aktivierenden Untereinheit an den Komplex. Sowohl SCF und APC enthalten Bindungsstellen, die spezifische Aminosäure-Sequenzen des Zielproteins zu erkennen. Zellzyklus-Regulation – Checkpoints in S. cerevisiae: Zwei wichtige Kontrollpunkte: G1 Checkpoint: Wenn DNA-Schäden festgestellt werden, ist Paarungspheromon vorhanden, oder die Zelle hat Cell Biology SS12 (Kovarik, Martens) Seite 8 von 65 Christian Husch die kritische Größe nicht erreicht. Die Zelle verbleibt in G1 und kann den Start der Transition, welche eine neue Runde der DNA-Synthese und Mitose durchläuft, nicht beginnen. Spindel Checkpoint: Wenn DNA-Schäden festgestellt werden, ist die DNA nicht vollständig vervielfältigt oder Chromosomen sind nicht auf der Metaphaseplatte ausgerichtet, verbleibt die Zelle in der Metaphase und ist nicht in der Lage, den Übergang zu beenden, wo Schwesterchromatiden getrennt und auf die Zellen aufgeteilt werden. Diese Kontrollpunkte werden durch die Cdk / Cyclin-Komplexe, eine Familie von Proteinkinasen erzwungen. Es gibt verschiedene Mechanismen zur Regulierung Cdc28-Aktivität in der Zelle, nämlich: durch die Synthese von Cycline durch verschiedene Transkriptionsfaktoren. durch den Abbau der Cycline (gefördert von APC und SCF). durch die Verbindung mit stöchiometrischen CDK-Inhibitoren (Sic1 und Cdc6 und Far1). durch Phosphorylierung und Dephosphorylierung von Cdc28 durch SWE1 und Mih1. Veranschaulichung der allgemeinen Aspekte der CDK Regulierung: CDK-Aktivierung erfordert Cyclin (CYC) – Expression und -Verbindung. Cyclin/CDKKomplexe werden inaktiv gehalten durch die Verbindung mit CDK-inhibitorischer Proteine (CKIs) und inhibitorischer Phosphorylierung durch Wee1/Myt1-Kinasen (schwarze Kreise). Die Aktivierung erfordert Ubiquitin-abhängige Proteolyse des CKI, Phosphorylierung der CDK durch eine CDK-aktivierende Kinase (CAK, roter Kreis), und Entfernen der inhibitorischen Phosphate durch eine Cdc25-Phosphatase. Cyclin Zerstörung führt zur Inaktivierung. Ubiquitin-abhängigen Proteolyse von Regulatoren des Zellzyklus in der späten G1und S-Phase beinhaltet Cullin-basierte E3Ligasen wie SCF, während in der M- und der frühen G1-Phase der Anaphase-Promoting Complex (APC) aktiv ist. Das Ausrufezeichen bezeichnet den aktive Kinase-Komplex, der große Pfeil zeigt die Zeit. Zellzyklus-Regulation – allgemeine Aspekte: Die Aktivierung von M-Cdk. Cdk1 assoziiert mit M-Cyclin wenn das Niveau des M-Cyclin allmählich steigt. Der resultierende M-Cdk-Komplex wird auf einer aktivierenden Seite phosphoryliert durch die Cdk-aktivierende Kinase (CAK) und einem Paar inhibitorischer Seiten der Wee1-Kinase (nur einer ist der Einfachheit halber gezeigt). Der resultierende inaktive M-CdkKomplex wird dann am Ende des G2 durch die Phosphatase Cdc25 aktiviert. Cdc25 wird zum Teil durch Polo-Kinase, die der Einfachheit halber nicht gezeigt wird, stimuliert. Cdc25 wird weiterhin durch aktive M-Cdk stimuliert, was zu einer positiven Resonanz führt. Diese Rückkopplung wird durch die Fähigkeit von M-Cdk, WeeI zu inhibieren, verbessert. Kontrolle der G1-Progression in Säugetier-Zellen – E2F und Rb: Mechanismen, der die S-Phase Initiation in Säugetier-Zellen kontrolliert. G1-CDK-Aktivität (Cyclin D-cdk4) initiiert Rb-Phosphorylierung. Dies inaktiviert Rb, befreit E2F, um die Transkription der S-Phasen-Gene zu aktivieren, einschließlich der Gene für G1/S-cyclin (Cyclin E) und S-Cyclin (Cyclin A). Die resultierenden Aussehen G1/S-Cdk und S-Cdk-Aktivitäten und zusätzliche Rb-Phosphorylierung zur Bildung einer positiven Rückkopplungsschleife. Cell Biology SS12 (Kovarik, Martens) Seite 9 von 65 Christian Husch E2F wirkt zurück, um die Transkription seines eigenen Gens zu stimulieren, bildet eine weitere positive Feedback-Schleife. Kontrolle des Zellzyklus durch Wachstum/Zellgröße: Die Zellgröße wird durch Kontrolle des Zellzyklus in Hefen gesteuert. Diese Grafiken zeigen die Beziehung zwischen Wachstumsgeschwindigkeit, Zellgröße und Zellzyklus-Zeit. (A) Wenn die Zellteilung weiter mit unveränderter Rate, bei Fehlen von Nährstoffen und vermindertem Wachstum, stattfindet, würden die Tochterzellen bei jeder Teilung immer kleiner werden. (B) Hefezellen reagieren auf einige Formen von Nährstoffmangel durch Verlangsamung der Prozessgeschwindigkeit durch den Zellzyklus, so dass die Zellen mehr Zeit zum Wachsen haben. Als Ergebnis bleibt die Zellgröße unverändert oder wird etwas verringert. (Eine Zeiteinheit ist die Zykluszeit, wenn Nährstoffe im Überschuss vorhanden sind.) Kontrolle des Zellzyklus durch Wachstum/Zellgröße – ein Model für die Cln3-Funktion: Ein hypothetisches Modell, wie Bäckerhefe Zellen bezüglich Zellwachstum und Zellzyklus-Progression koordinieren könnte. Die Zelle enthält eine feste Anzahl von Proteinen (rot), die an DNA binden und Cln3-Moleküle (grün) binden und inhibieren. Da die Zelle wächst, steigt die Gesamtzahl der Cln3-Moleküle parallel zum gesamten Zellprotein. Wenn die Zelle klein ist (links), wird das gesamte Cln3 durch den Überschuss an Cln3-bindendes Protein inaktiviert. Da die Zelle jedoch wächst, erreicht die Größe einen Schwellenwert, bei der die Anzahl der Moleküle Cln3 gleich der Anzahl der Cln3-bindende Proteine (Mitte) ist. Wenn die Zelle diese Größe überschreitet, können freie Cln3 an Cdk binden, und so den nächsten Zellzyklus (rechts) auslösen. Kontrolle des Zellzyklus durch DNA-Schäden – p53-Protein (DNA-Schaden-Kontrollpunkt): Wie DNA-Schäden den Zellzyklus in der G1-Phase verbleiben lassen. Wenn DNA beschädigt ist, werden Proteinkinasen, welche p53 aktivieren, phosphoryliert. Mdm2 bindet in der Regel an p53 und fördert seine Ubiquitinierung und Zerstörung in Proteasomen. Die Phosphorylierung von p53 blockiert die Bindung dessen Bindung an Mdm2, als Ergebnis akkumuliert p53 in hohen Konzentrationen und stimuliert die Transkription des Gens, das CKI-Protein p21 codiert. Das p21 bindet und inaktiviert G1/S-Cdk und S-CDK-Komplexe und hält die Zelle in G1. In einigen Fällen induziert DNA-Schädigung auch entweder die Phosphorylierung von Mdm2, oder eine Abnahme der Mdm2 Produktion, die einen Anstieg des p53 bewirkt (nicht gezeigt). Cell Biology SS12 (Kovarik, Martens) p53 ist eines der stärksten Tumorsuppressor-Gene p53 mutiert/inaktiviert/fehlt in mehr als 50% der menschlichen Krebserkrankungen Zellen ohne funktionelles p53 akkumulieren Mutationen oft schneller Seite 10 von 65 Christian Husch Kovarik – Foliensatz 4: Zellzyklus: Regulation der Schwesternchromatiden-Separation (Spindel-Checkpoint): Die Steuerung der SchwesterchromatidenTrennung vom APC. Die Aktivierung von APC durch Cdc20 führt zur Ubiquitinierung und Zerstörung des Securin, das normalerweise Separase in einem inaktiven Zustand hält. Die Zerstörung des Securin erlaubt Separase, eine Untereinheit des Kohäsin-Komplexes zu spalten, welcher die Schwesterchromatiden zusammenhält. Die Zugkräfte der mitotischen Spindel ziehen dann die Schwesterchromatiden auseinander. In knospenden Hefen, wird die Kohäsinspaltung durch Separase durch Phosphorylierung des Kohäsin-Komplexes nahe der Spaltstelle ermöglicht, kurz bevor die Anaphase beginnt. Die Phosphorylierung wird durch Polo-Kinase vermittelt und bietet eine zusätzliche Kontrolle über den Zeitpunkt des Metaphase-Anaphase-Übergangs. Zelltod: Apoptose = programmierter Zelltod Nekrose = meist zufälliger Zelltod Warum programmierten Zelltod? eine Notwendigkeit für sauberes Entfernen von abgestorbenen Zellen (bei gesunden Erwachsenen Millionen von Zellen sterben jede Stunde) Notwendig für die Entfernung von potentiell gefährlichen Zellen (zB autoreaktiven T-Zellen oder Krebszellen) Notwendig für die Entfernung von Zellen, die nicht mehr benötigt werden (z. B. während der Entwicklung oder nach erfolgreicher Immunreaktion) ein Bedarf für ausbalancierte Proliferation (zB um ursprüngliche Größe der Leber nach einer Verletzungs-induzierten Proliferation aufrecht zu erhalten) Beispiele: Zellen in den Fingerzwischenräumen, Schwanz bei der Kaulquappe, mehr Nerven als Zielzellen vorhanden, ….. Cell Biology SS12 (Kovarik, Martens) Seite 11 von 65 Christian Husch Charakteristisch für Apoptose: Zellschrumpfung, Kondensation von Organellen Chromatin-Kondensation, Fragmentierung der DNA Zelle "Blasenbildung", die Integrität der Zellmembran ist erhalten, aber Phosphatidylserin transloziert von der Innenseite der Zellmembran auf die äußere Oberfläche Zelle zerfällt in apoptotische Körper (kein Entweichen des Zellinhalts! keine Entzündung) apoptotischen Zellen/apoptotische Körper werden von Fresszellen (z. B. Makrophagen) durch Phagozytose (keine Entzündung) entfernt Molekulare Mechanismen der Apoptose: Die Caspase-Kaskade ist bei der Apoptose beteiligt (A) Jede Selbstmord-Protease liegt als inaktives Pro-Enzym (Procaspase), die in der Regel durch proteolytische Spaltung durch ein anderes Mitglied der Familie Caspase aktiviert wird, vor. Wie angedeutet, verbinden sich zwei der gespaltenen Fragmente, die dann die aktive Stelle der Caspase bilden. Das aktive Enzym ist gedacht als ein Tetramer von diesen zwei Einheiten (nicht gezeigt). (B) Jedes aktivierte CaspaseMolekül kann viele Procaspase-Moleküle abspalten, damit sie aktiviert werden, und diese können dann noch mehr Procaspase-Moleküle aktivieren. Auf diese Weise kann eine anfängliche Aktivierung einer kleinen Anzahl von Procaspase-Molekülen (Initiator-Caspasen) dazu führen, daß eine verstärkte Kettenreaktion (Kaskade) gestartet wird, um eine explosive Aktivierung einer großen Anzahl von Procaspase-Molekülen zu starten. Einige der aktivierten Caspasen (so genannte Effektor-Caspasen) spalten dann eine Reihe von wichtigen Proteinen in der Zelle, einschließlich spezifischer cytosolischen Proteine und nukleare Lamine, was zum kontrollierten Zelltod führt. Regulation von Caspase-Aktivierung durch die Bcl-2-Proteinfamilie und Freisetzung von Cytochrom c. In einer lebensfähigen Zelle, gibt es die proapoptotischen Bcl-2Familienmitglieder Bax, Bak und BH3 – nur Proteine werden durch antiapoptotischen Elemente, wie Bcl-2, antagonisiert. Als Reaktion auf einen apoptotischen Stimulus, werden nur BH3 Mitglieder durch transkriptionelle Hochregulation (Bax, Noxa, Puma), subzelluläre Relokalisation (Bim, BMF), Dephosphorylierung (Schlecht) oder Proteolyse (Bid) aktiviert. Aktivierte nurBH3-Proteine schützen antiapoptotische Bcl-2-Mitglieder vor inhibierenden proapoptotischen Mitgliedern. Darüber hinaus könnten sie direkt eine Konformationsänderung von Bax und Bak induzieren, die anschließend oligomerisieren und in der mitochondrialen Membran inserieren und dort Poren entweder selbst oder durch den Umgang mit dem permeability transition pore komplex bilden. Infolgedessen werden proapoptotischen Faktoren, wie Cytochrom c, aus der inneren mitochondrialen Membran in das Cytosol entlassen, das zur Bildung des Apoptosom und die anschließende Aktivierung der Caspase-Freigabe beiträgt. Cell Biology SS12 (Kovarik, Martens) Seite 12 von 65 Christian Husch Kovarik – Foliensatz 5: Detektion der Apoptose: Während der Apoptose, erzeugt die DNase-Aktivität nicht nur doppelsträngige, niedrig-molekulare DNAFragmente (Mono-und Oligonukleosome), sondern führt auch Strangbrüche ("nicks") in die hoch-molekularen DNA ein. Diese Prozesse können durch Markierung der freien 3'-OH-Enden mit terminaler Transferase (TdT), die markierten Nukleotide an allen 3'-OH-Enden bindet, (; TdT-vermittelte dUTP nick end labeling TUNEL-Reaktion) identifiziert werden. In lebenden Zellen wird Phosphatidylserin durch Aminophospholipidtranslokase (ein Mg++ ATP-abhängige Enzym) zum Inneren der Lipid-Doppelschicht transportiert. Zu Beginn der Apoptose wird Phosphatidylserin zur äußeren Membran transloziert und dient als ein Erkennungssignal für Phagozyten. Annexine sind allgegenwärtige homologe Proteine, welche Phospholipide binden. Da die Umverteilung von Phosphatidylserin von der inneren zur äußeren Membranoberfläche einen frühen Indikator für Apoptose darstellt, kann Annexin V und deren Konjugate zum Nachweis von Apoptose verwendet werden, da sie stark und spezifisch mit freiliegenden Phosphatidylserin interagieren. Regulation der Apoptose: extrinsischer Weg: FasR/ FasL In der Zellmembran sitzt der FasR mit einer extrazellularen und einer intrazellularen Domane. Bindet an die extrazellulare Domane der FasL, so kommt es intrazellular zur Trimerisierung des Rezeptors. Die Trimerisierung hat zur Folge, dass die Death Domaine binden kann und weitere Adaptorproteine rekrutiert, wie Cas-8. Diese schneidet (aktiviert) Cas-3, welche wiederum ICAD (inaktive Caspase Aktivierende DNase) aktiviert. CAD wandert schließlich in den Kern undfragmentiert die DNA. Intrinsischer Weg: (Foliensatz 4) Intrazelluläre Aktivierung. Mitochondrien setzen Cytochrom c frei. die bindet an Apaf-1 und bewirkt, dass die Aggregation des AdapterProtein Apaf-1. Apaf-1 bindet und aggregiert Procaspase-9-Moleküle, was zur Spaltung dieser Moleküle und zur Auslösung einer Caspase-Kaskade führt. Andere Proteine, die Apoptose bewirken, werden auch vom mitochondrialen Intermembranraum (zB Apoptose induzierenden Faktors (AIF)) freigegeben. TNF TNF – tumor nekrose faktor, produziert von T-Zellen TNFR ist ein Membranrezeptor, der TNF bindet. Dadurch kommt es zur Trimerisierung und zum Clustering der Death Domain. Somit kann das Adaptermolekul TRADD (TNFR associated death domain) binden. Dies rekrutiert wiederum Traf-2 und aktiviert den NFκB bzw. JNK Pathway. Bindet jedoch FADD statt TRADD, so wird die Procaspase-8 rekrutiert und die Apoptose eingeleitet. Apoptose im Immunsystem: Cytotoxische T-Cell-Assoziierte Apoptose: Cytotoxische T-Zellen induzieren Apoptose durch 2 Mechanismen: A) Zytotoxische Granula aus zytotoxischen T-Zellen oder natürliche Killerzellen-Zellen werden bei Kontakt mit potentiell gefährlichen Zellen (zB Virus-infizierte Zellen) freigesetzt. Das Granulat löst Apoptose in Zielzellen durch die Wirkung der granulierten Proteasen, den Granzymen, aus. Granzyme (eine Protease in zytotoxischen Granula) löst die Apoptose direkt durch Aktivierung von Caspasen oder indirekt über Freisetzung von Cytochrom c und die Aktivierung des Apoptosom aus. B)Fas / Fas-Liganden-induzierte Apoptose. Hier muss die T-Zelle die Zielzelle erkennen und an den Liganden binden (über die T-Zell-Rezeptor auf der T-Zelle und das Antigen zusammen mit den MHC-Molekülen auf den Zielzellen). Cell Biology SS12 (Kovarik, Martens) Seite 13 von 65 Christian Husch Autophagie: Autophagie beinhaltet dynamische Membran-Umlagerung, um Teile des Zytoplasmas zu verschlingen, und es hat die Kapazität für die Speicherung von großen Strukturen, wie ganze Organellen. Hierbei werden Teile des Zytoplasmas in ein de novo-gebildetes Doppel-Membran-Vesikel (bezeichnet als Autophagosom) abgesondert. Anschließend verschmilzt das Autophagosom mit dem Lysosom/Vakuole und die inneren einzelnen Membranvesikel werden in das Lumen freigegeben. In jedem Fall wird die Membran des resultierenden autophagischen Körpers lysiert, um damit die phagozytierten Komponenten aufzuschlüsseln, und die resultierenden Makromoleküle zurück in das Cytosol, durch die Membran-Permeasen, zu transportieren zur Wiederverwendung. Grundlegende Autophagie: - Umatz von langlebigen Proteinen - Entfernung von beschädigten Organellen - Rolle in Verlängerung der Lebensspanne? Induzierte Autophagie: Zelluläre Antwort auf unterschiedlichste Streßverhältnisse (zB limitierter Nährstoff, Hitze, Oxidative Ereignisse) - Eliminierung von invasiven Pathogenen inkl. Viren, Parasiten und Bakterien Autophagie ist in einer Vielzahl von Erkrankungen, wie Krebs und neurodegenerative Erkrankungen wie Alzheimer, Parkinson und Chorea Huntington, involviert. Kovarik – Foliensatz 6: Typen des Programmierten Zelltods: Stimuli Apoptose: Bindes des death-Receptors DNA-Schäden Reaktive Sauerstoff-Arten Erscheinungsbild Pyknosis DNA-Fragmentierung Plasmamembran-Blasen Konsequenzen Formierung von apoptotischen Körpern Phagozytose Necroptose: TNFR1-Bindung + SMAC mimetics TNFR1 ligation + Caspase-Inhibierung Cytoplasma-Schwellung Freisetzung von intrazellulären Komponenten Inflammation Bakterielle Infektion Virale Infektion Stroke Cytoplasma-Schwellung DNA-Fragmentierung Porenformation Pyroptose: Organell-Schwellung Freisetzung von Cytokinen Inflammation Replikative Zellalterung: Replikative Seneszenz ist im Wesentlichen auf die Erosion der Telomere, Schutzbauten, die das Ende aller eukaryotischen Chromosomen bedecken. Telomere begrenzen SäugetierChromosomen und bestehen aus TTAGGG repetitiven Sequenzen, die in einem 3' einzelsträngige (ss) Überhang münden. Telomer-DNA wird komplexiert durch den sechs-Protein-Komplex shelterin Komplex, bestehend aus Telomer-Repeat-binding factor 1 (TRF1), TRF2, RAP1, TERF1interagierender nuclear factor 2 (TIN2), TPP1 und POT1. Das TPP1-POT1 Heterodimer reguliert Telomerase Zugriff auf das Telomer-Substrat. Der ss-Überhang kann in den doppelsträngigen Bereich des Telomer eindringen, um einen schützende Telomer (t)-Loop mit einem ss-displacement (D)-Loop bei der Invasion-Seite zu bilden. Säugetier-Telomere können auch vorübergehend mit einer Reihe von anderen Faktoren interagieren, von denen viele in der DNA-Schadens-Reaktion beteiligt sind. Telomerase-Expression ist gering oder fehlt in den meisten menschlichen somatischen Geweben. Telomerase ist in der frühen proliferativen Vorläufer-Keim-und Stammzellen robust. In Abwesenheit von Telomerase, wird jede Runde der DNA-Replikation durch die Verkürzung der Telomere begleitet. Ein totaler Lebensdauer-Verlust von rund 4.2 kb in durchschnittlichen Telomerlängen konnte in menschlichen Zellen beobachtet werden. Cell Biology SS12 (Kovarik, Martens) Seite 14 von 65 Christian Husch Anbetracht dessen, dass die menschliche Telomere nur 8-12 kb Länge oder weniger bei der Geburt haben, ist dieses Maß an Fluktuation über die natürliche Lebensspanne des Menschen bedeutsam, und könnte verantwortlich sein für bestimmte Aspekte des menschlich alternden Phänotypen. Replicative cell senescence (Replikative Zellalterung):. Replikative Seneszenz ist im Wesentlichen auf die Erosion der Telomere, Schutzbauten, die das Ende aller eukaryotischen Chromosomen bedecken. Telomere begrenzen Säugetier- Chromosomen und bestehen aus TTAGGG repetitiven Sequenzen, die in einem 3' einzelsträngige (ss) Überhang münden. Telomer-DNA wird komplexiert durch den sechs-Protein-Komplex shelterin Komplex, bestehend aus Telomer-Repeat-binding factor 1 (TRF1), TRF2, RAP1, TERF1- interagierender nuclear factor 2 (TIN2), TPP1 und POT1. Das TPP1-POT1 Heterodimer reguliert Telomerase Zugriff auf das Telomer-Substrat. Der ss-Überhang kann in den doppelsträngigen Bereich des Telomer eindringen, um einen schützende Telomer (t)-Loop mit einem ss-displacement (D)-Loop bei der Invasion-Seite zu bilden. SäugetierTelomere können auch vorübergehend mit einer Reihe von anderen Faktoren interagieren, von denen viele in der DNA-Schadens-Reaktion beteiligt sind. Contact inhibition: Der Verlust der Kontakthemmung in der Zellkultur: Die meisten normalen Zellen stoppen die Proliferation, wenn sie die Schale mit einer einzigen Schicht von Zellen bedeckt haben: Proliferation scheint abhängig vom Kontakt mit der Schale zu sein, und beim Kontakt mit anderen Zellen inhibiert zu werden. Krebszellen hingegen missachten in der Regel diese Beschränkungen und wachsen weiter, so dass sie stapelweise auf aufeinander zu liegen kommen. Cell Junctions - Okklusive Verbindungen versiegeln Zellen in einem Epithel in einer Weise, daß kleine Moleküle nicht von einer Seite der Membran auf die andere entweichen können. - Verankernde Verbindungen verbinden mechanisch Zellen (und ihr Cytoskelett) mit ihren Nachbarn oder mit der Extracellulär-Matrix. - Kommunizierende Verbindungen vermitteln die Passage von chemischen oder Elektrischen Signalen von einer Zelle zur anderen. Funktionelle Klassifikation von Zellverbindungen: Okklusive Verbindungen 1. tight junctions(nur in Wirbeltieren) 2. septate junctions (event. Wirbellose) Verankernde Verbindungen Actin filament-bindende Stelle 1. cell-cell junctions (haftende Bindung) 2. cell-matrix junctions (zentrale Bindung) Intermediate filament-bindende Stelle 1. cell-cell junctions (Desmosomen) 2. cell-matrix junctions (hemidesmosomes) Kommunizierende Verbindungen 1. gap junctions 2. Chemische Synapsen 3. Plasmodesmen (nur Pflanzen) Cell Biology SS12 (Kovarik, Martens) Seite 15 von 65 Christian Husch Kovarik – Foliensatz 7: Die Plasmodesmen in Pflanzen haben teilweise die gleiche Funktion wie die GAP-Junctions. Verankerungen: Verankernde Kreuzungen treten in zwei funktionell unterschiedlichen Formen auf: 1. Adhärenzverbindung und Desmosomen halten Zellen zusammen und werden durch haftende transmembrane Proteine, die zur Cadherin-Familie gehören, gebildet. 2. Fokalen Adhäsionen und Hemidesmosomen binden Zellen an die extrazelluläre Matrix und werden durch transmembrane Adhäsionsproteine der Integrin-Familie gebildet. Auf der intrazellulären Seite der Membran, dienen Adhärenzverbindungen und fokale Adhäsionen als Verbindungsstellen für die Aktin-Filamente, während Desmosomen und Hemidesmosomen als Verbindungsstellen für Intermediärfilamente dienen. Desmosomen: Desmosomen sind knopfähnliche Punkte für interzellulären Kontakt, um Zellen zusammen zu halten. In der Zelle, sie dienen als Ankerpunkte für die strickartigen Intermediärfilamente, bilden sie einen strukturellen Rahmen von großer Zugfestigkeit. Durch Desmosomen werden die intermediären Filamente der benachbarten Zellen in einem Netz, das in den vielen Zellen eines Gewebes vorkommt, verlinkt. Die besondere Art der intermediären Filamente an den Desmosomen hängt vom Zelltyp ab: zB: es sind Keratinfilamente in den meisten epithelialen Zellen, und Desmin-Filamente in Herzmuskelzellen. gap-Junktions: In Geweben mit elektrisch erregbaren Zellen dient die Kopplung über Gap Junctions einem offensichtlichen Zweck. Einige Nervenzellen sind elektrisch verbunden, so dass Aktionspotentiale sich rasch von Zelle zu Zelle, ohne Verzögerung, die bei chemischen Synapsen auftritt, ausbreiten können. Dies ist vorteilhaft, wenn Geschwindigkeit und Zuverlässigkeit entscheidend sind, wie bei gewissen Reaktionen bei der Flucht von Fischen und Insekten. Ebenso bei den Wirbeltieren ist die elektrische Kopplung durch Gap Junctions synchronisiert, die Kontraktionen des Herzmuskels und der glatten Muskelzellen, die verantwortlich für die Peristaltik des Darms sind. Gap Junctions können auch in vielen Geweben auftreten, die keine elektrisch erregbaren Zellen besitzen. Im Prinzip bietet der Austausch von kleinen Metaboliten und Ionen einen Mechanismus für die Koordinierung der Tätigkeiten der einzelnen Zellen in einem solchen Gewebe und zum Ausgleich von zufälligen Fluktuationen in kleinen Molekül-Konzentrationen in verschiedenen Zellen. In der Leber, zum Beispiel, stimuliert die Freisetzung von Noradrenalin aus sympathischen Nervenendigungen, auf Antwort auf einen Sturz des Blutzuckerspiegels, Hepatozyten zu Glykogenabbau und Freisetzung von Glukose in das Blut zu erhöhen. Nicht alle der Hepatozyten werden jedoch von sympathischen Nerven innerviert. Mit Hilfe der Gap Junctions, die Hepatozyten verbinden, wird das Signal von den Hepatozyten zu den innervierten zu den nicht-innervierten übertragen. Mäuse mit einer Mutation im major Konnexin-Gens in der Leber, sind nicht in der Lage, Glucose zu mobilisieren, wenn der Blutzuckerspiegel fällt. Cell Biology SS12 (Kovarik, Martens) Seite 16 von 65 Christian Husch Kovarik – Foliensatz 8: ECM – extrazelluläre Matrix: komplexes Netzwerk von Polysacchariden (wie Glykosaminoglykane oder Cellulose) und Proteine (z. B. Kollagen), durch Zellen sekretiert. Dient als Strukturelement in Geweben und hat auch Einfluss auf ihre Entwicklung und Physiologie. Zellen, sind durch Räume, die mit extrazellulärer Matrix gefüllt sind, umgeben. Zwei Hauptklassen der extrazellulären Makromoleküle bilden die Matrix: (1) Polysaccharid-Ketten der Klasse Glykosaminoglykane (GAG), die in der Regel kovalent an Protein in Form von Proteoglykanen verknüpft vorgefunden werden. (2) Faserproteine, einschließlich Kollagen, Elastin, Fibronectin und Laminin, die sowohl strukturell als auch klebende Funktionen haben. ECM kann verkalken, um die steinharten Strukturen von Knochen oder Zähne zu bilden ECM bildet die transparente Matrix der Hornhaut ECM kann die strickartige Organisation adaptieren, die Sehnen ihre enorme Zugfestigkeit gibt an der Grenzfläche zwischen einer Epithel und Bindegewebe, bildet das ECM eine Basallamina Hyaluron: Funktionen: eine Rolle beim Widerstand gegen Druckkräfte in Gewebe und Gelenke wichtig als Raumfüller während der embryonalen Entwicklung Hyaluron wird von der basalen Seite eines Epithels synthetisiert was oft dazu dient, einen Zell-freien Raum, in denen Zellen wandern können, zu schaffen, die bei der Bildung des Herzens, der Hornhaut, und mehrere andere Organe mit wirken (falls Zellmigration endet, wird der Überschuss an Hyaluron in der Regel durch das Enzym Hyaluronidase abgebaut). Hyaluronsäure wird auch in großen Mengen während der Wundheilung produziert ein wichtiger Bestandteil der Gelenkflüssigkeit, wo es als Gleitmittel dient. Proteoglycan: Proteoglykane, so wird angenommen, spielen eine große Rolle bei der chemischen Signalübertragung zwischen den Zellen. Sie binden verschiedene sezernierte Signalmoleküle, wie zB bestimmte Protein-Wachstumsfaktoren und verstärken oder inhibieren ihre Signalaktivität. Beispielsweise binden die Heparansulfatketten von Proteoglykanen an Fibroblasten-Wachstumsfaktoren (FGF), das eine Vielzahl von Zelltypen zur Proliferation stimuliert; diese Wechselwirkung oligomerisiert die Wachstumsfaktor-Moleküle, so dass sie sich vernetzen und aktivieren ihre Rezeptoren an der Zelloberfläche, welche Transmembran-Tyrosin-Kinasen sind. Proteoglykane binden und regulieren die Tätigkeit von anderen sekretierenden Proteinen, einschließlich proteolytische Enzyme (Proteasen) und Protease-Inhibitoren. Die Bindung an ein Proteoglycan kann die Aktivität von einem sekretierten Protein auf folgende Arten ändern: kann das Protein immobilisieren in der Nähe der Stelle, wo es hergestellt wurde und schränkt so sein Aktionspotential ein es kann sterisch die Aktivität eines Proteins blockieren Es kann ein Reservoir des Proteins zur verzögerten Freisetzung bereitstellen kann das Protein vor proteolytischem Abbau schützen, was eine Verlängerung seiner Tätigkeit bewirkt sie kann das Protein verändern oder konzentrieren, für eine wirksamere Präsentation an Zelloberflächenrezeptoren. Kollagene: Die Kollagene sind eine Familie von faserigen Proteinen, welche in allen vielzelligen Tieren gefunden werden. Sie werden von Bindegewebszellen, sowie durch eine Vielzahl von anderen Zelltypen sezerniert. Als Hauptbestandteil von Haut und Knochen, sie sind die am häufigsten vorkommenden Proteine bei Säugetieren, welche 25% der gesamten Proteinmasse diesen Tieren bilden. Selektine: Selektine sind Kohlenhydrat-bindende Proteine (Lectine) auf der Zelloberfläche, die eine Vielzahl von transienten Ca++-abhängigen, Zell-Zell-Adhäsion-Wechselwirkungen in der Blutbahn vermitteln. Es gibt mindestens drei Arten: L-Selectin auf weißen Blutkörperchen, P-Selectin auf Thrombozyten und Endothelzellen, die lokal durch eine entzündliche Reaktion aktiviert werden E-Selektin auf aktivierten Endothelzellen. Cell Biology SS12 (Kovarik, Martens) Seite 17 von 65 Christian Husch Jedes Selektin ist ein Transmembranprotein mit einer hochkonservierten Lectin-Domäne, die ein bestimmtes Oligosaccharid auf eine andere Zelle bindet. Die Struktur und Funktion der Selektine. (A) Die Struktur von P-Selectin. Das Selektin wird an der Aktin-Zytoskelett durch Anker-Proteine. (B) Wie Selektine und Integrine vermitteln die Zell-Zell-Adhäsionen für ein weißes Blutkörperchen die Migration aus der Blutbahn in das Gewebe. Die Zell-Zell-Adhäsionen von beiden Selektinen und Integrinen sind heterophil: Selektine binden an spezifische Oligosaccharide auf Glykoproteinen und Glykolipiden, während Integrine an spezifische Proteine binden. Cell Biology SS12 (Kovarik, Martens) Seite 18 von 65 Christian Husch Fragen aus der Fragensammlung: 1. Was ist ein checkpoint? Erkläre anhand eines Beispiels? Ein Checkpoint ist ein jener Punkt im Zellzyklus, bei dem der Zyklus solange angehalten wird, bis alle nötigen Vorgänge korrekt ausgeführt worden sind. Beispiel: Spindel-Checkpoint In der Metaphase ordnen sich die Chromosomen in der Aquatorialebene an. Die Spindelapparat setzt an den Chromosomen an. Normalerweise binden die Kinetorchor-Mikrotubuli an die Kinetochore. Findet diese Bindung nicht statt, so kann statt dessen Mad2 an die Kinetochore binden. Infolge dessen findet eine Konformationsanderung statt und Mad2 bindet nun cdc20. Damit kann cdc20 den APCKomplex nicht aktivieren. Somit kann Securin nicht von der Separase getrennt werden und die Schwersternchromatiden werden nicht getrennt (Kohesin bleibt erhalten) 2. CdK Aktivität? Wie wird sie gesteuert? CdK – cyclin dependent kinase cdks sind nur aktiv, wenn Cycline an ihnen gebunden sind und sie somit aktivieren. Durch die Bindung kommt es zur Konformationsänderung und es wird eine ATP-Bindestelle frei. Ohne Cyclin-Bindung befindet sich cdk im inaktiven Zustand und ist durch eine Region des Proteins, der T-Schleife, blockiert. Durch Bindung des Cyclin wird die T-Schlaufe aus der aktiven Stelle verdrängt und es kommt zu einer partiellen Aktivierung von Cdk2. Die Phosphorylierung eines Threonin-Restes an der T-Schleife des Cdk2 durch CAK (cdk-aktivierende Kinase) führt zur Aktivierung des Enzyms durch Wechsel der Form der T-Schleife, wodurch sich die Fähigkeit des Enzyms verbessert, an die Proteinsubstrate zu binden. Dadurch ist der cdk-cyclin-Komplex vollständig aktiv. 3. Merkmale einer apoptotischen Zelle? Zellschrumpfung, Kondensation von Organellen Chromatin-Kondensation, Fragmentierung der DNA Zelle "Blasenbildung", die Integrität der Zellmembran ist erhalten, aber Phosphatidylserin transloziert von der Innenseite der Zellmembran auf die äußere Oberfläche Zelle zerfällt in apoptotische Körper (kein Entweichen des Zellinhalts! keine Entzündung) apoptotischen Zellen/apoptotische Körper werden von Fresszellen (z. B. Makrophagen) durch Phagozytose (keine Entzündung) entfernt 4. Was verhindert, dass sich der Zellkern in G2 und S-Phase nochmals repliziert (re-replication Block)? Die Zelle repliziert ihre DNA nur 1 Mal im Zellzyklus. In einem Experiment (1970) wurde dieser Kontrollmechanismus nachgewiesen, indem a) eine S-Phase Zelle mit einer G1-Phase Zelle fusioniert wurde. Der Zellkern, der in G1 war, wechselte sofort in die S-Phase: In der S-Phase ist die S-Phase-CdK Aktivität hoch → die S-Phase CdKs treiben den G1 Kern ebenfalls in die S-Phase. b) Bei der Fusion von G2-Zelle und S-Phase-Zelle blieben beide Kerne in der jeweiligen Phase (die G2-Zelle hat nicht erneut angefangen, DNA zu replizieren): Der G2 Zellkern verändert trotz S-Phase-CdKs seinen Zellzyklusstatus nicht. Hier ist ein Block vorhanden, der verhindert, dass die Zelle nochmalsrepliziert. c) Bei Fusion von G1 und G2 bleiben die Kerne auch in der jeweiligen Phase: Kontrollexperiment → G1 Zellkern kann nicht durch G2 Kern in die S-Phase kommen. Molekularer Mechanismus Während des ganzen Zellzyklus bindet ORC an einen replication origin. In G1 wird durch steigende G1-Cycline/ G1-CdK Aktivität Cdc6 hochreguliert, das an ORC bindet. Dieser Komplex kann wiederum Mcm-Proteine rekrutieren, die einen Ring um die DNA bilden. Ein pre-replikativer Komplex entsteht. In der S-Phase wird Cdc6 phosphoryliert, was zu dessen Abbau führt. Gleichzeitig wird ORC phosphoryliert und somit die DNA-Polymerase rekrutiert. Die Mcm Proteine wandern an der DNA als Helicasen entlang. Der G2-Zellkern kann nicht nochmals replizieren, weil Cdc6 nicht mehr da ist, somit Mcm nicht binden kann und kein Replikationskomplex entsteht. Cdc6 Mutanten arretieren in der G1 Phase. Cell Biology SS12 (Kovarik, Martens) Seite 19 von 65 Christian Husch 5. Regulation der Trennung der Schwesterchromatiden. In der Metaphase lagern sich die Chromosomen in der Äquatorialebene an. Die Schwesterchromatiden sind durch Cohäsine zusammengehalten. Die Länge der Kinetochormikrotubuli ist noch gleich (gleiche Polymerisation/ Depolimerisation am Plus/Minus Ende. In der Anaphase werden die Kinetochormikrotubuli durch überwiegende Depolymerisation an beiden Enden kürzer. Die Chromosomen werden dadurch zu den Spindelpolen bewegt (Anaphase A). Anaphase B: polare Mikrotubuli werden länger, weil anti-parallele Mikrotubuli aneinander vorbei gleiten und schieben die Centrosomen voneinander weg. Die Spindel verlängert sich zusätzlich. Spindle attachment checkpoint: Gleichzeitig mit der Verkürzung der Mikrotubuli müssen die Cohäsinringe gespalten werden. Das passiert dadurch, dass APC (anaphase promoting complex) durch Cdc20 aktiviert wird. In der Folge wird Securin ubiquitinyliert und abgebaut, das normalerweise die Separase inaktiv hält. So wird die Separase aktiv und kann eine Untereinheit des Cohäsinkomplexes spalten. Eine zusätzliche Timing Kontrolle geschieht (zumindest in der Bäckerhefe) durch Phosphorylierung des Cohäsinskomplexes (durch Polo Kinase) an der Cleavage site, kurz bevor die Anaphase beginnt. Normalerweise binden die Kinetorchor-Mikrotubuli an die Kinetochore. Findet diese Bindung nicht statt, so kann statt dessen Mad2 an die Kinetochore binden. Infolge dessen findet eine Konformationsänderung statt und Mad2 bindet nun Cdc20 → APC kann nicht aktiviert werden und die Schwesterchromatiden können sich nicht trennen. 6. Warum eignet sich Hefe gut für Zellzyklusstudien? Man kann die einzelnen Phasen des Zellzyklus gut erkennen, da die Buds der Sprosshefe u.a. durch ihre Grose die Phasen widerspiegelt. Auserdem lassen sich einfache genet. Analysen durch induzierte Mutationen durchfuhren. Singlemutationen mit einem geanderten Phanotyp lassen sich in haploiden Hefen untersuchen. Hefen lassen sich haploid als auch diploid kultivieren. 7. Asymmetrische Zellteilung an einem Beispiel (Bäckerhefe) (unterschiedliche Zelltypen nach der Teilung): In nährstoffreichem Medium sind die Zellen oval und haben ein ganz bestimmtes Teilungsmuster. In einem nährstoffarmen Medium wachsen die Zellen in die Länge und knospen am distalen Ende, es entstehen sogenannte Pseudohyphen. Haploide Zellen in einem Medium, das Pheromone des anderen Mating Types enthält, stoppen ihren Zellzyklus in G1 und wölben sich in die Richtung der Partnerzelle ("Shmoo" Form). Die Teilung der haploiden Hefe erfolgt axial, die der diploiden sowohl axial als auch bipolar. Bei der axialen Teilung erfolgt die Bildung der neuen Knospe unmittelbar neben/bei der Knospungsnarbe der vorherigen Teilung. Bei der bipolaren Teilung entsteht die neue Knospe distal oder proximal zu dieser Knospungsnarbe. Zum Budding kommt es durch Myosin V (Motorprotein) und der Verteilung von Actin patches und Actin cables. Damit die Zellen nach der Teilung unterschiedlichen Charakter haben, muss es bereits vorher eine asymmetrische Verteilung bestimmter Komponenten geben: Ein Schlüsselevent ist die asymmetrische Verteilung von mRNA. Die Mutterzelle transkribiert eine mRNA von ash1. Das Protein ash1 fungiert als Repressor des Maitingtype-Switching (inhibiert die HO- Endonuclease). Während des Buddings wird die mRNA in 2/15 den Bud transportiert. Dies geschieht auf Grund einer Sequenz am 3’ Ende. Diese Sequenz wird von She2 erkannt und anschließend bindet She3 und MyosinV (ein Motorprotein). Ash1 ist somit nur noch in der Tochterzelle vorhanden und inhibiert die HO- Endonuclease. Diese ist nun in der Mutter aktiv und bewirkt, dass der MAT-Lokus herausgeschnitten wird und durch den anderen ersetzt wird. Beide MAT Typen (a und α) sind als unabhängige Kopien an anderer Stelle erhalten. Daher kann nur die Mutterzelle den Matingtyp ändern. Cell Biology SS12 (Kovarik, Martens) Seite 20 von 65 Christian Husch 8. Mechanismus der Regelung der Apoptose durch äußere Faktoren. extrinsischer Weg: FasR/ FasL In der Zellmembran sitzt der FasR mit einer extrazellularen und einer intrazellularen Domane. Bindet an die extrazellulare Domane der FasL, so kommt es intrazellular zur Trimerisierung des Rezeptors. Die Trimerisierung hat zur Folge, dass die Death Domaine binden kann und weitere Adaptorproteine rekrutiert, wie Cas-8. Diese schneidet (aktiviert) Cas-3, welche wiederum ICAD (inaktive Caspase Aktivierende DNase) aktiviert. CAD wandert schließlich in den Kern undfragmentiert die DNA. TNF TNF – tumor nekrose faktor, produziert von T-Zellen TNFR ist ein Membranrezeptor, der TNF bindet. Dadurch kommt es zur Trimerisierung und zum Clustering der Death Domain. Somit kann das Adaptermolekul TRADD (TNFR associated death domain) binden. Dies rekrutiert wiederum Traf-2 und aktiviert den NFκB bzw. JNK Pathway. Bindet jedoch FADD statt TRADD, so wird die Procaspase-8 rekrutiert und die Apoptose eingeleitet. Intrinsischer Weg: Intrazelluläre Aktivierung. Mitochondrien setzen Cytochrom c frei. die bindet an Apaf-1 und bewirkt, dass die Aggregation des AdapterProtein Apaf-1. Apaf-1 bindet und aggregiert Procaspase-9-Moleküle, was zur Spaltung dieser Moleküle und zur Auslösung einer Caspase-Kaskade führt. Andere Proteine, die Apoptose bewirken, werden auch vom mitochondrialen Intermembranraum (zB Apoptose induzierenden Faktors (AIF)) freigegeben. 9. Wie funktioniert der DNA-damage Checkpoint? In Säugerzellen: Arretieren der Zelle in G1 durch DNA-Schäden: Wenn DNA beschädigt ist, werden Proteinkinasen aktiviert, die p53 phosphorylieren. Im Normalfall bindet Mdm2 an p53 und stimuliert dessen Ubiquitinierung und Abbau in den Proteasomen. Durch die Phosphorylierung wird diese Bindung an p53 jedoch inhibiert. In der Folge akkumuliert p53 in der Zelle und stimuliert die Transkription des p21 codierenden Gens. P21 inaktiviert G1/S-Cdk und S-Cdk Komplexe, die Zelle arretiert in G1. Alternativ können DNA Schäden auch zur Phosphorylierung oder verminderten Produktion von Mdm2 führen, was durch steigende p53 Level ebenfalls den gleichen Effekt hat. In Hefe: zwei Checkpoints a) G1 checkpoint: Wenn ein DNA Schaden detektiert wird, Mating Pheromone vorhanden sind oder die Zelle die kritische Größe nicht erreicht hat, arretiert die Zelle in G1 und verhindert den Übergang zur Mitose. b) Spindle assembly checkpoint: Wenn DNA Schäden entdeckt werden, die DNA nicht vollständig repliziert wurde oder Chromosomen nicht auf der Metaphasen-Platte angeordnet sind, bleibt die Zelle in der Metaphase stehen und es erfolgt kein Übergang zur Anaphase, die Schwesterchromatiden werden nicht getrennt, die Zelle teilt sich nicht. Zuständig für die Checkpointkontrolle sind CdK/Cyclin Komplexe. Regulation erfolgt durch die Synthese von Cyclinen durch verschiedene Transkriptionsfaktoren, die Degradation von Cyclinen, Assoziation mit CdK-Inhibitoren, Phosphorylierung/Dephosphorylierung…. 10. Welche Möglichkeiten gibt es, um Zellen zu synchronisieren? Um Zellen zu synchronisieren, d.h. Zellen einer Zellkultur in das selbe Zellzyklusstadium zu bringen, gibt es diverse Möglichkeiten: a) Mating Pheromone in Hefe (block in G1) b) Temperatur-sensitive Mutationen in Hefe c) Mikrotubuli Drugs: - Mikrotubuli-stabilisierend: Taxol → block in Mitose - Mikrotubuli-destabilisierend: Nocodazol → block in G1 d) "Aushungern"(Serum Entzug, Säugerzellen → block in G0) e) Isolation von 1n oder 2n Zellen (durch FACS) f) mitotic shake-off of adherent mammalian cells Cell Biology SS12 (Kovarik, Martens) Seite 21 von 65 Christian Husch 11. Autophagie: Funktion, Stadien der Autophagie Autophagie beinhaltet dynamische Membran-Umlagerung, um Teile des Zytoplasmas zu verschlingen, und es hat die Kapazität für die Speicherung von großen Strukturen, wie ganze Organellen. Hierbei werden Teile des Zytoplasmas in ein de novo-gebildetes Doppel-Membran-Vesikel (bezeichnet als Autophagosom) abgesondert. Anschließend verschmilzt das Autophagosom mit dem Lysosom/Vakuole und die inneren einzelnen Membranvesikel werden in das Lumen freigegeben. In jedem Fall wird die Membran des resultierenden autophagischen Körpers lysiert, um damit die phagozytierten Komponenten aufzuschlüsseln, und die resultierenden Makromoleküle zurück in das Cytosol, durch die Membran-Permeasen, zu transportieren zur Wiederverwendung. Grundlegende (basale) Autophagie: - Umatz von langlebigen Proteinen - Entfernung von beschädigten Organellen - Rolle in Verlängerung der Lebensspanne? Induzierte Autophagie: Zelluläre Antwort auf unterschiedlichste Streßverhältnisse (zB limitierter Nährstoff, Hitze, Oxidative Ereignisse) - Eliminierung von invasiven Pathogenen inkl. Viren, Parasiten und Bakterien Autophagie ist in einer Vielzahl von Erkrankungen, wie Krebs und neurodegenerative Erkrankungen wie Alzheimer, Parkinson und Chorea Huntington, involviert. 12. Apoptose: Vorgang, Regulation, Inhibition: Keyplayer der Apoptose: Die Hauptrolle in der Apoptose spielt eine bestimmte Klasse von "Selbstmord" - Proteasen, die CASPASEN. (Sie haben Cystein im aktiven Zentrum und spalten ihre Zielproteine bei Aspartat - daher der Name Caspase). Jede Caspase liegt zunächst als inaktives Proenzym vor (=Procaspase). Aktivierung erfolgt i.d.R. durch proteolytische Spaltung durch ein anderes Mitglied der Caspase Familie. Da jedes Caspase Molekül mehrere Procaspase Moleküle spalten kann, welche ihrerseits wiederum viele Procaspasen spalten können, kommt es zu einer Kaskade → amplifying chain reaction, durch eine kleine Zahl von Initiator Caspasen. Einige der aktivierten Caspasen, die Effektor Caspasen, spalten dann eine Reihe von Schlüsselproteinen in der Zelle (z.B. bestimmte Cytosolproteine und Zellkernlamina) was letztendlich zum kontrollierten Zelltod führt. Eine solche Caspase-Kaskade kann z.B. von den Mitochondrien ausgehen: Cytochrom C als proapoptotischer Faktor trägt zur Bildung des Apoptosoms (=eine "Plattform", die Caspase 9 aktiviert) und in der Folge zur Aktivierung der Caspase-Kaskade bei. → intrinsischer Pathway Regulation: extrinsischer Weg: Fas/ FasL Ein Killer Lymphozyt mit Fas Liganden bindet an die Zielzelle und aktiviert Fas Proteine an deren Oberfläche. Im Zellinneren können nun Adaptorproteine (enthalten death domains) an Fas binden, wodurch Procaspase-8 Moleküle binden und in Folge gespalten werden können. Durch die aktivierte Caspase 8 kann die Caspase Kaskade gestartet werden. (Caspase 8 spaltet Caspase 3, diese aktiviert CAD, welches die DNA fragmentiert.) Cell Biology SS12 (Kovarik, Martens) Seite 22 von 65 Christian Husch TNF Der Tumor Nekrose Faktor wird von T-Zellen und aktivierten Makrophagen produziert (als Antwort auf eine Infektion). In manchen Zellen kann TNF Apoptose induzieren, wenn es auch i.d.R. als alleiniger Faktor für die Einleitung der Apoptose nicht ausreicht. Dabei bindet TNF an den Membranrezeptor TNFR. Es kommt zur Rezeptor-Trimerisierung und zum intrazellulären Clustering von Death Domains. Somit kann das Adaptermolekül TRADD (TNFR associated death domain) binden. Dieses kann zur Aktivierung des NFκB bzw. JNK Pathways führen oder mit FADD interagieren, wobei die Pro-Caspase-8 gespalten wird und somit die Apoptose induziert wird. Intrinsischer Weg: Intrazelluläre Aktivierung. Mitochondrien setzen Cytochrom c frei. die bindet an Apaf-1 und bewirkt, dass die Aggregation des AdapterProtein Apaf-1. Apaf-1 bindet und aggregiert Procaspase-9-Moleküle, was zur Spaltung dieser Moleküle und zur Auslösung einer Caspase-Kaskade führt. Andere Proteine, die Apoptose bewirken, werden auch vom mitochondrialen Intermembranraum (zB Apoptose induzierenden Faktors (AIF)) freigegeben. Inhibierung: In einer lebensfähigen Zelle, gibt es die proapoptotischen Bcl-2- Familienmitglieder Bax, Bak und BH3 – nur Proteine werden durch antiapoptotischen Elemente, wie Bcl-2, antagonisiert. Als Reaktion auf einen apoptotischen Stimulus, werden nur BH3 Mitglieder durch transkriptionelle Hochregulation (Bax, Noxa, Puma), subzelluläre Relokalisation (Bim, BMF), Dephosphorylierung (Schlecht) oder Proteolyse (Bid) aktiviert. Aktivierte nur- BH3-Proteine schützen antiapoptotische Bcl-2-Mitglieder vor inhibierenden proapoptotischen Mitgliedern. Darüber hinaus könnten sie direkt eine Konformationsänderung von Bax und Bak induzieren, die anschließend oligomerisieren und in der mitochondrialen Membran inserieren und dort Poren entweder selbst oder durch den Umgang mit dem permeability transition pore komplex bilden. Infolgedessen werden proapoptotischen Faktoren, wie Cytochrom c, aus der inneren mitochondrialen Membran in das Cytosol entlassen, das zur Bildung des Apoptosom und die anschließende Aktivierung der Caspase-Freigabe beiträgt. 13. Detektion der Apoptose: - DNA laddering: Beobachtung von Apoptose aufgrund Apoptose-induzierter DNA Fragmentation - Tunel assay: Detektion von Apoptose-induzierten Brüchen im DNA Strang. Während der Apoptose entstehen durch DNAse Aktivität nicht nur kleine doppelsträngige DNA-Fragmente, sondern auch Strangbrüche (Nicks) in DNA mit hohem Molekulargewicht. Das kann man nachweisen, indem man die 3'-OH Enden mit TdT (=terminal transferase) markiert, welche an alle 3'-OH Enden markierte Nukleotide anhängt. - Annexin V staining assay: Detektion von Phosphatidylserin an der Außenseite der Zellmembran. Phosphatidylserin dient als Erkennungsmerkmal für die Phagozyten, wenn es sich an der Membranaußenseite befindet. In gesunden Zellen wird es von einer ATP abhängigen Translokase auf die Membraninnenseite gebracht. Annexin V interagiert mit Phosphatidylserin (bzw. allgemein mit Phospholipiden) und dient somit als früher Indikator für Apoptose. 14. Erkläre kurz Replicative Senescence und Contact inhibition: Replicative senescence Senescence = Zellen teilen sich nicht/replizieren keine DNA mehr, aber sie sterben auch nicht. Es ist von vornherein festgelegt, wie oft sich menschliche Zellen teilen können. Die meisten somatischen Zellen enthalten keine Telomerase (im Gegensatz zu Keimzellen und diversen Stammzellen), d.h. in jeder Replikationsrunde werden die Telomere etwas kürzer. Da die Telomere lange repetitive Sequenzen ohne codierende Sequenzen enthalten, hat das bis zu einem bestimmten Grad keine Auswirkung auf die Zelle. (Zusätzlich sind die Telomerenden von Proteinkomplexen geschützt und das freie Ende durch looping geschützt) Eine menschliche Zelle verliert durchschnittlich 2-4kb an Telomerlänge im Verlauf des Lebens. 8-12kb lang sind die Telomere von Geburt an → verkürzte Telomere könnten für diverse Alterserscheinungen verantwortlich sein. Contact inhibition Zellen in Zellkultur wachsen nur so lange, bis ein Monolayer entstanden ist. Tumorzellen ignorieren die schon bestehenden Kontakte und wachsen unkontrolliert übereinander. Zellen "fühlen" nicht nur, dass Nachbarn vorhanden sind, sie brauchen sogar Kontakte, um zu überleben. Alle Zellen mit Ausnahme von Blutzellen wachsen adherent. In Experimenten konnte gezeigt werden, dass Zellen (Fibroblasten) dabei sehr wohl zwischen Unterlage und Nachbarzelle unterscheiden können. Cell Biology SS12 (Kovarik, Martens) Seite 23 von 65 Christian Husch 15) Wie (Prinzip!) binden Kinetochormikrotubuli an den Kinetochoren? Welche Proteine sind dabei beteiligt? Wie wird der Kontakt ermöglicht? Schritt 1: Das Kinetochor (KT) interagiert zunächst mit der seitlichen Oberfläche eines einzigen Mirkotubuli (MT) (seitlicher Ansatz), welches sich von einem Spindel-Pol (Spindel-Pol-MT) (rechts) erstreckt. Dieses Verfahren wird oft durch die Wechselwirkung zwischen einem KT-abgeleiteten MT und einer Spindel-Pol-MT (links) erleichtert. Es ist noch nicht bekannt, ob beide oder ob nur eines der Schwester-KTs sich an einem MT während der seitlichen Anlagerung anhaftet. Schritt 2: Sobald an einem Spindel-Pol-MT gebunden wurde, wird das KT entlang der MT-Mantelfläche in Richtung eines Spindel-Pols transportiert (gleiten, links). Anschließend wird, zumindest in Hefe, das KT am Ende eines Spindel-Pol-MT (End-Befestigung) angebunden und transportiert, wenn die MT schrumpft (Endanziehen, rechts). Schritt 3: Nach dem KT-Transport in Richtung eines Spindel-Pols, könnten beide Schwester KTs an MTs binden. Wenn beide Schwester-KTs an am gleichen Spindel-Pol eines MT binden, muss die KT-MT-Bindung umgeformt werden, bis die richtige Bi-Orientierung etabliert ist. Schritt 4: Der Umbau der KT-MT-Anlage stoppt, sobald Spannung über Schwester KTs nach Errichtung der BiOrientierung erzeugt wurde. Die Zahl der MTs, deren Plus-Enden mit einer einzigen KT befestigt sind, steigt, je mehr Spannung auf Metazoen-KTs aufgebracht wird, während. nur ein einziges MT an jedes Schwester-KT in der Bäckerhefe bindet. Schritt 5: Nachdem alle Schwester-KTs an der Spindel bi-orientiert sind, wird die Kohäsion zwischen den Schwesterchromatiden entfernt, wodurch Schwesternchromatid-Segregation zu entgegengesetzten Spindelpolen während der Anaphase beginnt. In der Bäckerhefe geschieht die Trennung der Schwesterchromatiden entlang eines Chromosomenarms vom dem Zentromer zum Telomer. Die End-on KT-MT-Anlage wird solange beibehalten, bis die MTs depolymerisieren und Schwester-KTs zu den gegenüberliegenden Polen, in der Anaphase A, gefolgt vom Pol-zu-Pol-Abstand , welcher in der Anaphase B vergrößert wird. 16. Wie können Zellen in einer Zellzyklusphase arretiert werden? Um Zellen in einer Zellzyklusphase zu arretieren gibt es diverse Möglichkeiten, z.B. mit bestimmten Chemikalien (Nocodazol → Spindelgift, das die Mikrotubuli destabilisiert, Zellzyklus stoppt in G1 bzw. mit Taxol gibt es einen Block in der Mitose). Entzieht man Säugerzellen das Serum, stoppen sie in G0. Mit temperatursensitiven cdc-Mutanten von Hefe kann der Zellzyklus an verschiedenen Stellen gestoppt werden (cdc genes = cell division cycle genes) Beispiel anhand der Bäckerhefe, welche in einer gewissen Zellzyklus-Phase verweilte: Die Morphologie der knospenden Zellen der Bäckerhefe wurde durch eine CDC-Mutation verhindert. In einer normalen Population proliferierender Hefezellen, variieren die Knospen in der Größe entsprechend des Zellzyklus-Stadiums. Cdc15-Mutanten, die bei eingeschränkter Temperatur gewachsen sind, beendeten zwar die Anaphase, aber konnten nicht den Austritt aus der Mitose und Zytokinese abschließen. Infolgedessen verblieben sie alle mit großen Knospen, die charakteristisch für die späte M-Phase sind. Wie DNA-Schäden den Zellzyklus in der G1-Phase verbleiben lassen: Wenn DNA beschädigt ist, werden Proteinkinasen, welche p53 aktivieren, phosphoryliert. Mdm2 bindet in der Regel an p53 und fördert seine Ubiquitinierung und Zerstörung in Proteasomen. Die Phosphorylierung von p53 blockiert die Bindung dessen Bindung an Mdm2, als Ergebnis akkumuliert p53 in hohen Konzentrationen und stimuliert die Transkription des Gens, das CKI-Protein p21 codiert. Das p21 bindet und inaktiviert G1/S-Cdk und S-CDK-Komplexe und hält die Zelle in G1. In einigen Fällen induziert DNA-Schädigung auch entweder die Phosphorylierung von Mdm2, oder eine Abnahme der Mdm2 Produktion, die einen Anstieg des p53 bewirkt. Arretierung durch Nährstoffmangel, bis Nährstoffe wieder ausreichend verfügbar sind. Arretierung durch biochemisches Ungleichgewicht. Wenn für die Zellteilung wichtige Enzyme/Proteine wieder verfügbar sind, teilt sich die Zelle weiter. Cell Biology SS12 (Kovarik, Martens) Seite 24 von 65 Christian Husch 17. Unterschiede in der Morphologie von nekrotischen und apoptotischen Zellen Nekrose: • Organellen angeschwollen • Chromatin klumpt • Mitochondrien haben schwammige Struktur • Zelle zerfällt komplett Zellinnere wird freigesetzt Entzündungsreaktionen Apoptose: • Chromatin kondensiert sehr stark • Zellkern zerfällt in kleinere Teile (Nuclear fragmentation) • Membran macht Ausstülpungen = Blebbing / Blebs • Blebs machen apoptotic bodies17 • Zellinhalt bleibt in den apoptotic bodies diese von Phagozyten aufgenommen • was in der Zelle war kommt nie nach außen (bei Nekrose schon!) 18. Wie kann die Zelle wissen, daß sie die richtige Größe hat: • Mechanismus ist noch nicht komplett geklärt • folgendes Modell ist eine mögliche Erklärung • Cln 3: Cyclin das ein bisschen aus der Reihe tanzt, man beobachtet keine Akkumulation, wird in G1 gebraucht • Zellen ohne Cln 3 Zellen werden kleiner Cln 3 spielt eine Rolle bei der Erkennung der Größe • Menge an DNA verändert sich in G1 nicht • auf der DNA Proteine (ev. Histone, könnten aber auch andere sein) quantitiv, dh. bedecken die DNA • Cln 3 kann nicht direkt an DNA binden aber an das Protein (rot auf Folie) • Zellteilung passierte wenn sich die Zelle teilt, hat die Tochterzelle weniger Material als sie vorher hatte • Cln 3 nimmt zu • wenn jedes rote Protein von Cln 3 besetzt ist kann freies Cln 3 an CdK binden und CdK auf diese Weise aktivieren • wenn Cln 3 an rotes Protein gebunden hat kann es nicht gleichzeitig an CdK binden • Check-point aktiviert Zelle verlängert den Zellzyklus solange der Check-point aktiviert ist • Check-point funktioniert vermutlich so, wie das Modell mit Cln 3 19. Wie wird in der Mitose APC reguliert? • Abbau von Clbs notwendig damit Zelle in Zytokinese geht • Aktivieren von APC durch Cdc 20 (APC ansich nicht aktiv) • APC wird erst gegen Ende der Mitose abgebaut • mitotische CdK-Aktivität sinkt durch APC • Cdc 20: hochreguliert durch mitotische Aktivität der CdK • wenn genug von der mitotischen CdK-Aktivität da ist dann wird APC aktiviert durch Cdc 20 = Feedback-Mechanismus • wenn CdK-Aktivität verschwindet Cdc20-Aktivität sinkt wieder APC wieder inaktiv • Cdc20: Cell division cycle Gen: Mutation Zellen bleiben in der Mitose hängen 20. Checkpoints der Hefe • die Hefe hat 2 wichtige Check-points: • G1-Check-point: überprüft DNA-Schäden, Mating Pheromon und Größe der Zelle, wenn das alles passt START: Zelle ist verpflichtet den Zellzyklus zu durchlaufen • Spindle-assembly-Check-point: MT zerstört Zelle bleibt in Mitose stehen, prüft ob DNA-Schäden vorhanden sind, ob DNA vollständig repliziert ist, ob Chromosomen in der Äquatorebene sind wenn Check-point nicht exekutiert wird, darf die Zelle die Schwesterchromatiden trennen • Abbau von Cyclinen beim Spindle-assembly-Check-point verhindert Arrest • CdK-Aktivität inhibieren (durch Synthese von CdK-Inhibitor) Zellen gehen nie in S-Phase • Phosphorylierung von Cdc28 Arrest • Synthese von Cyclinen Arrest • Cycline primär über Abbau gesteuert: in jeder Zellzyklusphase mehr oder weniger gleich stark exprimiert, aber Proteinlevels unterschiedlich, weil in unterschiedlichen Phasen unterschiedlicher Abbau • manche Cycline aber auch unterschiedlich exprimiert Transkription von Zyklinen mit Hilfe von Transkriptionsfaktor hoch- oder runterreguliert Cell Biology SS12 (Kovarik, Martens) Seite 25 von 65 Christian Husch 21. CdK-Aktivität? Wie wird sie gesteuert? CdKs (Cyclin dependent kinases) und Cycline sind zwei Schlüsselkomponenten der ZellzyklusKontrollsystems. Der CdK-Cyclin Komplex hat Kinasefunktion, um bestimmte Zellzyklusevents auszulösen. Ohne das jeweilige Cyclin sind die CdKs inaktiv. Aktivierung: Im inaktiven Zustand ohne Cyclin, ist die active Site des CdKs durch einen sogenannten T-loop blockiert. Durch die Cyclinbindung wird der T-Loop von der active site verdrängt, der Komplex ist nun teilweise aktiviert. Erst durch die Phosphorylierung einer Stelle im T-Loop (durch CAK - CdK aktivierende Kinase) ändert sich dessen Struktur und das Enzym erlangt die volle Aktivität. Inhibition : Durch das Dissoziieren von Cyclin werden CdKs wieder inaktiv. Jedoch kann es durch Phosphorylierung bestimmter Stellen nahe der active site auch zur Inhibition des Komplexes kommen. 22. Warum ist Hefe besonders gut geeignet Zellzyklus zu analysieren? Zum einen kann man die einzelnen Phasen des Zellzyklus gut erkennen, da die Buds der Sprosshefe durch ihre Größe und Form ("Shmoo") die jeweilige Zellzyklusphase widerspiegelt. Außerdem lässt sich genetische Manipulation durch induzierte Mutationen einfach durchführen. Hefen lassen sich sowohl haploid als auch diploid kultivieren. Das ist sehr nützlich, da sich im haploiden Organismus Single Mutationen schon in einem Phänotyp äußern. [S.cerevisiae, S.pombe] Besonders wichtig: Man kann T-sensitive Mutanten erzeugen. Wenn ein mutiertes Gen im Zellzyklus eine Rolle spielt arretiert die Zelle dort, wo es gebraucht werden würde. → z.B. cdc Mutanten: die Zelle kommt zu dem Punkt im Zellzyklus wo das Gen notwendig ist, zusätzlich lässt sich der Zellzyklusstatus bei Hefe phänotypisch gut erkennen. 23. Voraussetzungen für Zellzyklus (WICHTIG!!!) • „Uhr“, die ein Ereignis erst dann einschaltet wenn es gebraucht wird (nicht vorher und nicht nachher) • Mechanismus, der die Reihenfolge der Ereignisse vorgibt • binary switches: gewährleisten, dass ein Ereignis während des Zellzyklus nur einmal vorkommt (z.B.: DNA-Replikation) • Mechanismen, die gewährleisten, dass alles robust ist (voll eingeschalten und voll abgeschalten, nicht „ein bisschen“) Feedback-Mechanismen • Zellen müssen Signale von außen annehmen (z.B.: Mating factor) 24. Funktion von SNARE Die meisten intrazellulären Membranfusionen werden gesteuert durch SNARE-Proteine. Studien bei Hefe- und Säugetierzellen haben gezeigt, daß verschiedene SNARE-Proteine vorhanden sind und verschiedene Schritte benötigen, um intrazellulären Transportschritte durchzuführen. Alle SNARE-Proteine enthalten mindestens ein SNARE-Motiv. Manche SNAREs haben also eine C-terminale Transmembran-Domäne, welche unmittelbar der SNAREDomäne folgt. Die transmembrane-enthaltenden SNARE-Proteine sind Schwanz-verankernde Proteine, die zuerst im Cytosol produziert werden und dann posttranslational in das ER eingefügt werden. Manche SNARE-Proteine enthalten keine Transmembran-Domäne und interagieren mit der Membran mit anderen Mitteln (zB Lipid-Modifikationen). Struktur des SNARE-Komplexes: Vier einzelne SNARE-Motive fügen sich zur einem sehr stabilen 4-helikalen Bündel zusammen. (rote Box). Die Helices der Transmembrandomäne beinhalten SNARE-Ausläufer, die sich in die Membran erstrecken. Mindestens eine Transmembrandomäne mit SNARE muß in jedem Kompartiment zur Fusion vorhanden sein. Cell Biology SS12 (Kovarik, Martens) Seite 26 von 65 Christian Husch 25. Wie wird gewährleistet, dass 2 Matingtypes in der Hefe entstehen? S. cerevisiae existiert als diploide und haploide Zelle. Beide Zelltypen vermehren sich durch Mitose, indem Tochterzellen von der Mutterzelle abknospen. Haploide Zellen können sich mit haploiden Zellen des anderen Mating Types fortpflanzen (a Zelle mit α Zelle und umgekehrt). Dabei entsteht eine stabile diploide Zelle. Diploide Zellen können unter Stressbedingungen (z.B. Nahrungsknappheit) Meiose betreiben und es entstehen vier haploide Sporen (2 a und 2 α Sporen). Gesteuert wird das Ganze durch den MAT-Lokus, der sich auf einem der 16 Chromosomen befindet. Dieser codiert in der haploiden Zelle entweder für a oder α. Die Transkription des Gens wird durch den MAT-Promotor reguliert, die Gene selbst sind regulatorische Gene, die u.a. die Produktion von Peptidhormomen kontrollieren. Diese a-Faktor bzw α-Faktor genannten Pheromone binden an Zellen des jeweils anderen mating types und bewirken eine Fusion der beiden Zellen. Es entsteht eine diploide "Zygote", die nun über die Gene beider Ausgangszellen verfügt und so eine Kombination von Genen, die ihr und ihren Nachkommen möglicherweise einen Vorteil verschafft. Das Vorliegen beider Allele a und α in diploiden Zellen bewirkt, dass das transkriptionelle Programm für diploide Zellen abläuft (z.B. die Einleitung einer Meiose). Im Genom der Zellen befinden sich aber auch immer inaktive Kopien der MAT-Gene. Die Zelle kann aktive MAT-Gen gegen eine der "silent copies" auszutauschen. Das wird durch das HO-Gen reguliert, das für eine Endonuclease codiert, die spezifisch DNA in der MAT Region schneidet. HO wird nur während der G1Phase in haploiden Hefe-Zellen exprimiert. Die durch HO entstehende Lücke wird durch eine der inaktiven MAT-Allele gefüllt, so dass ein neues Gen im aktiven MAT-Lokus vorliegt und so ein mating-type-switch möglich ist (allerdings nur in der Mutterzelle → siehe Frage 2) 26. Zellzyklus-Checkpoint + Beispiel Ein Checkpoint ist ein jener Punkt im Zellzyklus, bei dem der Zyklus solange angehalten wird, bis alles nötigen Vorgange korrekt ausgeführt worden sind. Beispiele: Spindel-Checkpoint In der Metaphase ordnen sich die Chromosomen in der Äquatorialebene an. Der Spindelapparat setzt an den Chromosomen an. Normalerweise binden die Kinetorchor-Mikrotubuli an die Kinetochore. Findet diese Bindung nicht statt, so kann statt dessen Mad2 an die Kinetochore binden. Infolge dessen findet eine Konformationsänderung statt und Mad2 bindet nun cdc20. Damit kann cdc20 den APC-Komplex nicht aktivieren. Somit kann Securin nicht von der Separase getrennt werden und die Schwersternchromatiden werden nicht getrennt (Kohesin bleibt erhalten). DNA-Damage Checkpoint: In Säugerzellen: Arretieren der Zelle in G1 durch DNA-Schäden: Wenn DNA beschädigt ist, werden Proteinkinasen aktiviert, die p53 phosphorylieren. Im Normalfall bindet Mdm2 an p53 und stimuliert dessen Ubiquitinierung und Abbau in den Proteasomen. Durch die Phosphorylierung wird diese Bindung an p53 jedoch inhibiert. In der Folge akkumuliert p53 in der Zelle und stimuliert die Transkription des p21 codierenden Gens. P21 inaktiviert G1/S-Cdk und S-Cdk Komplexe, die Zelle arretiert in G1. 27. Nenne die Typen des programmierten Zelltods Apoptose: Necroptose: Pyroptose: Stimuli Bindes des death-Receptors DNA-Schäden Reaktive Sauerstoff-Arten Erscheinungsbild Pyknosis DNA-Fragmentierung Plasmamembran-Blasen Konsequenzen Formierung von apoptotischen Körpern Phagozytose TNFR1-Bindung + SMAC mimetics TNFR1 ligation + Caspase-Inhibierung Cytoplasma-Schwellung Freisetzung von intrazellulären Komponenten Inflammation Bakterielle Infektion Virale Infektion Stroke Cytoplasma-Schwellung DNA-Fragmentierung Porenformation Organell-Schwellung Freisetzung von Cytokinen Inflammation Intrinsischer und Extrinsischer Pathway siehe Frage 12 Cell Biology SS12 (Kovarik, Martens) Seite 27 von 65 Christian Husch 28. Aufbau und Funktion der Extrazellulärmatrix ECM – extrazelluläre Matrix: komplexes Netzwerk von Polysacchariden (wie Glykosaminoglykane oder Cellulose) und Proteine (z. B. Kollagen), durch Zellen sekretiert. Dient als Strukturelement in Geweben und hat auch Einfluss auf ihre Entwicklung und Physiologie. Zellen, sind durch Räume, die mit extrazellulärer Matrix gefüllt sind, umgeben. Zwei Hauptklassen der extrazellulären Makromoleküle bilden die Matrix: (1) Polysaccharid-Ketten der Klasse Glykosaminoglykane (GAG), die in der Regel kovalent an Protein in Form von Proteoglykanen verknüpft vorgefunden werden. (2) Faserproteine, einschließlich Kollagen, Elastin, Fibronectin und Laminin, die sowohl strukturell als auch klebende Funktionen haben. ECM kann verkalken, um die steinharten Strukturen von Knochen oder Zähne zu bilden ECM bildet die transparente Matrix der Hornhaut ECM kann die strickartige Organisation adaptieren, die Sehnen ihre enorme Zugfestigkeit gibt an der Grenzfläche zwischen einer Epithel und Bindegewebe, bildet das ECM eine Basallamina 29. Aufbau und Funktion der Tight-Junctions? Kommen in Vertrebraten vor. Tight Junctions befinden sich im apikalen Bereich der seitlichen Zellmembran und stellen dadurch, dass sie den Raum zwischen den Zellen abdichten, einen sehr engen Kontakt zwischen den Membranen zweier benachbarter Zellen her. Diese Kontakte bilden eine Diffusionsbarriere, die den parazellulären Transport von gelösten Molekülen, Proteinen und Lipiden über das Epithel verhindert bzw. kontrolliert ("barrier-function"). zB im Darm: Tight Junctions sind dazu gedacht, die Transport-Proteine daran zu hindern, zu ihren Transportdomänen zu kommen. Dies machen sie durch Diffusionsbarriere innerhalb des Lipid-Bilayers der Plasmamembran; sie blockieren also den Rückfluss von Glucose von der basalen Seite des Epitheliums in das Darmlumen. Tight Junctions kommen in Epithelzellen (z.B. Nieren-, Harnblasen- und Darmepithel) vor. Cell Biology SS12 (Kovarik, Martens) Seite 28 von 65 Christian Husch 30. Aufbau und Funktion von gap-junctions: gap-Junktions: In Geweben mit elektrisch erregbaren Zellen dient die Kopplung über Gap Junctions einem offensichtlichen Zweck. Einige Nervenzellen sind elektrisch verbunden, so dass Aktionspotentiale sich rasch von Zelle zu Zelle, ohne Verzögerung, die bei chemischen Synapsen auftritt, ausbreiten können. Dies ist vorteilhaft, wenn Geschwindigkeit und Zuverlässigkeit entscheidend sind, wie bei gewissen Reaktionen bei der Flucht von Fischen und Insekten. Ebenso bei den Wirbeltieren ist die elektrische Kopplung durch Gap Junctions synchronisiert, die Kontraktionen des Herzmuskels und der glatten Muskelzellen, die verantwortlich für die Peristaltik des Darms sind. Gap Junctions können auch in vielen Geweben auftreten, die keine elektrisch erregbaren Zellen besitzen. Im Prinzip bietet der Austausch von kleinen Metaboliten und Ionen einen Mechanismus für die Koordinierung der Tätigkeiten der einzelnen Zellen in einem solchen Gewebe und zum Ausgleich von zufälligen Fluktuationen in kleinen Molekül-Konzentrationen in verschiedenen Zellen. In der Leber, zum Beispiel, stimuliert die Freisetzung von Noradrenalin aus sympathischen Nervenendigungen, auf Antwort auf einen Sturz des Blutzuckerspiegels, Hepatozyten zu Glykogenabbau und Freisetzung von Glukose in das Blut zu erhöhen. Nicht alle der Hepatozyten werden jedoch von sympathischen Nerven innerviert. Mit Hilfe der Gap Junctions, die Hepatozyten verbinden, wird das Signal von den Hepatozyten zu den innervierten zu den nicht-innervierten übertragen. Mäuse mit einer Mutation im major Konnexin-Gens in der Leber, sind nicht in der Lage, Glucose zu mobilisieren, wenn der Blutzuckerspiegel fällt. Die Plasmodesmen in Pflanzen haben teilweise die gleiche Funktion wie die GAP-Junctions. 31. Verankerungen, fokale Adhäsionen, und Hemi- Desmosomen Verankerungen: Verankernde Kreuzungen treten in zwei funktionell unterschiedlichen Formen auf: 1. Adhärenzverbindung und Desmosomen halten Zellen zusammen und werden durch haftende transmembrane Proteine, die zur Cadherin-Familie gehören, gebildet. 2. Fokalen Adhäsionen und Hemidesmosomen binden Zellen an die extrazelluläre Matrix und werden durch transmembrane Adhäsionsproteine der Integrin-Familie gebildet. Auf der intrazellulären Seite der Membran, dienen Adhärenzverbindungen und fokale Adhäsionen als Verbindungsstellen für die Aktin-Filamente, während Desmosomen und Hemidesmosomen als Verbindungsstellen für Intermediärfilamente dienen. Desmosomen: Desmosomen sind knopfähnliche Punkte für interzellulären Kontakt, um Zellen zusammen zu halten. In der Zelle, sie dienen als Ankerpunkte für die strickartigen Intermediärfilamente, bilden sie einen strukturellen Rahmen von großer Zugfestigkeit. Durch Desmosomen werden die intermediären Filamente der benachbarten Zellen in einem Netz, das in den vielen Zellen eines Gewebes vorkommt, verlinkt. Die besondere Art der intermediären Filamente an den Desmosomen hängt vom Zelltyp ab: zB: es sind Keratinfilamente in den meisten epithelialen Zellen, und Desmin-Filamente in Herzmuskelzellen. Cell Biology SS12 (Kovarik, Martens) Seite 29 von 65 Christian Husch Warren/Martens: 1) Qualitätskontrolle im ER - Was unternimmt die Zelle zur Gewährleistung der richtigen Proteinfaltung? Was tut sie um falsch gefaltete Proteine wieder loszuwerden? ER - unfolded Proteins (one example)/Mechanismus, der verhindert, dass ungefaltete Proteine das ER verlassen Proteinkomplexe müssen voll zusammengebaut sein, bevor sie das ER verlassen können (z.B. Light chains und heavy chains eines Immunglobulins). Ungefaltete oder falsch gefaltete Proteine sind an Chaperone gebunden, solange bis sie die richtige Faltung haben, und können somit das ER nicht vorher verlassen. Ein ungefaltetes Protein kann nicht an seinen entsprechenden Exit Signal Rezeptorbinden und wird somit nicht exportiert. Wenn Proteine sich trotz mehreren Versuchen von Chaperonen nicht falten lassen, werden sie ins Cytoplasma retro-transloziert und dort abgebaut. ER Stress Abbau: Ungefaltete Proteine werden von einem Sensor namens IRE1 gebunden. Bei Bindung ungegefalteter Proteine dimerisiert/oligomerisiert der IRE1 Rezeptor und aktiviert dadurch seine Ribonuklease-Funktion. Somit wird ein Intron aus XBP1 mRNA herausgeschnitten, in der Folge kann der XBP1Transkriptionsfaktor translatiert werden. Im Nukleus werden dadurch Gene aktiviert, die den ungefalteten Proteinen beim Falten helfen. Sind also zu viele ungefaltete Proteine vorhanden, ist das ein Weg, um ER Stress abzubauen. (Es gibt auch Alternativen, den PERK und ATF6 Pathway.) 2. Warum benötigt der Transport in Mitochondrien ATP? Proteine, die in die Mitochondrienmatrix transportiert werden sollen, werden zunächst vollständig im Cytosol als Vorläuferproteine synthetisiert. Diese Vorläuferproteine bleiben jedoch durch Wechselwirkungen mit anderen Proteinen ungefaltet. I.d.R besitzen sie eine N-terminale Signalsequenz, die notwendig und ausreichend für einen Transport in die Mitochondrien ist (und nach dem Import von einer Signalpeptidase abgespalten wird). Die Proteintranslokation durch mitochondriale Membranen wird von Multiproteinkomplexen vermittelt, die als Proteintranslokatoren wirken: Der TOM-Komplex schafft Proteine durch die Außenmembran, die beiden TIM-Komplexe (TIM23 und TIM22 → translocase of the inner membrane) sind für die Translokation an der inneren Membran der Mitochondrien verantwortlich. Um den mitochondrialen Proteinimport anzutreiben, wird ATP Hydrolyse und ein H+ Ionengradient genutzt. Der mitochondriale Proteinimport wird an zwei voneinander getrennten Stellen durch ATPHydrolyse angetrieben: Eine Stelle befindet sich außerhalb des Mitochondriums, die andere in der Matrix. Eine weitere Energiequelle ist der elektrochemische H+ Gradient an der inneren Mitochondrienmembran. Proteine, die IN die innere Mitochondrienmembran oder in den Membranzwischenraum transportiert werden sollen, benötigen 2 verschiedene Signalsequenzen. Cell Biology SS12 (Kovarik, Martens) Seite 30 von 65 Christian Husch 3. Der Weg des LDL zum Lysosom: Bindet ein LDL (low density lipoprotein) an einen LDL Rezeptor an der Membranoberfläche, kann an der Innenseite der Membran Clathrin zusammen mit Adaptorproteinen an den Rezeptor binden. Es entsteht ein clathrin coated pit und in der Folge ein coated vesicle. Der Clathrinmantel fällt ab und das Vesikel fusioniert mit dem Endosom. Durch den niedrigeren pH im Endosom kommt es zur Dissoziation von LDL Rezeptor und LDL. Der Rezeptor wird in Transportvesikeln zurück zur Plasmamembran gebracht und ist dort wieder einsatzbereit. LDL wird im Lysosom degradiert, freies Cholesterol wird frei. (Clathrin selbst bindet nicht an die Membran oder an Transportmoleküle; das wird durch Cargo Adaptoren mediiert (z.B. Epsin1, AP2 Komplex…)) 4. SRP: was macht es und wie, Proteintransport zum Ziel (ER), SRP: SRP ist ein Ribonucleopartikel und besteht aus 5 Proteinuntereinheiten und RNA. SRP spielt eine Rolle in der cotranslationalen Translokation von Proteinen ins ER: Es erkennt die N-terminale (ER-) Signalsequenz der wachsenden Polypeptidkette im Ribosom und bindet daran. Dadurch wird die Translation gestoppt. Somit kann SRP das Ribosom an die ER Membran bringen, wo der Komplex an den SRP Rezeptor in der Membran bindet. Dort wird die Translation fortgesetzt und die Translokation der Peptidkette beginnt (Durch einen Protein Translokator, z.B. den SEC61 channel). SRP und der SRP Rezeptor dissoziieren und werden recycled. der Transport von Proteinen ins ER geschieht bereits während der Translation. → der Transportprozeß beginnt mit der spezifischen Erkennung der N-terminalen Signalsequenz (Proteine, die für den Transport ins ER bestimmt sind, besitzen am N-Terminus eine ER-Import-Sequenz) → sobald die Signalsequenz an der Oberfläche des Ribosoms erscheint, wird sie von SRP (signal recognition particle/Signalsequenzerkennungspartikel) gebunden → das gebundene SRP interagiert nicht nur mit der Singalsequenz/N-Terminus, sondern auch mit dem Ribosom (dadurch wird die Synthesegeschwindigkeit der naszierenden Polypeptidkette drastisch herabgesetzt; das SRP ist in der Lage, das Ribosom zur ER-Membran zielzuleiten, ohne dass vorher das zu transportierende Protein fertiggestellt wird) → der SRP-Rezeptor in der ER-Membran erkennt das SRP des SRP-Protein-Ribosom-Komplex (SRP bindet an den SRP-Rezeptor) + bringt das Ribosom mit der Membran in Kontakt (Ribosom interagiert mit dem Sec61-Komplex) → nachdem der SRP-Prot.-Ribosom-Komplex an die ER-Membran gebunden hat, fällt dem Signalpeptid die weitere Aufgabe zu, den ER-Translokationskanal zu öffnen → das Peptid bleibt im Kanal gebunden, während der Rest des Proteins in einer großen Schleife durch die Membran gefädelt wird (übergibt dadurch die naszierende Polypetitkette an den eigentlichen Translokationsapparat) → nach der Translokation (= Verlagerung) wird SRP mit der ER-Import-Sequenz von einer Signalpeptitase abgetrennt (befindet sich auf der luminalen Seite der ER-Membran) → unter GTP-Hydrolyse löst sich das SRP schließlich vom Ribosom ab → das Signalpeptid wird aus dem Translokationskanal verdrängt + rasch in AS abgebaut → sobald der Carboxyterminus des Prot. die Membran passiert hat, wird das Prot. ins ER-Lumen entlassen Cell Biology SS12 (Kovarik, Martens) Seite 31 von 65 Christian Husch 5. Ran GTP-GDP-Zyklus; Nuclear Pore Complex - a model explaining the selectivity / GTPase activity and Nucleus / Ran GTPase in Nuclear Transport / How is GTP hydrolysis involved into nuclear transport? Um Proteine im oder außerhab des Nucleus zu akkumulieren, muss Energie aufgewendet werden. Das geschieht durch die kleine Ran GTPase. Es herrscht ein Stufengradient von Ran-GTP im Nucleus und Ran-GDP im Cytoplasma vor. Dieser Gradient entsteht durch die asymmetrische Lokalisation von Ran-GAP (GTPase-activating protein) im Cytoplasma und Ran-GEF (guanine nucleotide exchange factor) im Nucleus. Der eigentliche Transport wird durch nuclear import receptors (→Importine) und export receptors (→Exportine) durchgeführt . Ran GTP-GDP vermittelter IMPORT: 1) Das zu importierende Makromolekül bindet an den Import Rezeptor. 2) Der Import Rezeptor ermöglicht dem Molekül die Bewegung durch die Kernpore (z.B. lokaler Abbau des FG-Hydrogels). 3) Im Nukleus sorgt Ran-GTP für das Dissoziieren des Importmoleküls und des Rezeptors. 4) Der Ran-GTP beladene Rezeptor kehrt ins Cytoplasma zurück, wo durch den Abbau von Ran-GTP zu Ran-GDP (durch GEF) Energie frei wird. An den unbeladenen Importrezeptor können nun weitere Makromoleküle binden. Ran GTP-GDP vermittelter EXPORT: 1) Das Exportmolekül kann nur an den Export Rezeptor binden, wenn Ran-GTP vorhanden ist. 2) Der Exportrezeptor ermöglicht den Transport des Moleküls durch die Kernpore ins Cytoplasma. 3) Außerhalb des Nucleus hydrolysiert GTP zu GDP durch die Interaction mit dem GEF. 4) Somit dissoziiert das exportierte Molekül vom Rezeptor. 5) Der Exportrezeptor kehr unbeladen wieder in den Nucleus zurück. Phosphorylierung Der Transport durch die Kernpore kann auch durch Phosphorylierung und Dephosphorylierung reguliert sein. 6. Die Schritte die stattfinden wenn ein Vesikel von einem in ein anderes Kompartiment geht Stages of vesicle-mediated transport Zuerst werden die zu transportierenden Moleküle an einer Stelle an der Membran angereichert. 1) Budding - durch Coat Proteine (Clathrin, COPI, COPII) und Adaptorproteine 2) Scission - Trennung des Vesikels vom Donor Kompartment (durch GTPasen wie Dynamin) 3) Uncoating (Coat fällt ab, das "nackte" Vesikel bleibt übrig) 4)Targeting & Tethering: Es gibt über 60 Mitglieder der Rab GTPasen Familie im Menschen. Diese kommen jeweils nur sehr spezifisch an diversen Kompartments der Zelle vor. Rab GTPasen sind Shuttle zwischen dem Cytosol und der Membran. In der Membran können sie sich durch eine C-terminale Isoprenyl-Modification verankern. Im Cytosol jedoch sind sie löslich, da diese Lipidmodifikation in GDI vergraben wird. (guanine nucleotide dissociation inhibitor) An die Rab-Domänen der Membran binden Kompartment-spezifische Tethering Faktoren (diese können extrem lang sein.) 5) Fusion (durch SNARE Proteine/ V-SNAREs, t-SNAREs) Cell Biology SS12 (Kovarik, Martens) Seite 32 von 65 Christian Husch 7. Wie kann man experimentell feststellen, dass eine Signalsequenz auf einem Protein sowohl notwendig als auch ausreichend für dessen Lokalisation ist? Woher weiß man, daß die erforderliche NLS-Sequenz nun auch suffizient war? Die Sequenz wird auf ein anderes Protein übertragen. Wenn Import erfolgt, war es erforderlich. 8. Snare + cell based evidence / SNARE hypothesis Die meisten intrazellulären Membranfusionen sind von SNARE Proteinen mediiert. SNARE Proteine bestehen aus mindestens einem SNARE Motif und meist noch eine C-Terminale Transmembran-Domäne (alternativ eine Struktur mit z.B. Lipidmodifikationen, die mit der Membran interagieren kann). Beispiel: Aus vier einzelnen SNARE Motiven entsteht eine stabile Struktur aus Helices, wobei die Transmembrandomänen sich mit den Membranen assoziieren. Der Komplex aus mehreren SNARE-Proteinen "zippert" dann zusammen und bringt somit die Vesikelmembran nahe an die Membran des jeweiligen Kompartments heran. Nur wenn V-SNARE Vesikel und t-SNARE Vesikel aufeinandertreffen, kommt es zu einer Fusion. Durch ihre Spezifität für den jeweiligen Fusionsprozess tragen SNAREs auch zur Kommunikation der Kompartmentidentität bei. In Nervenzellen wird die Exocytose der synaptischen Vesikel durch Ca 2+- Ionen mediiert. Dieser Vorgang beruht ebenfalls u.a. auf einem SNARE Protein, das gleichzeitig als Ca 2+- Sensor fungiert (Synaptotagmin) Die Neurotoxine von Clostridium botulinum und C.tetani sind Zn-Endoproteasen, die spezifisch bestimmte SNARE Proteine schneiden. Durch die ATPase NSF können SNARE Komplexe wieder in die Einzelbestandteile zerlegt werden. 9. Mannose and protein folding Glykosylierungen haben einen großen Einfluss auf die Struktur eines Proteins. Eine Glykosylierung kann essentiell für die richtige Faltung eines Proteins sein. Das Paradoxe dabei ist, dass der Ort der Glykosylierung dabei keine Rolle zu spielen scheint. Warum? Die Glykosylierung hilft dem falsch/ unvollständig gefalteten Protein, mit Chaperonen zu interagieren. Beispiel: Interaktion mit Calnexin Ein ungefaltetes Protein wird glykosyliert, es werden dann Glucosemoleküle abgespalten, erst dann kann das Protein mit der modifizierten Glykosylierung (die immer noch ein Glucosemolekül enthält) mit Calnexin interagieren. Cell Biology SS12 (Kovarik, Martens) Seite 33 von 65 Christian Husch 10. Bilayer – 2 Experiments suggesting it: FRAP: Fluorescence Recovery After Photobleaching Proteine können innerhalb des Bilayers lateral gleiten. FLIP: Fluorescence Loss In Photobleaching Proteine können innerhalb des Bilayers lateral gleiten. 11. Functions of the Golgi apparatus - Verteilungssystem: Im Golgi Apparat werden Proteine aus dem ER in die verschiedenen Zellkompartimente sortiert - Glycanprozessierung (Oligosaccharidmodifizierungen an Proteinen) - Remodellierung der Lipid-Doppelmembran 12. The Lysosome & lysosomal enzyme biogenesis & different transport pathways. Das Lysosom hat die Hauptaufgabe in der Degradierung von zellulärem Material. In seinem inneren herrscht ein niedrigerer pH (~5.0 im Gegensatz zu ~7.2 im Cytoplasma) und es sind viele Enzyme darin enthalten, u.a. Nucleasen, Proteasen, Lipasen, Glycosidasen etc. Durch Endocytose (oder Phagozytose) gelangt extrazelluläres Material in die Lysosomen, durch Autophagie intrazelluläres Material (z.B. auch breakdown von ganzen Mitochondrien) ER → Lysosom Die Proteine, die das Lysosom braucht, um zu funktionieren, werden vom ER über den trans-Golgi-Apparat in die Lysosomen gebracht. Das Hauptsignal für den Proteintransport ins Lsyosom über das Endosom ist Mannose-6-Phosphat, das im Golgi Apparat an das Protein gehängt wird. Lysosomale Proteine haben ein Nlinked Oligosaccharid, das an dieses Mannosephosphat binden kann. Plasmamebran → Lysosom Rab conversion reguliert die Reifung von frühen Endosomen zu späten Endosomen. 13. Welche Mechanismen gibt es, damit die Membran gebogen wird (membrane curvature)? Membranen können nur schwer gestreckt werden (ohne zu reißen), sind aber dennoch relativ biegsam. In Zellen gibt es Regionen, wo die Membran sehr viele Biegungen aufweist (sei es Plasmamebran, ER oder Golgi). Solche Wölbungen können durch das Cytoskelett entstehen (Aktinfasern und Motorproteine), aber auch einfach durch eine asymmetrische Verteilung von unterschiedlich großen Lipiden, durch asymmetrische Proteine, das Einfügen von amphiphatischen Helices oder das Assoziieren mit Scaffolds. Die BAR-Domänen in Proteinen (welche selber eine gekrümmte Form haben) können verschiedene Krümmungen in Membranen induzieren (mehr oder weniger "konvex" oder "konkav") Biegung im Zuge der Clathrin Bud formation: Epsin inseriert eine amphiphatische Helix in die Membran und unterstützt so die Biegung. BAR Domänen, z.B. in Amphiphysin stabilisieren den "Hals" des Budding Vesicle. Cell Biology SS12 (Kovarik, Martens) Seite 34 von 65 Christian Husch 14. Unterschiedliche Formen der Autophagie, Bildung des Autolysosoms Man unterscheidet zwischen a) Macroautophagie (verdauen von ganzen Organellen, Proteinaggregaten etc.) b) Microautphagie (lösliche Proteine, einzelne Organellen) c) CMA (=chaperone mediated autophagy) Funktionen der Autophagie - Autophagie kann durch Hungersignale induziert werden (Beispiel Glucagon-induzierte Autophagie in Leberzellen von Ratten; Hungerinduzierte Autophagie in Hefe) → Recycling von Nährstoffen - Schutz vor Neurodegeneration, Leberversagen (breakdown von Proteinaggregaten) - Verlangsamung des Alterns der Zelle (indem kaputte Organellen recycled werden) - Immunabwehr (Zerstörung von cytosolischen Pathogenen) Im Zusammenhang mit Krebs spielt die Autophagie eine ambivalente Rolle. Der Abbau von kaputten Mitochondrien und anderen beschädigten Zellkomponenten im Zuge der Autophagie ist positiv, denn sonst käme es zur Akkumulation von freien Radikalen und schädlichen Substanzen. Besteht jedoch bereits ein Tumor, so trägt die Autophagie zum Überleben des Tumors bei, ein Knockout von Autophagiefunktionen würde die Tumorzelle sterben lassen. Bildung des Autolysosoms Es existiert eine Vielzahl an Adaptoren, welche die Isolationsmembran an die verschiedensten Moleküle binden kann. Die Membran entsteht aus einem Reservoir bestimmter Membrankomponenten, in komplexen Eukaryoten ist noch nicht genau aufgeklärt, woher die Membran stammt, sie wird jedoch häufig parallel zum ER aufgebaut. (Atg1 Kinase Komplex und PI3K sind notwendig für die Bildung des Autophagosoms) 15. Organelle Inheritance Mitochondrien Mitochondrien können ihre DNA während des ganzen Zellzyklus, weitestgehend unabhängig von dem Zellzyklusstatus bzw. der Replikation der genomischen DNA replizieren. Sie können sich spalten (fission) oder miteinander verschmelzen (fusion). Die Fusion wird u.a. von GTPasen mediiert. Je nach Spezies können sie bei der Zellteilung zufällig oder geordnet auf die Tochterzellen aufgeteilt werden. Vakuole In Hefe sorgen Proteinkomplexe an einem Aktin-Gerüst dafür, dass Segregationsstrukturen der Vakuole auch in die Tochterzelle gelangen. ER Auch für die Positionierung des ER Gerüsts während der Zellteilung sind Aktinfasern & Motorproteine zuständig. Während der Metaphase wird das Gleichgewicht zwischen ER sheets und tubules in Richtung tubules verschoben. Golgi Während der Mitose wird die Interphase-Golgi Struktur in der Nähe des Kerns aufgelöst - es entstehen kleine mitotische Golgi Fragmente (in Form kleiner Vesikel), die sich gleichmäßig in der Zelle verteilen. In der Telophase beginnt der Golgi-Apparat wieder zu assemblieren. Auf der Golgi Membran befinden sich viele Thethering factors, durch welche die Golgi Vesikel nahe an der Membran bleiben. 16. Wie kann man biochemisch Feststellen, daß ein Protein in ein bestimmtes Kompartiment transportiert wird? Es wird ein markiertes Protein, das eine spezifische Signalsequenz trägt, in isolierte Organellen in vitro transportiert. Das markierte Protein wird üblicherweise durch zellfreie Translation einer gereinigten mRNA, welche das Protein codiert, produziert; Radioaktive Aminosäuren werden verwendet, um das neu synthetisierte Protein zu markieren, so dass es von den vielen anderen Proteinen, die im in vitro-Translationssystem sind, unterschieden werden kann . Drei Methoden werden zum Nachweis gemacht: 1.) Das markierte Protein co-fraktioniert mit der Organelle während der Zentrifugation 2.) Die Signalsequenz wird durch eine spezifische Protease entfernt, die innerhalb der Organelle ist. 3.) Das Protein ist vor Verdauung geschützt, wenn Proteasen zum Inkubationsmedium hinzugefügt werden, aber ist anfällig, wenn ein Detergent als erstes hinzugefügt wird, das die Organellmembran zerstört. Durch die Nutzung solcher in vitro-Assays, kann man feststellen, welche Komponenten (Proteine, ATP, GTP, etc.) für den Translokations-Prozess erforderlich sind. Cell Biology SS12 (Kovarik, Martens) Seite 35 von 65 Christian Husch 17. Transfektionsansatz für Definierung von Signalsequenzen: A Transfection Approach for Defining Signal Sequences Ein Weg, um zu zeigen, daß eine Signalsequenz benötigt und suffizient ist, um ein Protein in ein gewisses Kompartment zu transportieren ist, ein Fusionsprotein zu kreieren, in welchem die Signalsequenz durch Gentechnik an ein Protein angeheftet wurde, welches sich normalerweise im Cytosol befindet. Nachdem die cDNA das Protein enkodiert und Zellen damit „infiziert“ hat, kann durch Immunfärbung oder Zellfraktionierung das Protein nachgewiesen werden. Durch Verändern der Signalsequenz mit gerichteter Mutagenese, können wir bestimmen, welche strukturellen Features wichtig für ihre Funktion sind. Cell Biology SS12 (Kovarik, Martens) Seite 36 von 65 Christian Husch Martens – Foliensatz 1 Langmuir through Durch Nutzung eines Langmuir throug können amphiphile Moleküle, die sich an der Wasseroberfläche (WasserLuft-Grenze) angesammelt haben, vermessen werden. Phopholipide können aber nicht die Seite wechseln (flip-flop)! Proteine können auf verschiedene Arten in Kontakt mit Membranen treten: Die Länge der Transmembrandomäne der Proteine ist bestimmend für die Lokalisation. Da die Plasmamembran dicker ist als die ER- und Golgi-Membran, sind Proteine mit mehr Transmembrandomänen eher dort zu finden. Periphere Membranproteine können nur vorübergehend mit der Membran interagieren. Cell Biology SS12 (Kovarik, Martens) Seite 37 von 65 Christian Husch FRAP: Fluorescence Recovery After Photobleaching Proteine können innerhalb des Bilayers lateral gleiten. FLIP: Fluorescence Loss In Photobleaching Proteine können innerhalb des Bilayers lateral gleiten. TIRF-M: Total Internal Reflection Fluorescence Microscopy Nur Moleküle im flüchtigen Feld fluoreszieren! Proteine können innerhalb des Bilayers lateral gleiten. Dieses laterale Gleiten von Proteinen ist wichtig für die Signalweiterleitung in Membranen! Weiters erlaubt es Rezeptoren zu Dimerisieren, nachdem es ein Signalmolekül gebunden hat. Cell Biology SS12 (Kovarik, Martens) Seite 38 von 65 Christian Husch Detergenzien (Reinigungsmittel) können Lipide ersetzen und dadurch Membranproteine auflösen. Detergenzien können auch wieder entfernt werden um gereinigte Membranproteine in Vesikel einzusetzen. - lösliches Membranprotein mit Detergenz - gereinigtes, lösliches Membranprotein - wässrige Detergenzien unterhalb des CMC bei Anwesenheit von Lipiden - Das Protein ist nun in Vesikel eingeschlossen Problem: Das Membranprotein hat seine Asymmetrie verloren. BAR-Domänen können verschiedene Membran-Kurvaturen induzieren. Wichtige Messages: - Die Lipid-Doppelmembran besteht aus amphiphilen Lipiden (polarer Kopf, apolarer Schwanz) - Lipide nutzen 2 Acyl-Ketten (meist 16 oder 18 C-Atome lang) - Eine Acyl-Kette ist normalerweise ungesättigt - Einzelne Lipide können innerhalb der Doppelmembran rotieren und wandern - Einzelne Lipide können nur sehr selten zwischen den Einzelmembranen springen (flip-flop) - Membranen sind asymmetrisch (Lipide als auch Proteine) - Die Doppelmembran hat den hydrophoben Kern nicht nach außen gekehrt (Membranen sind dadurch verschlossen) - Membranen sind schwer zu dehnen, aber leicht zu krümmen - Cholesterol versteift die Membran bei hohen Temperaturen, aber macht sie flüssiger bei niedrigen Temperaturen - Proteine können innerhalb der Membran lateral diffundieren. Diese Fähigkeit ist zB wichtig für die Signaltransduktion. - Membranproteine können mit Detergenzien löslich gemacht werden - Membranen können mit zellulären Proteinen gekrümmt werden. Cell Biology SS12 (Kovarik, Martens) Seite 39 von 65 Christian Husch Martens – Foliensatz 2: Kerntransport: Zellen sind hochgradig gegliedert. Der Hauptvorteil dieser Gliederung ist die Fähigkeit, verschiedene biochemische Reaktionen voneinander zu trennen (zB niedriger pH und Proteasen in Lysosomen) Jedoch besteht das Problem, daß Proteine und andere Substanzen in die für sie bestimmten Bereiche transportiert werden müssen. Die Lösung ist ein hochkomplexes und geregeltes Zielsystem. Signalsequenzen: Dieses Transportsystem ist an das Vorhandensein von Signalsequenzen gebunden und erforderlich, um Proteine an den Bestimmungsort zu bringen. Der Nuclear pore-Complex: - 8fach symmetrisch gefalten - Molekulargewicht ungefähr 1,25 x 105 kDa - besteht aus ca. 30 verschiedenen Proteinen, manche in mehrfacher Ausführung - Eine durchschnittliche Säugetierzelle hat 2 – 4000 Kernporen/Kern - Erlaubt Transport von mehr als 500 Makromoleküle/Sekunde in beide Richtungen - In unserem Körper wird jede Minute ungefähr ein Kilo Ladung durch Kernporen geschleust - Hochselektiv - Proteine, die größer als 40 – 60 kDa sind, können frei diffundieren - Kernporenkomplexe können große Partikel transportieren (zB Ribosomen; diese werden im Kern zusammengesetzt und durch den Kernporenkomplex ins Cytoplasma transportiert) Nuclear Localisation Signal (NLS) Woher weiß man, daß die erforderliche NLS-Sequenz nun auch suffizient war? Die Sequenz wird auf ein anderes Protein übertragen. Wenn Import erfolgt, war es erforderlich. FG repeats (F = Phenylalanin, G = Glycin): Multiple FG repeats sind in der Kernpore lokalisiert. Diese FG repeats haben sehr spezielle Eigenschaften: eigensicher entfaltet spielen eine untergeordnete aber wichtige Rolle beim selektiven Transport Interagieren miteinander Interagieren mit Kerntransport-Rezeptoren Cell Biology SS12 (Kovarik, Martens) Seite 40 von 65 Christian Husch Versuch mit Hydrogel: Eine Kernpore ist gefüllt mit einem Hydrogel, daß aus FG repeats besteht. Dieses Hydrogel formt eine Matrix, die nur kleine Partikel durch die Pore lässt. Aber warum erleichtert die Bindung von Kernimport-Rezeptoren den Transport, obwohl die Größe des Partikels durch die Rezeptoren noch mehr wächst? Die Antwort ist, daß der Kernimport-Rezeptor mit den FG repeats interagiert und dadurch lokal die Struktur des Hydrogels aufgelöst wird. Ran GTP-GDP-Zyklus betreibt Kernimport und –export: Um Makromoleküle sowohl innerhalb als auch außerhalb des Kerns zu konzentrieren, ist Energie erforderlich. Dafür sorgt die Ran GTPase. Dies ist ein Stufengradient von Ran-GTP im Kern und Ran-GDP im Cytoplasma. Dieser Gradient wird durch die asymmetrische Lokalisation von Ran-GAP im Cytoplasma und Ran-GEF im Kern gebildet. Kernimport-Rezeptoren (Importine) und Kernexport-Rezeptoren (Exportine) verknüpfen den Ran-GTP/GDP-Zyklus für den Import und Export. Import: Fracht bindet Import-Rezeptoren Der Import-Rezeptor ermöglicht Transport durch die Kernpore. Im Kern löst Ran-GTP die Fracht vom Rezeptor. Export: Fracht bindet bei Anwesenheit von RanGTP an Exportrezeptor. Der Export-Rezeptor ermöglicht Transport durch Kernpore. Außerhalb des Kerns hydrolysiert Ran GTP zu GDP nach dessen Interaktion mit GEF. Cell Biology SS12 (Kovarik, Martens) Seite 41 von 65 Christian Husch Die Fracht löst sich vom Rezeptor. Lösen der Fracht vom Rezeptor mit Ran-GTP Transport von Fracht durch die Pore wird reguliert durch de-Phosphorylierung und Phosphorylierung Transport in Mitochondrien: Funktionen von mitochondrialen Proteinen: Energiestoffwechsel Stoffwechsel der Aminosäuren, Lipide, des Eisens und des Häms Transportsystem für Metaboliten Proteintransport und –faltung Proteinabbau Signalprozesse Membranumbau, -fusion, -spaltung Involviert beim programmierten Zelltod Die meisten mitochondrialen Proteine werden zuerst im Cytosol synthetisiert und dann post-translational ins Mitochondrium transportiert. Das Signal für den Import in die Mitochondrien besteht aus einer N-terminal amphipathischen Helix, welche positiv geladene Aminosäuren auf der einen Seite und hydrophobe Aminosäuren auf der anderen Seite enthält. Transportweg: Matrixproteine passieren beide Membranen auf einmal. Der N-terminus passiert zuerst. N-terminales Peptid ist vom Protein getrennt, weil die Peptidase in der Matrix lokalisiert ist. Das Protein überquert daher beide Membranen auf einmal. Cell Biology SS12 (Kovarik, Martens) Seite 42 von 65 Christian Husch Mitochondrien wurden isoliert und unter unterschiedlichsten Bedingungen mit einem Precursor-Protein inkubiert. Dies erlaubte eine detaillierte Analyse des mitochondrialen Imports. Import-Export-Komplexe: Proteintransport in äußere Membran: Proteintransport in innere Membran: Proteinimport in die Matrix: Proteinimport in innere Membran und Membranzwischenraum: Proteintransport in innere Membran: Proteinimport benötigt Energie: Cell Biology SS12 (Kovarik, Martens) Seite 43 von 65 Christian Husch Proteinimport in Peroxisomen: Einzelne Membran Alle Proteine werden im Kern kodiert In allen Zellen in unterschiedlicher Anzahl vorhanden Steuert Beta-Oxidation und Detoxifikation Katalysieren die erste Reaktion bei der Synthese von Plasmalogenen, welche das am häufigsten vorkommende Lipid im Myelin sind Können sich durch Teilung vermehren, aber auch ganz neu entstehen Das Signal für den Import in Peroxisomen ist ein C-terminales SKL-Motiv Entwicklung von Peroxisomen: PTS1 ist ein Protein, daß im Cytoplasma produziert wird und dann zu den Peroxisomen transportiert wird. Aber warum wird PTS nicht ins ER-Lumen transportiert? Die Lösung ist, daß die Import-Pore des Peroxisoms nur in der Peroxisom-Membran vorkommt. Während der Neubildung fusionieren 2 verschiedene Klassen von ER-abgeleiteten Vesicel. Nur nach der Fusion kann ein neu zusammengesetztes Translocon den Transport von Proteinen in das neue Peroxisom bewerkstelligen. Später können Peroxisomen sich durch Teilung vermehren. Import in Peroxisomen: Peroxismale Proteine werden alle gleichzeitig durch eine Pore transportiert. Daher muß diese Pore einen großen Durchmesser haben, weil transportierte Proteine sich nicht entfalten müssen, um importiert zu werden. SRP (Signal Recognition Particle) SRP erkennt die N-terminale Signalsequenz, bindet daran und hält die Translation an. Dies erlaubt SRP, die Ribosomen zur ER-Membran zu bringen. Cell Biology SS12 (Kovarik, Martens) Seite 44 von 65 Christian Husch Martens – Foliensatz 3: SRP und Ribosomen-Zyklus: Der Translokationsapparat: Ein Verschluß hindert kleine Moleküle daran, daß sie aus dem ER austreten oder ins ER eintreten, wenn der Kanal keine Translokation durchführt. Während der Translokation füllen ungefaltete Polypeptide den Kanal und verhindern ein passieren. Erste Schritte der Protein-Translokation: Transport von löslichen Proteinen: Cell Biology SS12 (Kovarik, Martens) Seite 45 von 65 Christian Husch Translokation von einfach umspannenden Membran-Proteinen: Translokation von zweifach umspannenden Membran-Proteinen: Glycosylierung: Glycosylierung hat einen großen Einfluß auf die Struktur des Proteins! Paradox: Manche Proteine benötigen Glycosylierung, um sich richtig zu falten. Jedoch der Ort der Glycosylierung dürfte nicht egal sein. Zudem, Glukoseteile werden zuerst entfernt vom Protein und nachher wieder angefügt? Warum? Antwort: Der scheinbar nutzlose Zyklus hilft falsch gefalteten Proteinen, mit Chaperon-Proteinen zu interagieren. Dabei bindet Glycosyl-Transferase an hydrophobe Bereiche auf der Oberfläche von falsch gefalteten Proteinen. Proteinfaltung: Cell Biology SS12 (Kovarik, Martens) Seite 46 von 65 Christian Husch Proteinfaltung in vivo: In vitro werden für die Proteinfaltung Stunden oder Tage benötigt In vivo dauert es nur Minuten 5 – 10 % von allen E. coli-Proteinen falten sich zu einem beliebigen Zeitpunkt Diese gefalteten Proteine sind gefährlich für die Zelle, weil sie zu Aggregation neigen Bei Konzentrationen in vitro, die in Zellen gefunden wurden, waren die Proteine komplett aggregiert. Daher haben Zellen spezielle Proteine, genannt Chaperone, die helfen, das Protein zu falten. Was passiert, wenn ungefaltete Proteine im ER akkumulieren (ER stress)? Ungefaltete Proteine binden an einen Sensor, IRE1 Nach der Bindung an das ungefaltete Protein, dimerisiert und event. oligomerisiert IRE1. Oligomerisation aktiviert eine Ribonuklease-Domäne, die ein Intron von einer mRNA spaltet, die den Transkriptionsfaktor XBP1 codiert Nachdem das Intron entfernt wurde verbindet die tRNA-Ligase die mRNA-Enden wieder und kann mit der Transkription von XBP1 beginnen. Im Nucleus aktiviert XBP1 die Transkription von Genen, die helfen, das ungefaltete Protein zu falten und das ER zu erweitern. Nur korrekt gefaltete Proteine können das ER verlassen! Wenn Proteine nach einigen Zyklen noch immer nicht gefaltet sind und nicht an die am ER ansässigen Chaperone binden, werden sie abgebaut. Dies passiert, nachdem sie zurück ins Cytosol transportiert wurden. Vesikeltransport: Transport von Proteinen, Lipiden und anderer Fracht zwischen verschiedenen Abschnitten und außerhalb der Zelle wird im Wesentlichen durch kleine Transport-Carrier bewerkstelligt, welche zusammengefasst als Transportvesikel bezeichnet werden. Die Hauptschritte des Vesikeltransports: Fracht wird an einem Teil der Membran angereichert/angelagert. An diesem Membranteil geschieht die Knospung. Das Vesikel trennt sich durch einen Schneidevorgang von der Membran Die Coat-Proteine (Ummantelung) fallen ab Das Transportvesikel steuert das Ziel an und binden dann an den Ziel-Abschnitt Das Vesikel wird in die unmittelbare Nähe der Zielmembran gebracht (Andocken) Das Vesikel fusioniert mit dem Zielabschnitt Substratkonzentration und Knospung: Coat-Proteine und Adaptorproteine Schneiden: GTPase (zB Dynamin) Ansteuerung und Anbindung: rab-Proteine und Tethering-Proteine Cell Biology SS12 (Kovarik, Martens) Seite 47 von 65 Christian Husch Fusion: SNARE-Proteine Wie wurde der Mechanismus entdeckt? 1. Es wurden Mutanten generiert, welche einen Defekt in einem gewissen Schritt der Membranpassage zeigen. 2. Dann wird eine Bibliothek von beliebig rekombinanten DNA-Molekülen angefertigt und in die Zellen, welche den Defekt in der Membranpassage aufweisen, transformiert. 3. Dann werden Zellen selektiert, welche nicht den Defekt aufweisen, die rekombinante DNA wird isoliert und die eingefügten Gene durch Sequenzierung identifiziert. Martens – Foliensatz 4: Molekulare Maschinerie für Vesikeltransport – Coat (= Mantel)-Proteine: Es gibt verschiedene Typen von Mantel-Proteinen, welche in unterschiedlichen Transportschritten wirken. Diese wurden bisher gut verstanden: - COPII coat, welches Funktionen beim Transport vom ER zum Golgi hat - COPI coat, welches Funktionen innerhalb des Golgi und Transportfunktionen vom Golgi zum ER hat - Clathrin coat, welches Funktionen beim Transport vom trans-Golgi zur Plasmamembran und bei der Endocytose hat Clathrin selbst bindet nicht an die Fracht oder Membranen. Dies wird durch Cargo-Adapter bewerkstelligt. Darüber hinaus gibt es mehrere andere Hüllproteine und Komplexe, die weniger gut verstanden oder noch zu entdecken sind. Endocytose von LDL: Ist eine Endocytose, welche vermittelt wird durch clathrin-coated Vesikel: Niedriger pH im Endosom führt zur Dissoziation des Liganden vom Rezeptor. Transferrin-Transport: Eine Variation des rechten Schemas betrifft die Endocytose von Transferrin und seines Rezeptors. Hier dissoziiert nur der Ligand vom Transferrin. Transferrin selbst bleibt an seinen Rezeptor gebunden und wird zur Plasmamembran zurückgeführt, wo es vom Rezeptor dissoziiert. Andere Rezeptoren wie der EGFP-Rezeptor werden in Lysosomen abgebaut. Cell Biology SS12 (Kovarik, Martens) Seite 48 von 65 Christian Husch Vesikelbildung, Trennung und Transport: AP2-Adaptor: PI(4,5)P2 ist ein Teil der Familie der phosphorylierten Phosphatidylinositol-Lipide. Diese gering vorkommenden Lipide spielen eine wichtige Rolle während des Vesikeltransports, weil sie eine sehr spezifische Bindestelle für periphere Membran-Proteine, wie AP2, Epsin oder AP180 haben. Da verschiedene Formen von phosphorylierten Phosphatidylinositol-Lipiden bevorzugt an verschiedenen Kompartimenten lokalisiert sind, tragen sie auch zur Organell-Identität bei. Dies ist aufgrund ihrer Fähigkeit, verschiedene Proteine zu den verschiedenen Kompartimenten zu bringen, möglich. zB: PI(3)P: Endosomen PI(4)P: Golgi-Apparat PI(4,5)P: Plasmamembran PI(3,4,5)P: Plamamembran PI(3,5)P: späte Endosomen, Multivesikuläre Körper AP2 bindet an die Ladung nur in Gegenwart von PI(4,5)P 2 an der Membran. Diese Art der Interaktion wird auch als Co-inzidenz-Erkennung bezeichnet. Das Konzept der Co-Inzidenz-Erkennung spielt eine sehr wichtige Rolle in der Zellbiologie und sorgt dafür, daß Interaktionen nur dann passieren, wenn sie wirklich gebraucht werden. Die Co-Inzidenz-Erkennung von Frachtmolekülen und PI(4,5)P 2 an der Plasmamembran führt zur Bindung der Adaptoren an der Membran und anschließender Clathrin-Rekrutierung. - - Die kombinierte Wirkung von Clathrin-Mantel und manchen Adaptoren (zB Epsin) führt zur Knospung der Clathrin-bedeckten Membranstelle. Epsin inseriert eine amphipatische Helix in die Membran und hilft so Clathrin während der Knospenbildung. BAR-domain-Proteine (zB Amphiphysin) stabilisieren die Einschnürung des ausknospenden Vesikel. Zum Schluß formt sich ein Vesikel. Nachdem sich der Vesikel von der Plasmamembran getrennt hat, löst sich der Clathrin-Mantel und fällt ab. Dynamin formt eine Helix um die Vesikel-Einschnürung und bekommt einen „Kraftschlag“. Dadurch drückt sich die Membraneinschnürung zusammen. Dieses Zusammendrücken vermittelt das Abschneiden des Vesikel von der Membran. Die Energie für diese Reaktion stammt von der Hydrolyse von GTP zu GDP durch Dynamin. Nachdem der Vesikel von der Plasmamembran gelöst ist, löst sich der Clathrin-Mantel durch die Aktionen von Auxilin und hsc70. Cell Biology SS12 (Kovarik, Martens) Seite 49 von 65 Christian Husch Tethering (Anbindung) und Fusion eines Vesikel mit seinem Ziel: Rab GTPase: Es gibt über 60 Rab GTPase Familienmitglieder, welche im menschlichen Genom kodiert sind. Einzelne Mitglieder dieser Familie zeigen sehr spezifische Lokalisierungen auf verschiedenen Kompartimenten der Zelle. Rab GTPasen verkehren zwischen dem Cytosol und der Membran. Diese können mit der Membran assoziieren, weil sie eine C-terminale Isoprenyl-Modifikation transportieren. Rab GTPasen werden im Cytosol löslich gehalten, weil ihre Lipid-Modifikation eingebettet ist, in einem Protein namens Guanin nucleotid dissociation inhibitor (GDI). Wenn auf der Membran und im GTP Rab GTPasen eingebunden sind, interagieren sie mit einer Vielzahl von Effektor-Molekülen. Zwischen diesen Effektor-Molekülen befinden sich spezifische Lipid-Kinasen, welche die Produktion von phophorylierten Phosphatidylinositol katalysieren. Beim frühen Endosom ist Rab5 diese Rab GTPase, welche PI3-Kinase rekrutiert, um zur Generation der PI(3)P zu werden. PI(3)P führt wiederrum zur Rekrutierung von bestimmten Effektoren, welche Rab5 und PI(3)P binden. Es wird angenommen, daß dies zur Bildung einer Formation führt, die Rab-Domäne genannt wird. Experiment zur Identifizierung von Rab5-bindenden Partnern über Affinitäts-Chromatographie: Rab5 wurde auf einer Matrix fixiert und mit Cytosol aus einem Rinderhirn wurde darübergegeben. Ungebundene und unspezifische Proteine wurden abgewaschen. Die bindenden Proteine wurden eluiert (herausgelöst) und durch SDS-Page analysiert. Die Proteine in den einzelnen Bändern wurden durch Massenspektrometrie identifiziert. Tethering-Faktoren fangen spezifische Vesikel ein, welche für die Fusion mit dem jeweiligen Kompartiment bestimmt sind. Nachdem der Vesikel gebunden hat, fusioniert er mit der Zielmembran. Die meisten intrazellulären Membranfusionen werden gesteuert durch SNARE-Proteine. Studien bei Hefe- und Säugetierzellen haben gezeigt, daß verschiedene SNARE-Proteine vorhanden sind und verschiedene Schritte benötigen, um intrazellulären Transportschritte durchzuführen. Alle SNARE-Proteine enthalten mindestens ein SNARE-Motiv. Manche SNAREs haben also eine C-terminale Transmembran-Domäne, welche unmittelbar der SNAREDomäne folgt. Die transmembrane-enthaltenden SNARE-Proteine sind Schwanz-verankernde Proteine, die zuerst im Cytosol produziert werden und dann posttranslational in das ER eingefügt werden. Manche SNARE-Proteine enthalten keine Transmembran-Domäne und interagieren mit der Membran mit anderen Mitteln (zB Lipid-Modifikationen). Struktur des SNARE-Komplexes: Vier einzelne SNARE-Motive fügen sich zur einem sehr stabilen 4-helikalen Bündel zusammen. (rote Box). Die Helices der Transmembrandomäne beinhalten SNARE-Ausläufer, die sich in die Membran erstrecken. Mindestens eine Transmembrandomäne mit SNARE muß in jedem Kompartiment zur Fusion vorhanden sein. Cell Biology SS12 (Kovarik, Martens) Seite 50 von 65 Christian Husch Die Fusionsreaktion: Zuerst müssen die Membranen in Kontakt kommen. Als nächstes fusionieren die gegenüberliegenden einfachen Membranen zu einem halbfusionierten Stiel. Zu diesem Zeitpunkt hat noch keine Vermischung des Inhalts stattgefunden. Als nächstes fusionieren die beiden distalen Einzelmembranen und eine Fusionspore wird gebildet. Nun vermischen sich die Inhalte der beiden Kompartimente. Beweis für SNARE-vermittelte Membranfusion: Die SNAREs werden zuerst gereinigt und dann in das Liposom integriert. Ca++-abhängige synaptische Vesikel-Exocytose: Ist abhängig von SNARE-Protein, dem Ca++-sensor-Synaptotagmin (und anderen Proteinen). Beweis dafür, daß SNAREs während der Membranfusion benötig werden – in vivo: - TeNT (Tetanus-Neuro-Toxin) - BoNT (Botulinum-Neuro-Toxin) SNARES sind Ziel für verschiedene Neurotoxine: Clostridium botulinum: Sekretiert Botulinum Neurotoxin, Typen A – G, (BoNT/A-G) Clostridium tetani: Entdeckt als Spore im Schmutz oder als Parasit im Gastrointestinaltrakt von Tieren. Produziert ein starkes biologisches Toxin, Tetanospasmin (Tetanuxtoxin). Das Toxin ist der Auslöser von Tetanus, einer Krankheit, die charakterisiert ist durch schmerzhafte, oft fatale, Muskelkrämpfe. Diese Toxine sind Zn-Endoproteasen, jede spezifisch für ein bestimmtes SNARE-Protein. Ratten und Hühner sind resistent gegenüber bestimmten Toxinen wegen einer Aminosäure-Substitution in der Spaltstelle. Cell Biology SS12 (Kovarik, Martens) Seite 51 von 65 Christian Husch SNARE-Komplexe werden durch die große ATPase NSF zerlegt. Infektion mit einem Influenza-Virus: So wie die SNARE-Proteine bilden auch virale Fusionsproteine hairpin-ähnliche Strukturen, welche die beiden Membranen in unmittelbare Nähe zueinander bringen. Martens – Foliensatz 5: Membrantransportwege: Über das Golgi und darüber hinaus! Die kleine GTPase Sar1 wird aktiviert durch Sar1-GEF, welche an der ER-Ausgangsseite lokalisiert ist. Die Aktivierung geschieht beim Wechsel von GDP zu GTP und führt zu einer Exposition einer amphipatischen Helix, welche in die Membran inseriert und somit Sar1 membrangebunden macht. Sar1 gehört zur Gruppe der Arf-Familie der GTPasen, welche alle nach einem ähnlichen Prinzip funktionieren. Arf-familien GTPasen sind gelöst, wenn sie an GDP binden. In der GTP-gebundenen Form zeigt sich eine N-terminal hydrophobe Region, welche erlaubt, sich an Membranen zu binden, wo sie andere Proteine rekrutieren. Sar1-GTP rekrutiert 2 Komponenten des COPII-Mantels, Sec24 und Sec23. Sec24 bindet an das Frachtprotein und diese Fracht wird in der ausknospenden Membran angereichert. 2 weitere componenten des COPII-Mantels, Sec13 und Sec31, verbinden sich und helfen nach der Knospung des Vesikel. Cell Biology SS12 (Kovarik, Martens) Seite 52 von 65 Christian Husch Sar1-GTP und COPII vermitteln Knospung (budding) und Abtrennung der Vesikel in vitro! Die Größe des COPII-Vesikel kann durch Ubiquitination von SEC31 reguliert werden, um Platz zu machen für große Fracht-Moleküle wie Collagen. Sec23 bindet nicht nur an Sar1, sondern kann auch als GAP (aktiviert GTP-hydrolyse) fungieren. Es wirkt also wie eine Zeitschaltuhr. Wenn die Knospung aufgrund fehlender oder zu geringer Ladung zu langsam verläuft, fällt der Mantel von der Membran, bevor sich das Vesikel formen kann. Nachdem die Fracht das ER in COPII-ummantelten Vesikel verlassen hat, erreicht es das zwischen ER und Golgi liegende Kompartiment. Hier werden ERProteine, die aus dem ER entwichen sind, in COPI-ummantelten Vesikel zum ER rücktransferiert. ARF-GAP1 vermittelt Membrankrümmung: Arf-GAP bindet an die Membranen in einer krümmungsabhängigen Weise. Krümmungsvermittlung durch ARF-GAP stimuliert GTP-hydrolyse durch ARF nur während oder nach der Knospung. Wie wird verhindert, daß ER-Proteine dem ER verloren gehen? Dies verhindert, daß sie in die COPII-Knospen kommen. Sterische Hinderung: Da ist kein Platz in der Knopse, wenn das Cell Biology SS12 (Kovarik, Martens) Verlorene ER-Proteine werden durch eine C-terminales –KDEL-Sequenz geborgen, Seite 53 von 65 Christian Husch sekretorische Protein eintritt. Funktionen des Golgi-Apparat: - welche durch –KDEL-Rezeptor erkannt wird. Sortierung der Proteine, die vom ER kommen und Verteilung auf die Kompartimente Verarbeitung von Glykanen Umbau der Lipid-Doppelmembran Wie kann die Fracht durch den Golgi transportiert werden, während die modifizierenden Enzyme bleiben wo sie sind? Die Tatsache, dass die Fracht in Vesikel verpackt werden kann, unterstützt das vesikuläre Transport-Modell. COPI-Vesikel transportieren rückgerichtete Fracht. Der Alpha-Faktor-Rezeptor (Ste2) wird benötigt, um MATa-Zellen mit MAT-Alpha-Zellen zusammen zu fügen. Ein anhaftendes –KK-Motiv verhindert die Zusammenfügung. In S. cerevisiae (Bäckerhefe) sind die einzelnen Zisternen des Golgi-Apparates nicht gestapelt, sondern verteilt über das Cytoplasma. Daher können die einzelnen Zisternen durch Lichtmikroskopie aufgelöst werden. Lysosomen verrichten den Großteil des Abbaus von Zellmaterial. Endozytose und Phagozytose verrichten die Lieferung von extrazellulärem Material zu Lysosomen, während Autophagie intrazelluläres Material zu Lysosomen liefert. Der Weg von der Plasmamembran zum Lysosom. Damit das Lysosom funktioniert, müssen Proteine, die im ER synthetisiert werden, über den trans-Golgi-Apparat zu den Lysosomen verschickt werden. (Satz auf Englisch: In order for lysosomes to function proteins synthesized in the ER have to be delivered to lysosomes via the trans-Golgi apparatus.) Das Hauptsignal für den Proteintransport in die Lysosomen via Endosomen ist Mannose-6-Phosphat, welches am N-linked Oligosaccharid von lysosomalen Proteinen angebracht ist. Cell Biology SS12 (Kovarik, Martens) Seite 54 von 65 Christian Husch Rezeptor-Recycling: Das Retromer ist der Mantel für den röhrenförmigen Transporter, welcher den Mannose-6-Phosphat-Rezeptor zurück zum trans-Golgi-Netzwerk bringt. Martens – Foliensatz 6: Aufbau des Golgi-Apparats: Wenn auf der Membran und im GTPgebundenen Zustand, interagieren Rab GTPasen mit einer Vielzahl von Effektor-Molekülen. Unter diesen Effektormolekülen sind spezifische Lipid-Kinasen, welche die Produktion von phosphorylierten Phosphatidylinositol katalysieren. Im frühen Endosom ist diese Rab GTPase Rab5, welche PI 3-Kinase rekrutiert, was zur Erzeugung von PI(3)P führt. PI(3)P wiederrum führt zur Rekrutierung von bestimmten Effektoren, welche Rab5 und Pi(3)P binden. Es wird angenommen, daß dies zur Bildung von sogenannten Rab-Domänen führt. Rab-Konversion reguliert die Reifung von frühen zu späten Endosomen. Rab5 rekrutiert ein GEF für Rab7 an die Membran. Cell Biology SS12 (Kovarik, Martens) Seite 55 von 65 Christian Husch Manche extrazellulären Rezeptoren, wie zB der EGF-Rezeptor werden in Lysosomen abgebaut. Das Signal für den Abbau ist ein Ubiquitin-Protein, daß am Rezeptor angefügt wird. Epsin und Eps15 sind Adaptorproteine an der Plasmamembran, welche ubiquitinierte Rezeptoren erkennen. Der ESCRT-Komplex vermittelt die Verpackung der Fracht in die Vesikel und die Abschnürung der Vesikel. Die Rekonstitution von ESCRT-vermittelter Vesikel-Einschnürung und –Abschnürung benötigt große unilamellare Vesikel und gereinigte Proteine. Zusammenfassung des Ubiquitin-abhängigen Abbaus von Rezeptoren im Körper und multivesikulären Lysosomen. Manche professionellen Phagozyten, wie die Macrophagen, sind in der Lage, große Partikel aufzunehmen. Das Phagosom fusioniert dann mit dem Endosom und dem Lysosom. Pathogene Bakterien, wie Salmonella und Listeria, induzieren aktiv ihre phagozytische Verschlingung (Phagocytose), um Zugang auf Wirtszellen-Cytoplasma zu bekommen. Salmonella benutzt dabei einen Trigger-Mechanismus, Listeria einen Zipper-Mechanismus. Der Transport von Virulenzfaktoren in die Wirtszelle geschieht über den Typ3-Sekretionsapparat. Das Mycobacterium tuberculosis verhindert die Fusion von Phagosomen und Lysosomen. Cell Biology SS12 (Kovarik, Martens) Seite 56 von 65 Christian Husch Autophagie: Autophagie ist der Hauptweg für den Vesikel-Abbau von intrazellulärem Material. Die verschiedenen Formen der Autophagie: Schritte der Autophagosom-Bildung: Funktionen der Autophagie: Hungerzustände Recycling von Nährstoffen Proteinaggregate Schutz vor Neurodegeneration, Leberschäden, …… Beschädigte Organellen Verzögerung der Alterung Cytosolische Pathogene Immunantwort Autophagie ist essentiell für das Überleben der Mäuse während des neonatalen Hungerzustands. Bei Mäusen formen sich kurz nach der Geburt Autophagosomen. Diese helfen der Maus, in den ersten Tagen nach der Geburt zu überleben, bis sie genug Milch von der Mutter bekommen können. Suppression der Autophagie in neuronalen Zellen führt zur spontanen Neurodegeneration. Cell Biology SS12 (Kovarik, Martens) Seite 57 von 65 Christian Husch Autophagie und Krebs – ein doppelschneidiges Schwert: Bevor sich der Tumor formiert: Autophagie Reinigung von defekten Mitochondrien und anderen gefährlichen Komponenten Tumorsuppression Autophagie Tumorfördernd Akkumulierung von reaktiven Sauerstoffarten und anderen gefährlichen Komponenten Nachdem sich der Tumor formierte: Autophagie Tumor überlebt Autophagie Tumor überlebt nicht Genetische Untersuchungen haben Gene identifiziert, die für Autophagie notwendig sind. Protease-defiziente Hefezellen wurden ausgehungert und die Akkumulation von Autophagie in den Vakuolen gezählt. Unter 5000 mutierten Hefe-Kolonien zeigten 10 keine Akkumulation von Autophagie-Körper. Atg1-Kinase-Komplex und PI3K sind nötig, um Autophagosomen zu formen. Woher kommt die Membran für die Autophagosomen Atg9 Autophagosom-Formation in Hefe: Woher kommt die Membran für die Autophagosomen Atg9? Der Membranursprung für die Autophagosomen in komplexen Eukaryoten ist offenbar nicht klar. Autophagosomen könnten durch einem Omegasome-Zwischenschritt beim ER geformt werden. Zwei ubiquitin-ähnliche Verbindungssysteme sind involviert bei der isolierten Membranerweiterung und der Frachtbereitstellung Atg12, Atg8 In Säugetierzellen existieren viele Adapter, welche die Autophagen-Membran binden können, für viele mögliche Transporte. Autophagie hat daher sehr viele Funktionen. Cell Biology SS12 (Kovarik, Martens) Seite 58 von 65 Christian Husch Martens – Foliensatz 7: Die 3 Ziele für Proteine, wenn sie das Golgi-Netzwerk verlassen wollen: Der Mechanismus der Vesikel-Sekretion vom trans-Golgi: Schritte der regulierten Exocytose: Die meisten regulierten Fusionen werden durch eine intrazelluläre Erhöhung des Ca ++-Spiegels. Das Kalzium wird durch die Mobilisation von extrazellulären Speichern bereitgestellt. Die Kalzium-Kontzentration extrazellulär ist 10.000 Mal höher als intrazellulär. Mechanismus: SNARE reagieren nicht auf Ca++ C2-Domänen sind kalziumabhängige Membran- und Proteinbindedomänen. Complexin vereinigt den SNARE-Komplex zu einer Gruppe, vor dem Ca++-Signal. Cell Biology SS12 (Kovarik, Martens) Seite 59 von 65 Christian Husch Mechanismus der erneuten Versiegelung der Plasmamembran in Muskelzellen: Dieser Vorgang basiert auf extrazellulärem Kalzium und Dysferlin (DYSF). In einem Experiment wurden Muskelzellen mit einem starken Laser beschossen und die Versiegelung des Lecks konnte durch grünen Farbstoff, welcher fluoresziert, nur innerhalb der Zelle beobachtet werden. Beispiel für polarisierte Zelle: T-Zellen können funktionelle Polarisation induzieren nach Kontakt mit der Zielzelle. Cell Biology SS12 (Kovarik, Martens) Seite 60 von 65 Christian Husch Sortierung von Membranproteinen: Transcytose reguliert den Transport von Substanzen durch die Zelle: Organellen-Vererbung: Entwicklungsgeschichtliche Strategien: Vermehrung von Organellen (Duplikation) Obwohl sie sich nicht gegenseitig ausschließen, kann die de-novo-Strategie nicht bei Organellen angewendet Cell Biology SS12 (Kovarik, Martens) Seite 61 von 65 Christian Husch werden, welche DNA enthält (Mitochondrien, Plastiden), noch durch das ER. Aufteilung der Organellen (Vererbung): Mitochondriale Teilung: Eine Zell-Zell-Fusion kann Defekte in der mitochondrialen Fusion zeigen. Cell Biology SS12 (Kovarik, Martens) Seite 62 von 65 Christian Husch Martens – Foliensatz 8: Vererbung bei der Vakuole (Hefe): Vererbung beim ER (Hefe): Proteine der Reticulon- und der YOP-Familie können Membranen in Tubuli umformen. Kleine Oligomere der Reticulon- und der YOP-Familie-Proteine sind dazu gedacht, ER-Tubuli zu stabiliseren. Ein Zusammenspiel zwischen Reticulons an den Kanten und abstandhaltenden Proteinen (Climp63) stabilisert die ER-Blätter. Cell Biology SS12 (Kovarik, Martens) Seite 63 von 65 Christian Husch Golgi-Entwicklung in Säugetierzellen: Die Golgi-Membran ist mit bindenden Faktoren bedeckt, welche die Golgi verlassenden Vesikel in der Nähe halten: Cell Biology SS12 (Kovarik, Martens) Seite 64 von 65 Christian Husch