Die Protonenpumpe der pflanzlichen Zellmembran
Werbung

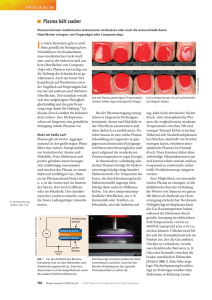
Überblick 27 Die Protonenpumpe der pflanzlichen Zellmembran Claudia Oecking Zentrum für Molekularbiologie der Pflanzen (ZMBP), Universität Tübingen In Höheren Pflanzen wird die Energie für nahezu alle bekannten Transportprozesse über die Plasmamembran durch eine protonentranslozierende P-Typ ATPase zur Verfügung gestellt. Diese H+-ATPase ist somit für eine elementare Lebensfunktion – die Aufrechterhaltung des Stoffaustauschs mit der Umgebung – von entscheidender Bedeutung und sie ist in eine Vielzahl physiologischer Prozesse eingebunden, die von der Energetisierung des Membrantransports abhängen. Die molekularen Mechanismen, die die Aktivität dieses Enzyms regulieren, sind daher von großem Interesse. Die C-terminale Domäne der H+-ATPase konnte als ein intramolekularer Inhibitor der Enzymaktivität identifiziert werden; lange Zeit waren jedoch die Regulationsmechanismen, die der – auf einer Konformationsänderung dieser autoinhibitorischen Domäne beruhenden – Aktivierung der H+-ATPase zugrunde liegen, unbekannt. Einblicke wurden hier durch die Aufklärung des Wirkmechanismus des Phytotoxins Fusicoccin erhalten. Dieses nichtselektive Pilztoxin aktiviert die Protonenpumpe praktisch irreversibel und löst demzufolge bei Pflanzen Welkeerscheinungen aus. An diesem Effekt sind eukaryotische 14-3-3 Proteine maßgeblich beteiligt. BIOspektrum · 1/02 · 8. Jahrgang Die primär aktive H+-ATPase der pflanzlichen Plasmamembran transloziert unter Hydrolyse von ATP Protonen aus dem Cytoplasma in den extrazellulären Raum und erzeugt so eine protonmotorische Kraft (∆p), die sich aus einer elektrischen Potentialdifferenz (∆E) und einer Differenz in der Wasserstoffionenkonzentration (∆pH) zusammensetzt (∆p = ∆E – 59∆pH (mV)). Diese protonmotorische Kraft stellt die Grundlage vieler sekundärer Transportprozesse dar, die über Carrier und Kanäle vermittelt werden (Abb. 1). Demnach ist die protonentranslozierende ATPase für die Energetisierung des Membrantransports verantwortlich und sie trägt zu Wachstum und Differenzierung pflanzlicher Organismen bei. Dieser Beitrag gibt einen kurzen Überblick über den derzeitigen Kenntnisstand in bezug auf die Struktur, Funktion und Regulation dieser bedeutenden pflanzlichen Ionenpumpe. Struktur der pflanzlichen H+-ATPase Abb. 1: Schema zu primär aktiven sowie sekundären Transportprozessen über die Plasmamembran. Die H+-ATPase erzeugt als primär aktive Pumpe eine protonmotorische Kraft, die sekundäre Transportprozesse über Kanäle und Carrier ermöglicht. Kanäle vermitteln den Transport von Ionen entlang ihrer elektrochemischen Gradienten. Carrier arbeiten entweder passiv (Uniporter) oder sekundär aktiv (Kotransporter: Symporter oder Antiporter), indem sie den Transport von Metaboliten über die Plasmamembran an die elektrochemisch begünstigte Translokation von Protonen ins Zellinnere koppeln. H+-ATPasen bestehen aus einem einzigen Polypeptid mit einer molekularen Masse um 100 kDa. Die Elektronenkristallographie zweidimensionaler Kristalle der H+-ATPase von N. crassa belegt die Existenz von zehn transmembranen α-Helizes und vier cytoplasmatischen Domänen (Abb. 2) [1]. Weniger als 10% des Proteins sind extrazellulär orientiert; im Cytoplasma befinden sich sowohl der N- als auch der C-Terminus des Enzyms sowie die sogenannte kleine und große Schleife. Letztere beinhaltet die ATPBindestelle und den Aspartatrest, der im Verlauf der Katalyse phosphoryliert wird [2]. Die C-terminale Domäne der pflanzlichen Protonenpumpe wirkt autoinhibitorisch: erfolgt eine selektive Abspaltung dieser Domäne vom Rest des Enzyms mittels einer milden Proteolyse (Trypsin), so weist das C-terminal verkürzte Enzym eine eindeutig erhöhte Aktivität auf [3]. Physiologische Funktionen der pflanzlichen H+-ATPase P-Typ ATPasen bilden eine große Familie von Membranproteinen, die ATP nutzen, um Ionengradienten über Zellmembranen aufzubauen. Die Bezeichnung ‚P-Typ’ geht auf die Phosphorylierung eines Aspartatrestes im Verlauf der Katalyse zurück. Das Synonym E1/E2-ATPase nimmt Bezug auf detaillierte Untersuchungen von Säuger P-Typ ATPasen, deren katalytischer Zyklus auf einer – phosphorylierungsabhängigen – Konversion zweier Konformationszustände beruht. Diese unterscheiden sich hinsichtlich ihrer Affinität gegenüber dem zu translozierendem Ion; der Mechanismus der Ionentranslokation ist jedoch bislang unbekannt. Viele physiologische Prozesse, die von der protonmotorischen Kraft abhängen, werden durch die Aktivität der H+-ATPase beeinflusst. Besonders eindrücklich macht dies eine Behandlung pflanzlicher Gewebe mit dem Pilztoxin Fusicoccin (FC, Abb. 3) deutlich. Dieses Diterpenglucosid ist das Haupttoxin des phytopathogenen Pilzes Fusicoccum amygdali und aktiviert die Protonenpumpe in vivo nahezu irreversibel [4]. Darüber hinaus wird u.a. die Aufnahme und der Transport von Nährstoffen stimuliert, eine unphysiologisch starke Stomataöffnung induziert und die Zellstreckung gefördert; Wirkungen, die in Summe auf die primäre Überblick 28 Abb. 2: Topologie der H+-ATPase der Plasmamembran. Nähere Erläuterungen sind dem Text zu entnehmen. Stimulation der H+-ATPase zurückzuführen sind. Essentielle mineralische Nährelemente (z. B. Stickstoff, Schwefel, Phosphor, Kalium) nimmt die Pflanze in ionischer Form aus dem Bodenwasser in die Wurzel auf. Zudem werden Aminosäuren als bedeutende Transportform reduzierter Stickstoffverbindungen und Kohlenhydrate als Produkt der photosynthetischen CO2-Fixierung über das Xylem und/oder Phloem in der Pflanze verteilt. Der Transport von Ionen und Metaboliten in die Zelle erfolgt dabei häufig entgegen Konzentrationsgradienten bzw. elektrochemischen Gradienten und erfordert daher die Kopplung an eine exergone Reaktion. Sogenannte Protonen-Symporter (sekundär aktive Carrier) nutzen die Energie, die in dem elektrochemischen Protonengradienten gespeichert ist und ermöglichen – auf der Grundlage des elektrochemisch begünstigten Kotransports von Protonen (Abb. 1) – die Aufnahme vieler anorganischer und organischer Nährstoffe in die Zelle. Pflanzen bauen im Gegensatz zu Tieren einen hohen intrazellulären Druck, den sogenannten Turgor, auf. Eine Turgorerhöhung ist beispielsweise die Ursache für das Öffnen der Stomata (Abb. 3), die für den Gasaustausch von Pflanzen verantwortlich sind. Der Turgor kann über eine Veränderung des osmotischen Potentials der Zelle und den damit verbundenen Wassereinstrom oder -ausstrom über selektive Poren (Aquaporine) beeinflusst werden. Auch an diesem Prozess ist die H+-ATPase maßgeblich beteiligt: ihre Aktivierung führt über eine Hyperpolarisation des Membranpotentials zu einer Öffnung spannungsabhängiger K+-Kanäle und ermöglicht den passiven Einstrom osmotisch wirksamer K+-Ionen (Abb. 1). Dies hat einen Anstieg des osmotischen Potentials und des Turgors zur Folge, der mechanisch – bedingt durch die ungewöhnliche Zellwandstruktur der Schließzellen – in eine verstärkte Öffnung der Stomata umgesetzt wird. Die pflanzliche Zellwand stellt eine mechanische Barriere dar, die die Zellvergrößerung einschränkt. Eine Ansäuerung der Zellwand, die sich auf eine gesteigerte Aktivität der H+-ATPase zurückführen lässt, initiiert das Zellstreckungswachstum. Dieser Prozess ist als Säure-Wachstumshypothese bekannt und kann vereinfacht als säureinduzierte Zellwanderweichung aufgefasst werden, die die Aufnahme von Wasser und somit die Zellstreckung ermöglicht. Die H+-ATPase stellt eine bedeutende Komponente dieses Prozesses dar: neben der Initiierung des säureinduzierten Wachstums ist sie für die Aufrechterhaltung des Zellturgors im Verlauf der Zellstreckung verantwortlich (s. o.). Regulation der pflanzlichen H+-ATPase Berücksichtigt man den hohen Energieverbrauch der H+-ATPase und die Vielzahl der physiologischen Prozesse pflanzlicher Zellen, in die dieses Enzym eingebunden ist, so muss von einer strikten Regulation in vivo ausgegangen werden. Daher verwundert es nicht, dass die Aktivität der Protonenpumpe durch zahlreiche Stimuli beeinflusst wird. Hierzu gehören beispielsweise Licht, Phytohormone (Auxine, Brassinosteroide), Toxine und andere Umwelteinflüsse [5]. Physiologische Studien wiesen jeweils auf die H+ATPase als das hauptsächliche Zielsystem dieser regulatorischen Ereignisse hin; der molekulare Mechanismus der Regulation konnte jedoch nur in wenigen Fällen aufgeklärt werden. Im Folgenden werden die derzeit diskutierten Möglichkeiten zur Re- gulation der Aktivität der H+-ATPase in Kürze vorgestellt. Abschließend wird auf den Mechanismus der FC-Wirkung eingegangen, der zur Entdeckung sogenannter 14-3-3 Proteine als physiologische Aktivatoren der H+-ATPase führte. Die protonentranslozierende ATPase der pflanzlichen Plasmamembran wird von einer erstaunlich großen Multigenfamilie kodiert, die zum heutigen Zeitpunkt molekular bereits recht gut charakterisiert ist. So wurden beispielsweise zehn Gene in A. thaliana und neun in Nicotiana plumbaginifolia identifiziert [2,5]. Die Expression einzelner Isoformen erfolgt gewebespezifisch und entwicklungsabhängig und wird z.T. durch Umwelteinflüsse (mechanischer Stress, Salzstress) reguliert; dies stellt sicherlich den bedeutsamsten Faktor dar, der es Pflanzen ermöglicht, die Energetisierung der Plasmamembran den jeweiligen „Transportbedürfnissen“ einzelner Zellen und Geweben anzupassen [2,5]. Neben dieser transkriptionellen Regulation ist jedoch auch eine Regulation des Enzyms auf der Ebene einer posttranslationalen Modifikation notwendig, die insbesondere eine schnelle Antwort auf Umwelteinflüsse ermöglicht. Als bedeutender Botenstoff ist Calcium in diverse pflanzliche Signaltransduktionsprozesse eingebunden. Es gibt Hinweise darauf, dass physiologische Ca2+-Konzentrationen eine reversible Inaktivierung der H+-ATPase in Schließzellen zur Folge haben [6] und somit den turgorgesteuerten Stomataverschluss einleiten. Dieser Effekt scheint jedoch indirekt zu sein [2,5] und wird möglicherweise durch eine Phosphorylierung des Enzyms, die auf die Aktivität einer Ca2+-abhängigen Proteinkinase (CDPK) zurückzuführen ist, vermittelt [2]. Der C-Terminus der pflanzlichen H+ATPase stellt eine regulatorische Domäne dar, deren autoinhibitorische Aktivität in vivo nachgewiesen werden konnte [7].Über die molekulare Grundlage einer Aktivierung der H+-ATPase, die auf einer posttranslationalen Regulation ihrer C-terminalen Domäne beruht, war lange Zeit wenig bekannt. 1993 konnte gezeigt werden, dass die autoinhibitorische Aktivität dieses intramolekularen Regulators in Gegenwart von FC – vermutlich aufgrund einer Konformationsänderung – aufgehoben wird [8]. Dies hat eine irreversible Aktivierung der Protonenpumpe zur Folge und bedingt aufgrund einer irreversiblen Öffnung der Stomata (s.o.) letztendlich ein Welken der Pflanzen (Abb. 3). Die FC-Wirkung wird über ein hochaffines FC-bindendes Protein (FCBP) der Plasmamembran vermittelt, so dass man sich grundlegendere Aufschlüsse über den Mechanismus der FC-Wirkung durch die Identifizierung des FCBP erhoffte. BIOspektrum · 1/02 · 8. Jahrgang Überblick 29 Mechanismus der FC-Wirkung Die zahlreichen Untersuchungen zum FCBindeprotein haben zu einem klassischen Rezeptorkonzept geführt, demzufolge das FCBP als ein integrales Membranprotein mit einer extrazellulären Bindestelle für FC anzusehen ist [9]. Mit der Entdeckung, dass sogenannte 14-3-3 Proteine, die keinerlei hydrophobe Domänen aufweisen, zumindest eine Komponente des FCBP darstellen [10, 11, 12] , musste dieses klassische Rezeptorkonzept jedoch in Frage gestellt werden. 14-3-3 Proteine wurden 1967 als dominante Proteine des Säugerhirns identifiziert. Ihren ungewöhnlichen Namen haben sie aufgrund ihrer Wanderungsgeschwindigkeit im Verlauf einer zweidimensionalen Auftrennung erhalten. Nach heutigem Kenntnisstand sind Mitglieder der hochkonservierten 14-3-3 Proteinfamilie ubiquitär unter Eukaryoten verbreitet und sie gelten als multifunktionelle Regulatoren zellulärer Signalprozesse; ihre regulatorische Funktionen üben sie über direkte Proteininteraktionen aus [13]. 14-3-3 Homologe selbst bilden Homo- oder Heterodimere mit einer klammerartigen Struktur aus [14]; jedes Monomer verfügt dabei über eine amphipathische Grube, die eine Bindung an Zielpeptide ermöglicht. Die Interaktion von 14-3-3 Proteinen mit ihren Zielproteinen setzt i.a. die Phosphorylierung eines Serinrestes innerhalb eines spezifischen Sequenzmotives voraus. Die entsprechenden Proteine werden als Folge der 14-3-3 Bindung in ihrer Aktivität moduliert, bezüglich ihrer intrazellulären Lokalisation beeinflusst oder auf- grund der Adaptorfunktion der 14-3-3 Dimere in räumliche Nähe eines weiteren Proteins gebracht [13]. Die detaillierten Untersuchungen zur Interaktion von 14-3-3 Homologen mit der H+ATPase haben deutlich machen können, dass 14-3-3 Proteine direkt an den C-Terminus der pflanzlichen H+-Pumpe binden und so seine autoinhibitorische Funktion unterbinden [15, 16, 17]. Unter physiologischen Bedingungen wird die Aktivität der H+ATPase über eine Veränderung ihres Phosphorylierungsstatus reguliert. Erfolgt eine Phosphorylierung des Threoninrestes im extrem C-terminalen Tripeptid YTV [18, 19, 20] durch eine bislang unbekannte Kinase (YpTV), so wird das Gleichgewicht zwischen dem 14-3-3 komplexierten Enzym und den Einzelkomponenten in Richtung des Komplexes verschoben (Abb. 4). Cytoplasmatische 14-3-3 Proteine bewirken durch ihre Assoziation an die C-terminale Domäne der H+-ATPase eine Konformationsänderung dieses intramolekularen Regulators und bedingen so eine Aktivierung des Enzyms. FC bindet an den ausgebildeten Komplex, der das funktionelle FCBP repräsentiert, und kann ihn arretieren. Jedoch scheint FC die Aktivität der H+-ATPase auch unabhängig vom Phosphorylierungsstatus beeinflussen zu können [18, 19] und das Gleichgewicht in Richtung des Komplexes zu verschieben (Abb. 4). Interessanterweise weist das im Bereich des C-Terminus der H+-ATPase identifizierte Assoziationssignal für 14-3-3 Proteine (YpTV) keinerlei Ähnlichkeit zu bekannten, charakteristischen 14-3-3 Bindemotiven auf [13]; zudem konnte ein zweites essentielles Motiv identifiziert werden, das mit hoher Wahrscheinlichkeit eine amphipathische Helix ausbildet [21]. Die Möglichkeit der Bindung eines solchen Sekundärstrukturelements wurde bereits im Rahmen der Strukturaufklärung eines tierischen 14-3-3 Dimers diskutiert [14], bislang jedoch in keinem Fall gezeigt. So ist vorstellbar, dass jedes 14-3-3 Monomer eins dieser Motive in seiner amphipathischen Grube bindet und, dass ausschließlich diese Art der bivalenten Interaktion eine Konformationsänderung der autoinhibitorischen Domäne der H+-ATPase bewirkt. Zukünftig sollte insbesondere analysiert werden, welche Umwelteinflüsse zu einer phosphorylierungsabhängigen Bindung von 14-3-3 Proteinen und dementsprechend zu einer Aktivierung der H+-ATPase führen. So haben aktuelle Untersuchungen bereits zeigen können, dass die durch Blaulicht induzierte Stimulation der Protonenpumpe, die für die Öffnung der Stomata von Bedeutung ist, über 14-3-3 Proteine vermittelt wird [22]. Literatur [1] Auer, M., Scarborough, G.A., Kühlbrandt, W. (1998): Three-dimensional map of the plasma membrane H+-ATPase in the open conformation. Nature 392: 840–843 [2] Morsomme, P., Boutry, M. (2000): The plant plasma membrane H+-ATPase: structure, function and regulation. Biochim. Biophys. Acta 1465: 1–16 [3] Palmgren, M.G., Sommarin, M., Serrano, R., Larsson, C. (1991): Identification of an autoinhibitory domain in the C-terminal region of the plant plasma membrane H+-ATPase. J. Biol. Chem. 266: 20470–20475 [4] Marrè, E. (1979): Fusicoccin. A tool in plant physiology. Annu. Rev. Plant Physiol. 30: 273–288 [5] Palmgren, M.G. (1998): Proton gradients and plant growth: role of the plasma membrane H+-ATPase. Adv. Bot. Res. 28: 1–71 [6] Kinoshita, T., Nishimura, M., Shimazaki, K.-I. (1995): Cytosolic concentration of Ca2+ regulates the plasma membrane H+-ATPase in guard cells of fava bean. Plant Cell 7: 1333–1342 [7] Regenberg, B., Villalba, J., Lanfermeijer, F., Palmgren, M.G. (1995): C-terminal deletion analysis of plant plasma membrane H+-ATPase: yeast as a model system for solute transport across the plant plasma membrane. Plant Cell 7: 1655–1666 [8] Johansson, F., Sommarin, M., Larsson, C. (1993): Fusicoccin activates the plasma membrane H+-ATPase by a mechanism involving the C-terminal inhibitory domain. Plant Cell 5: 321–327 [9] Aducci, P., Marra, M., Fogliano, V., Fullone, M. (1995): Fusicoccin receptors: perception and transduction of the fusicoccin signal. J. Exp. Bot. 46: 1463–1478 Abb. 3: Strukturformel und Wirkung des „Welketoxins“ Fusicoccin. Im Hintergund ist die Epidermis der Blattunterseite von Commelina communis gezeigt; die Stomata sind infolge einer FC-Behandlung (3 µM) im Dunkeln geöffnet. BIOspektrum · 1/02 · 8. Jahrgang [10] Oecking, C., Eckerskorn, C., Weiler, E.W. (1994): The fusicoccin receptor of plants is a member of the 14-3-3 superfamily of eukaryotic regulatory proteins. FEBS Lett. 352: 163–166 Überblick 30 Abb. 4: Modell zur Interaktion von 14-3-3 Proteinen mit der autoinhibitorischen Domäne der H+ATPase der pflanzlichen Plasmamembran sowie der Wirkungsweise des Phytotoxins Fusicoccin. T bezeichnet die Aminosäure Threonin im hochkonservierten Tripeptid YTV der H+-ATPase, pT die entsprechende Aminosäure in phosphorylierter Form. Nähere Erläuterungen sind dem Text zu entnehmen [11] Marra, M., Fullone, M., Fogliano, V., Pen, J., Mattei, M., Masi, S., Aducci, P. (1994): The 30-kilodalton protein present in purified fusicoccin receptor preparations is a 14-3-3-like protein. Plant Physiol. 106: 1497–1501 [12] Korthout, H., de Boer, A.H. (1994): A fusicoccin binding protein belongs to the family of 14-3-3 brain protein homologs. Plant Cell 6: 1681–1692 [19] Fuglsang, A., Visconti, S., Drumm, K., Jahn, T., Stensballe, A., Mattei, B., Jensen, O., Aducci, P., Palmgren, M.G. (1999): Binding of 14-3-3 protein to the plasma membrane H+-ATPase AHA2 involves the three Cterminal residues Tyr946-Thr-Val and requires phosphorylation of Thr947. J. Biol. Chem. 274: 36774–36780 [20] Maudoux, O., Batoko, H., Oecking, C., Gevaert, K., Vandekerckhove, J., Boutry, M., Morsomme, P. (2000): A plant plasma membrane H+-ATPase expressed [13] Finnie, C., Borch, J., Collinge, D. (1999): 14-3-3 proteins: eukaryotic regulatory proteins with many functions. Plant Mol. Biol. 40: 545–554 in yeast is activated by phosphorylation at its penultimate residue and binding of 14-3-3 regulatory proteins in the absence of fusicoccin. J. Biol. Chem. 275: 17762–17770 [14] Liu, D., Bienkowska, J., Petosa, C., Collier, R.J., Fu, H., Liddington, R. (1995): Crystal structure of the zeta isoform of the 14-3-3 protein. Nature 376: 191–194 [21] Jelich-Ottmann, C., Weiler, E.W., Oecking, C. (2001): Binding of regulatory 14-3-3 proteins to the C terminus of the plant plasma membrane H+-ATPase involves part of ist autoinhibitory region. J. Biol. Chem. 276: 39852–39857 [15] Oecking, C., Piotrowski, M., Hagemeier, J., Hagemann, K. (1997): Topology and target interaction [22] Kinoshita, T., Shimazaki, K.-I. (1999): Blue light activates the plasma membrane H+-ATPase by phosphorylation of the C-terminus in stomatal guard cells. EMBO J. 18: 5548–5558 Korrespondenzadresse Prof. Dr. Claudia Oecking Zentrum für Molekularbiologie der Pflanzen Universität Tübingen Auf der Morgenstelle 5 D-72076 Tübingen Tel.: (07071) 29 76679 eMail: [email protected] of the fusicoccin-binding 14-3-3 homologs of Commelina communis. Plant J. 12: 441–453 [16] Jahn, T., Fuglsang, A., Olsson, A., Brüntrup, I., Collinge, D., Volkmann, D., Sommarin, M., Palmgren, M.G., Larsson, C. (1997): The 14-3-3 protein interacts directly with the C-terminal region of the plant plasma membrane H+-ATPase. Plant Cell 9: 1805–1814 [17] Piotrowski, M., Morsomme, P., Boutry, M., Oecking, C. (1998): Complementation of the Saccharomyces cerevisiae plasma membrane H+-ATPase by a plant H+-ATPase generates a highly abundant fusicoccin binding site. J. Biol. Chem. 273: 30018–30023 [18] Svennelid, F., Olsson, A., Piotrowski, M., Rosenquist, M., Ottmann, C., Larsson, C., Oecking, C., Sommarin, M. (1999): Phosphorylation of Thr-948 at the C terminus of the plasma membrane H+-ATPase cre- ates a binding site for regulatory 14-3-3 protein. Plant Cell 11: 2379–2391 Claudia Oecking geboren 1963, Studium der Biologie an der Ruhr-Universität Bochum, Promotion 1993 bei Prof. Dr. E. W. Weiler (Lehrstuhl für Pflanzenphysiologie), 1999 Arbeitsaufenthalt an der Université Catholique de Louvain, Belgien, 2001 Habilitation für das Fachgebiet Botanik, seit 2001 Professorin für Molekularbiologie der Pflanzen am ZMBP der Universität Tübingen. BIOspektrum · 1/02 · 8. Jahrgang