Eiweiß-Synthese, Modell 65562.00
Werbung

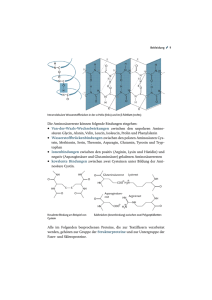
R Eiweiß-Synthese, Modell 65562.00 Betriebsanleitung Abb. 1 1. EINFÜHRUNG Alle lebenden Zellen enthalten Eiweißstoffe (Proteine). Sie stellen einerseits einen wesentlichen Baustoff zum Aufbau der Lebewesen dar, anderseits sind auch die Enzyme, auf deren Wirkung alle Stoffumsetzungen in den Lebewesen beruhen, Eiweißstoffe. Die Eiweiße bilden lange Kettenmoleküle mit einem Molekulargewicht von etwa 10.000 bis zu einigen Millionen. Bei Hydrolyse zerfallen sie in Aminosäuren. Nach unseren heutigen Kenntnissen sind 20 Aminosäuren am Aufbau der Eiweißstoffe beteiligt. Durch Peptidbindungen (Abb.2) werden sie miteinander verknüpft, indem die Carboxylgruppe der einen Aminosäure mit der Aminogruppe der anderen unter Wasserabspaltung reagiert. Auf diese Weise entsteht aus zwei Aminosäuren ein Dipeptid, aus drei ein Tripepted usw. Ketten bis zu 10 Aminosäuren nennt man Oligopeptide, Ketten mit mehr als 10 Aminosäuren Polypeptide. Die Eiweißstoffe sind also Polypeptide. Die Vielzahl der für jedes Lebewesen artspezifischen Eiweiße kommt dadurch zustande, daß die an ihrem Aufbau beteiligten Aminosäuren jeweils in einer bestimmten Reihenfolge angeordnet sind. Zur Bildung der Eiweißstoffe zur Proteinsynthese - muß deshalb die Reihenfolge, in der die Aminosäuren miteinander verknüft werden müssen, in irgendeiner Form als Bauvorschrift in den Zellen der Lebewesen angegeben sein. Wir wissen heute, daß genetische Informationen durch die Reihenfolge der Basen in den DNS-Molekülen der Chromosomen festgelegt sind. Im DNS-Molekül kommen vier verschiedene Basen vor: Adenin (A), Thymin (T), Guanin (G) und Cytosin (C) (Abb.3). Würde eine einzelne Base (bzw. ein einzelnes Nucleotid) jeweils eine Aminosäure bezeichnen, so könnten nur vier verschiedene Aminosäuren bestimmt und in die Peptidkette eingebaut werden. Würden für die Kennzeich- Abb. 2 Thymin Adenin Guanin Cytosin Abb. 3 nung einer Aminosäure jeweils zwei nebeneinanderliegende Basen (bzw. Nucleotide) verwendet, so könnten 16 Aminosäuren bestimmt und eingebaut werden, da vier Zeichen (hier Basen) in Zweiergruppen in 16 verschiedenen Anordnungen kombiniert werden können: AA GA AC GC AG GG AT GT CA TA CC TC CG TG CT TT Da 20 Aminosäuren am Aufbau der Eiweißstoffe beteiligt sind, reichen jedoch auch die Zweiergruppen (Dupletts) zur Kennzeichnung aller Aminosäuren nicht aus. Wir wissen heute, daß jeweils eine Dreiergruppe von Basen - ein Ba- PHYWE SYSTEME GMBH · Robert-Bosch-Breite 10 · D-37079 · Göttingen · Telefon (05 51) 6 04-0 · Telefax (05 51) 60 41 07 sentriplett (z.B. AAA, ACT, GAC usw.) - eine Aminosäure bezeichnet (codiert). Drei nebeneinanderliegende Nucleotide eines DNS-Stranges bezeichnet man als ein Codogen. Bei vier Zeichen (hier Basen), die jeweils in Dreiergruppen angeordnet werden, sind 64 verschiedene Kombinationen - d.h. Tripletts oder Codogens - möglich. Es könnten also 64 Aminosäuren gekennzeichnet werden. 20 Aminosäuren sind jedoch nur an der Bildung der Eiweißstoffe beteiligt. Der Code ist also umfangreicher und enthält mehr Möglichkeiten als erforderlich erscheint. Untersuchungen haben ergeben, daß manche Aminosäuren durch mehrere Tripletts bezeichnet werden und bestimmte Tripletts das Ende der Aminosäurekette bei der Proteinsynthese anzeigen. der DNS, nur immer die Basen Guanin und Cytosin einerseits und Adenin und Uracil (anstelle von Thymin) anderseits gegenüberliegend miteinander verbinden. Der entstehende RNS-Strang tastet infolgedessen als Negativ die auf dem Einzelstrang der DNS gespeicherte Information ab. Ein Basentriplett der RNS nennt man ein Codon. Der RNSStrang löst sich von dem Einzelstrang der DNS, wandert durch die Kernmembran in das Zytoplasma und überträgt die Information für die Reihenfolge der Aminosäure im Eiweißmolekül zu den Ribosomen. Diese RNS heißt deshalb Boten- oder messenger-RNS (m-RNS). Der messengerRNS-Strang legt sich an die Ribosomen. An seine Codons lagern sich passende Basentripletts - sogenannte Anticodons - kurzer, im Zytoplasma vorhandener RNS-Moleküle an. Die kurzen RNS-Stränge sind entsprechend ihrem Basentripletts (dem Anticodon) mit einer bestimmten Aminosäure gekoppelt, die auf diese Weise zum m-RNS-Strang transportiert wird. Diese RNS heißt deshalb transfer-RNS (t-RNS). Die m-RNS liegt jeweils nur mit zwei bis drei Codons an den Ribosomen an. Nur an dieser Stelle erfolgt die Anlagerung der passenden Anticodons der t-RNS. Die herangeführten Aminosäuren werden durch Peptidbindungen (s.o) miteinander verknüpft. Danach lösen sich die t-RNSStränge sowohl von den aneinandergereihten Aminosäuren als auch vom m-RNS-Strang und stehen zur Koppelung und Übertragung weiterer (jeweils gleichartiger) Aminosäurenmoleküle zur Verfügung. Der m-RNS-Strang gleitet ein Stück weiter am Ribosom entlang, so daß nunmehr die zwei bis drei danebenliegenden Codons an das Riobosom gelangen, wo die Anlagerung von Aminosäuren - die Proteinsynthese - weitergeht. Auf diese Weise entsteht entsprechend der von der DNS über die m-RNS weitergegebenen Information ein Eiweißstrang, dessen Aminosäuresequenz eben durch diese Information bestimmt wird. Der Sitz der genetischen Information sind die DNS-Moleküle in den Chromosomen der Zellkerne. Die Eiweißsynthese findet jedoch außerhalb der Zellkerne im Zytoplasma an den Ribosomen statt. Die Information muß infolgedessen von den Zellkernen zu den Ribosomen übertragen werden. Das geschieht auf eine komplizierte Art und Weise. Der DNS-Doppelstrang trennt sich - zumindest streckenweise - in die beiden Einzelstränge. An den DNS-Einzelstrang legen sich aus dem Zytoplasma stammende Nucleotide an und bilden einen Einzelstrang, der die in der Basensequenz des DNS-Stranges festgelegte Information abtastet. Es handelt sich dabei jedoch nicht, wie bei der identischen Vermehrung der DNS, um einen neuen DNSStrang, denn bei diesen Nucleotiden ist die Desoxyribose der DNS (Abb.4) durch die sehr ähnlich gebaute Ribose (Abb. 5) und die Base Thymin durch die Base Uracil (Abb. 6) ersetzt. Es können sich aber, wie bei der identischen Vermehrung Abb. 4 2-Desoxyribose Abb. 5 Ribose 2. DAS MODELL Das Modell besteht aus einem Satz von 52 Symbolen (Abb.7). Neben den abgebildeten Symbolen befinden sich deren Bezeichnungen (die Zahlen in Klammern bezeichnen die Anzahl der einzelnen Symbole im Satz). Abb. 6 Alle Symbole besitzen auf ihrer Rückseite einen Magneten, mit dem sie auf einer Metalltafel angeheftet werden können. Haken und schlitzförmige Ösen dienen ihrer Verknüpfung untereinander. Zur Demonstration der Eiweißsynthese wird zunächst aus den entsprechenden Nucleotiden ein DNS-Doppelstrang Uracil 2 65562.00 rot rot schwarz rot schwarz rot schwarz elfenbein schwarz gelb grün Nucleotid der DNS mit der Base Thymin (8) blau Nucleotid der DNS mit der Base Adenin (8) orangerot Nucleotid der DNS mit der Base Cytosin (8) orangerot schwarz Nucleotid der DNS mit der Base Guanin (8) orangerot schwarz orangerot schwarz violettblau schwarz gelb grün Nucleotid der m-RNS mit der Base Uracil (3) blau Nucleotid der m-RNS mit der Base Adenin (2) Nucleotid der m-RNS mit der Base Cytosin (3) Nucleotid der m-RNS mit der Base Guanin (4) orangerot schwarz t-RNS-Strang mit dem Anticodon Cytosin (C)/ Guanin (G)/Adenin (A) (1), überträgt die Aminosäure Alanin gelb blau grün orangerot schwarz braun t-RNS-Strang mit dem Anticodon Cytosin (C)/ Ademin (A)/Adenin (A) (1) , überträgt die Aminosäure Valin gelb grün grün Aminosäure Valin (1) orangerot schwarz braun t-RNS-Strang mit dem Anticodon Guanin (G)/ Cytosin (C)/Uracil (U) (2), überträgt die Aminosäure Arginin blau gelb violettblau Aminosäure Arginin (2) Abb. 7 3 65562.00 aufgebaut (Abb. 8). Bei einem seiner Einzelstränge muß die Reihenfolge der Nucleotide mit der Basensequenz der Anticodons der t-RNS-Symbole identisch sein. Dabei können sich nur die Basen Adenin und Thymin einerseits und Guanin und Cytosin anderseits miteinander paaren, d.h. einander gegenüber stehen, was durch die entsprechenden Passungen - Winkel oder Rundung - an den Symbolen zum Ausdruck kommt. Der DNS-Einzelstrang bei dem die Reihenfolge der Nucleotide nicht der Basensequenz der Anticodons entspricht, wird durch Verschieben auf der Metalltafel vom Partnerstrang getrennt. Um ein Ausbrechen der schlitzförmigen Ösen oder ein Abbrechen der Haken mit Sicherheit zu vermeiden, sollten zum Verschieben längerer Symbolketten auf der Tafel beide Hände benutzt und die Ketten möglichst gleichmäßig gebogen, aber nicht abgeknickt werden. Abb. 8 An den verbleibenden DNS-Einzelstrang wird aus den entsprechnenden RNS-Nucleotiden ein m-RNS-Strang angebaut, der die genetische Information zur Eiweißsynthese abtastet. Zur besseren Veranschaulichung der örtlichen Verhältnisse in der Zelle können die Kernmembran und ein Ribosom mit Kreide auf die Metalltafel gezeichnet werden. (Abb. 9). Der m-RNS-Strang wird zum Ribosom verschoben. Nach der Verknüpfung der t-RNS-Symbole mit den dazugehörigen Aminosäure-Symbolen (s.o.) werden die t-RNS-Symbole in der richtigen Reihenfolge zu den passenden Stellen an den m-RNS-Strang herangeführt (Abb. 10). Auf diese Weise entsteht eine kurze Kette von Aminosäuren. Abb. 9 Die identische Vermehrung - die Replikation - der DNS kann mit dem Modell ebenfalls sehr anschaulich gezeigt werden. Zu diesem Zweck wird zunächst ein DNS-Doppelstrang mit beliebiger Basensequenz aufgebaut. Die Kettenlänge soll etwa 10-12 Nucleotide betragen. Man zieht den Doppelstrang an einem Ende in seine beiden Einzelstränge auseinander und ergänzt mit den noch vorhandenen Nucleotiden beide Einzelstränge zum Doppelstrang (Abb. 11). Da sich immer nur Adenin und Thymin einerseits und Guanin und Cytosin andererseits gegenüber liegen können, entstehen zwei Doppelstränge mit völlig gleicher Basensequenz. 3. MATERIAL Eiweiß-Synthese, Modell Satz von 52 magnetisch haftenden Symbolen aus Kunststoff TAFEL; Metall 1480 mm x 980 mm Abb. 10 65562.00 60377.00 Abb. 11 4 65562.00