Größenbestimmung bei Mikroarrayexperimenten - Goethe
Werbung

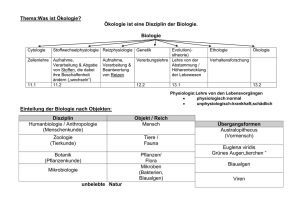
Johann Wolfgang Goethe Universität Frankfurt am Main Fachbereich 15: Biologie und Informatik Junior Prof. Dr. Dirk Metzler Größenbestimmung bei Microarrayexperimenten Klassenvergleiche und Classifier Sebastian Bremm 1 Johann Wolfgang Goethe Universität Frankfurt am Main Fachbereich 15: Biologie und Informatik Junior Prof. Dr. Dirk Metzler Microarrays • Dienen zur Erkennung von Expressionsprodukten. • Platten aus Glas, Silizium etc. • Enthalten die Gene des Organismus. • Position jedes Gens auf Platte ist bekannt. Sebastian Bremm 2 Johann Wolfgang Goethe Universität Frankfurt am Main Fachbereich 15: Biologie und Informatik Junior Prof. Dr. Dirk Metzler Versuchsablauf • Transkriptionsprodukte (Targets) werden auf das MA gegeben. Diese sind mit Fluoreszenz-Markern versehen. • Binden der Targets an den komplementären Strängen auf dem MA. • Waschen um nicht oder unzureichend gebundene Targets zu entfernen. Sebastian Bremm 3 Johann Wolfgang Goethe Universität Frankfurt am Main Fachbereich 15: Biologie und Informatik Junior Prof. Dr. Dirk Metzler Auswertung • Farbstoffe werden durch Laser zum Leuchten gebracht. • Scannen des Bildes. • Normalisierung. • Fehlerbeseitigung • Erstellen der Genexpressionsmatrix. Sebastian Bremm 4 Johann Wolfgang Goethe Universität Frankfurt am Main Fachbereich 15: Biologie und Informatik Junior Prof. Dr. Dirk Metzler Warum Versuchsgrößenbestimmung? • Beschränkungen durch: – Finanzmittel – Zeit – Vorhandene Proben Sebastian Bremm 5 Johann Wolfgang Goethe Universität Frankfurt am Main Fachbereich 15: Biologie und Informatik Junior Prof. Dr. Dirk Metzler Klassen-Vergleiche • Vergleich von zwei Gewebetypen z.B.: - Krebsgewebe normales Gewebe - histologisch verschiedene Krebsgewebe • Ziel ist es, unterschiedliche GenExpressionen zu identifizieren. Sebastian Bremm 6 Johann Wolfgang Goethe Universität Frankfurt am Main Fachbereich 15: Biologie und Informatik Junior Prof. Dr. Dirk Metzler Verschiedene MA-Versuchstypen • Single Label/ Double Label – Pooling – Dye Swap – Nutzen von technischen Replikaten Sebastian Bremm 7 Johann Wolfgang Goethe Universität Frankfurt am Main Fachbereich 15: Biologie und Informatik Junior Prof. Dr. Dirk Metzler Single Label Arrays • • • • DNA – Oligonukleotid MAs (Affymetrix) Nur Targets einer Zelle Hohe Spotdichte teuer Sebastian Bremm 8 Johann Wolfgang Goethe Universität Frankfurt am Main Fachbereich 15: Biologie und Informatik Junior Prof. Dr. Dirk Metzler Notation • Floureszens-Intensität: Ygadvfs – – – – – – g :1,2,...G | Gen a :1,...,n | Array d : 1 = Single Label; 2 = double Label | Farbe v : 1,2 | Phänotypen f : 1,2,...F | Individuen s : 1,2,...m | Unterprobe/technisches Replikat Sebastian Bremm 9 Johann Wolfgang Goethe Universität Frankfurt am Main Fachbereich 15: Biologie und Informatik Junior Prof. Dr. Dirk Metzler Single Label MAs • Log (Ygadvfs) = Gg + GVgv + (GF)gf(v) + gadvfs – – – – Gg = Genexpression von g in der Population GVgv = Effekt der Klasse oder des Typs (GF)gf(v) = individueller Effekt 2 gadvfs = unabh. Fehler mit Normal(0, x ) Sebastian Bremm 10 Johann Wolfgang Goethe Universität Frankfurt am Main Fachbereich 15: Biologie und Informatik Junior Prof. Dr. Dirk Metzler Single Label MAs Yg1fs z fs , Yg2fs w fs zij x xi ij ; i 1...n; j 1..m xi ~ Normal (0, x2 ) ij ~ Normal (0, 2 ) wkl y y k kl ; k 1...n; l 1..m y k ~ Normal (0, x2 ) kl ~ Normal( 0 ,σ 2 ) Sebastian Bremm 11 Johann Wolfgang Goethe Universität Frankfurt am Main Fachbereich 15: Biologie und Informatik Junior Prof. Dr. Dirk Metzler n 4m[ z / 2 z ] ( 2 2 g 2 g Single Label MAs ) m n totale Anzahl an benötigten Microarray s m Anzahl technisch er Replikate pro Sample Distanz der Klassendur chschnitte z/2 falsch positv Entdeckte z falsch negativ Entdeckte g2 biologisch e Varianz g2 technisch e Varianz Sebastian Bremm 12 Johann Wolfgang Goethe Universität Frankfurt am Main Fachbereich 15: Biologie und Informatik Junior Prof. Dr. Dirk Metzler Single Label MAs n / m 4[ z / 2 z ] ( 2 2 g 2 g ) m n / m Anzahl der biologisch unterschie dlichen Samples Sebastian Bremm 13 Johann Wolfgang Goethe Universität Frankfurt am Main Fachbereich 15: Biologie und Informatik Junior Prof. Dr. Dirk Metzler Dual Label Arrays • cDNA MAs • Targets von 2 Zellen. Dies erleichtert einen direkten Vergleich • Teilweise geringe Spotdichte Sebastian Bremm 14 Johann Wolfgang Goethe Universität Frankfurt am Main Fachbereich 15: Biologie und Informatik Junior Prof. Dr. Dirk Metzler Dual Label MAs • Log (Ygadvfs) = Gg + GAga + GDgd + GVgv+ (GF)gf(v) + gadvfs – – – – – – Gg = Genexpression von g in der Population GAga = Spot auf Array GDgd = Effekt des Färbemittels GVgv = Effekt der Klasse oder des Typs (GF)gf(v) = individueller Effekt 2 gadvfs = unabh. Fehler mit Normal(0, x ) Sebastian Bremm 15 Johann Wolfgang Goethe Universität Frankfurt am Main Fachbereich 15: Biologie und Informatik Junior Prof. Dr. Dirk Metzler Dual Label MAs • Referenz Design: A1 A2 B1 B2 R R R R Sebastian Bremm 16 Johann Wolfgang Goethe Universität Frankfurt am Main Fachbereich 15: Biologie und Informatik Junior Prof. Dr. Dirk Metzler Dual Label MAs • Reference Design n 4[ z / 2 z ]2 ( g2 2 g2 ) g2 2 g2 Varianz innerhalb einer der Klassen. Muss aus vorherigen Daten geschätzt werden. Sebastian Bremm 17 Johann Wolfgang Goethe Universität Frankfurt am Main Fachbereich 15: Biologie und Informatik Junior Prof. Dr. Dirk Metzler Dual Label MAs • Design mit technischen Replikaten und Dye Swap: n 4m[ z / 2 z ] ( 2 2 g 2 m 2 g ) Sebastian Bremm 18 Johann Wolfgang Goethe Universität Frankfurt am Main Fachbereich 15: Biologie und Informatik Junior Prof. Dr. Dirk Metzler Dual Label MAs Varianz Verhältnis technische Replikate / Sample Anzahl benötigter Arrays Anzahl benötigter Samples 2 1 2 3 4 1 2 3 4 49 74 99 124 49 82 114 148 49 37 33 31 49 41 38 37 4 α = 0,001 β = 0,05 δ=1 Sebastian Bremm 19 Johann Wolfgang Goethe Universität Frankfurt am Main Fachbereich 15: Biologie und Informatik Junior Prof. Dr. Dirk Metzler Dual Label Mas nm n1[ m( g2 / g2 ) 2 ( / ) 2 2 g 2 g ] n m ist die Anzahl der Benötigten Arrays bei m Replikaten im Verhältnis zur Anzahl n1 bei keinen tec hnischen Replikaten . Sebastian Bremm 20 Johann Wolfgang Goethe Universität Frankfurt am Main Fachbereich 15: Biologie und Informatik Junior Prof. Dr. Dirk Metzler Dual Label MAs • Block Design: A1 A2 A3 A4 B1 B2 B3 B4 Sebastian Bremm 21 Johann Wolfgang Goethe Universität Frankfurt am Main Fachbereich 15: Biologie und Informatik Junior Prof. Dr. Dirk Metzler Dual Label MAs • Balanced Block Design – Leichter Vergleich von 2 Klassen. – Weniger Arrays benötigt. – unflexibel n ( z / 2 z ) 2 2 ( g2,1 g2, 2 2 g2 ) Sebastian Bremm 22 Johann Wolfgang Goethe Universität Frankfurt am Main Fachbereich 15: Biologie und Informatik Junior Prof. Dr. Dirk Metzler Dual Label MAs • Single Paired Design – Natürliche biologische Paarungen ( z.B. von Individuum vor und nach einer Behandlung). – Je eine Seite mit einer Farbe pro Probe. nbalanced ( z / 2 z ) 2 (2 g2 g2 ) 2 – η an Stelle von τ. Varianz des veränderten Gewebes (z.B. Tumor) zum normalen Gewebe. Sebastian Bremm 23 Johann Wolfgang Goethe Universität Frankfurt am Main Fachbereich 15: Biologie und Informatik Junior Prof. Dr. Dirk Metzler Dual Label MAs • Dye-Swap Paired Design – Die gleichen Targets wie Single Paired Design. – Die gleichen Arrays werden nocheinmal mit dem jeweils anderen Fluoreszensstoff ausgewertet. 2 ndyeswap ( z / 2 z ) 2 (2 g2 g2 ) Sebastian Bremm 24 Johann Wolfgang Goethe Universität Frankfurt am Main Fachbereich 15: Biologie und Informatik Junior Prof. Dr. Dirk Metzler Prognostic Markers • Finden von Genen oder Genklassen, die bei einer Krankheit exprimiert werden. Sebastian Bremm 25 Johann Wolfgang Goethe Universität Frankfurt am Main Fachbereich 15: Biologie und Informatik Junior Prof. Dr. Dirk Metzler Prognostic Markers • Effekt von Pooling ( z / 2 z ) g2 2 g2 n 4m ( ) 2 k m k Anzahl an unabhängig en biologisch en Samples 2 • Mehr Samples weniger Arrays Sebastian Bremm 26 Johann Wolfgang Goethe Universität Frankfurt am Main Fachbereich 15: Biologie und Informatik Junior Prof. Dr. Dirk Metzler Prognostic Markers • α = Wahrscheinlichkeit für falsch positive Entdeckung. • 1-β = Wahrscheinlichkeit richtig positive Entdeckung. • Problem: Wie wählt man α? Sebastian Bremm 27 Johann Wolfgang Goethe Universität Frankfurt am Main Fachbereich 15: Biologie und Informatik Junior Prof. Dr. Dirk Metzler Prognostic Markers # FD FDR E[ ] # FD # TD # FD falsch Entdeckte # FD richtig Entdeckte E[#FD] α( 1 π)G E[#TD] (1 )G Anteil der anders exprimiert en Gene G Anzahl an Genen Sebastian Bremm 28 Johann Wolfgang Goethe Universität Frankfurt am Main Fachbereich 15: Biologie und Informatik Junior Prof. Dr. Dirk Metzler Prognostic Markers Sebastian Bremm 29 Johann Wolfgang Goethe Universität Frankfurt am Main Fachbereich 15: Biologie und Informatik Junior Prof. Dr. Dirk Metzler Prognostic Markers Sebastian Bremm 30 Johann Wolfgang Goethe Universität Frankfurt am Main Fachbereich 15: Biologie und Informatik Junior Prof. Dr. Dirk Metzler Training eines Classifiers • Ein Classifier soll Gene als Prognostic Marker erkennen. Sebastian Bremm 31 Johann Wolfgang Goethe Universität Frankfurt am Main Fachbereich 15: Biologie und Informatik Junior Prof. Dr. Dirk Metzler Training eines Classifiers • Bedingung: Solle wenige Samples brauchen • Lösung: Sequentielle Bestimmung • Vorteile – Lernt durch eigene Erfahrung. – Stopp-Kriterium wird bei jedem Schritt überprüft. – Erzielen der gewünschten Signifikanz garantiert – Mit jeder Klassifikationsmethode einsetzbar. Sebastian Bremm 32 Johann Wolfgang Goethe Universität Frankfurt am Main Fachbereich 15: Biologie und Informatik Junior Prof. Dr. Dirk Metzler Training eines Classifiers 2 ^ z1 k N N min , N 0 N 1 N Qi i 1 ^ kN 0 N 0 N 1 Qi i 1 Sebastian Bremm 33