Industrie-Applikationen
Werbung
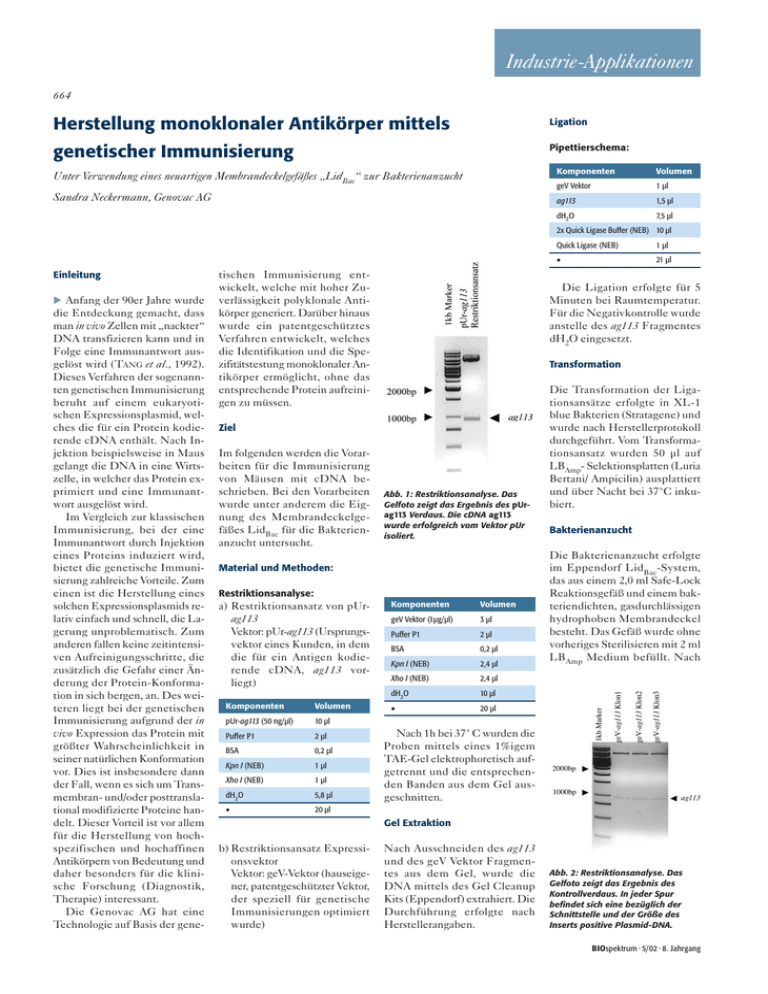
Industrie-Applikationen 664 Herstellung monoklonaler Antikörper mittels Ligation genetischer Immunisierung Pipettierschema: Komponenten Unter Verwendung eines neuartigen Membrandeckelgefäßes „LidBac“ zur Bakterienanzucht Sandra Neckermann, Genovac AG Volumen geV Vektor 1 µl ag113 1,5 µl dH2O 7,5 µl 2x Quick Ligase Buffer (NEB) 10 µl Einleitung Anfang der 90er Jahre wurde die Entdeckung gemacht, dass man in vivo Zellen mit „nackter“ DNA transfizieren kann und in Folge eine Immunantwort ausgelöst wird (TANG et al., 1992). Dieses Verfahren der sogenannten genetischen Immunisierung beruht auf einem eukaryotischen Expressionsplasmid, welches die für ein Protein kodierende cDNA enthält. Nach Injektion beispielsweise in Maus gelangt die DNA in eine Wirtszelle, in welcher das Protein exprimiert und eine Immunantwort ausgelöst wird. Im Vergleich zur klassischen Immunisierung, bei der eine Immunantwort durch Injektion eines Proteins induziert wird, bietet die genetische Immunisierung zahlreiche Vorteile. Zum einen ist die Herstellung eines solchen Expressionsplasmids relativ einfach und schnell, die Lagerung unproblematisch. Zum anderen fallen keine zeitintensiven Aufreinigungsschritte, die zusätzlich die Gefahr einer Änderung der Protein-Konformation in sich bergen, an. Des weiteren liegt bei der genetischen Immunisierung aufgrund der in vivo Expression das Protein mit größter Wahrscheinlichkeit in seiner natürlichen Konformation vor. Dies ist insbesondere dann der Fall, wenn es sich um Transmembran- und/oder posttranslational modifizierte Proteine handelt. Dieser Vorteil ist vor allem für die Herstellung von hochspezifischen und hochaffinen Antikörpern von Bedeutung und daher besonders für die klinische Forschung (Diagnostik, Therapie) interessant. Die Genovac AG hat eine Technologie auf Basis der gene- tischen Immunisierung entwickelt, welche mit hoher Zuverlässigkeit polyklonale Antikörper generiert. Darüber hinaus wurde ein patentgeschütztes Verfahren entwickelt, welches die Identifikation und die Spezifitätstestung monoklonaler Antikörper ermöglicht, ohne das entsprechende Protein aufreinigen zu müssen. Abb. 1: Restriktionsanalyse. Das Gelfoto zeigt das Ergebnis des pUrag113 Verdaus. Die cDNA ag113 wurde erfolgreich vom Vektor pUr isoliert. Restriktionsanalyse: Komponenten Volumen pUr-ag113 (50 ng/µl) 10 µl Puffer P1 2 µl BSA 0,2 µl Kpn I (NEB) 1 µl Xho I (NEB) 1 µl dH2O 5,8 µl ∑ 20 µl 21 µl Transformation Material und Methoden: a) Restriktionsansatz von pUrag113 Vektor: pUr-ag113 (Ursprungsvektor eines Kunden, in dem die für ein Antigen kodierende cDNA, ag113 vorliegt) 1 µl ∑ Die Ligation erfolgte für 5 Minuten bei Raumtemperatur. Für die Negativkontrolle wurde anstelle des ag113 Fragmentes dH2O eingesetzt. Ziel Im folgenden werden die Vorarbeiten für die Immunisierung von Mäusen mit cDNA beschrieben. Bei den Vorarbeiten wurde unter anderem die Eignung des Membrandeckelgefäßes LidBac für die Bakterienanzucht untersucht. Quick Ligase (NEB) Komponenten Volumen geV Vektor (1µg/µl) 3 µl Puffer P1 2 µl BSA 0,2 µl Kpn I (NEB) 2,4 µl Xho I (NEB) 2,4 µl dH2O 10 µl ∑ 20 µl Die Transformation der Ligationsansätze erfolgte in XL-1 blue Bakterien (Stratagene) und wurde nach Herstellerprotokoll durchgeführt. Vom Transformationsansatz wurden 50 µl auf LBAmp- Selektionsplatten (Luria Bertani/ Ampicilin) ausplattiert und über Nacht bei 37°C inkubiert. Bakterienanzucht Die Bakterienanzucht erfolgte im Eppendorf LidBac-System, das aus einem 2,0 ml Safe-Lock Reaktionsgefäß und einem bakteriendichten, gasdurchlässigen hydrophoben Membrandeckel besteht. Das Gefäß wurde ohne vorheriges Sterilisieren mit 2 ml LBAmp Medium befüllt. Nach Nach 1h bei 37° C wurden die Proben mittels eines 1%igem TAE-Gel elektrophoretisch aufgetrennt und die entsprechenden Banden aus dem Gel ausgeschnitten. Gel Extraktion b) Restriktionsansatz Expressionsvektor Vektor: geV-Vektor (hauseigener, patentgeschützter Vektor, der speziell für genetische Immunisierungen optimiert wurde) Nach Ausschneiden des ag113 und des geV Vektor Fragmentes aus dem Gel, wurde die DNA mittels des Gel Cleanup Kits (Eppendorf) extrahiert. Die Durchführung erfolgte nach Herstellerangaben. Abb. 2: Restriktionsanalyse. Das Gelfoto zeigt das Ergebnis des Kontrollverdaus. In jeder Spur befindet sich eine bezüglich der Schnittstelle und der Größe des Inserts positive Plasmid-DNA. BIOspektrum · 5/02 · 8. Jahrgang Industrie-Applikationen 665 der Inokulation wurden die LidBac-Gefäße mit ihrem Membrandeckel verschlossen und im Thermomixer comfort (Eppendorf) bei 1.400 rpm und 37 °C über Nacht inkubiert. Plasmidpräperation Zur Isolierung der Plasmid DNA wurde der Perfectprep Plasmid Mini kit (Eppendorf) verwendet. Die Durchführung erfolgte nach Herstellerangaben. Sequenzierung Die Sequenzierreaktionen wurden von der Firma MWG-Biotech mit den geV spezifischen Primern, geV for und geV rev durchgeführt. Transfektion Die Transfektionen wurden in einer Säugerzelllinie durchgeführt. Es handelt sich dabei um eine semiadhärente Zelllinie, die als Monolayer am Boden spezieller Kulturflaschen wächst. Die Kultivierung erfolgte im Brutschrank bei 37°C und einer Atmosphäre von 10 % CO2-Gehalt. Die Säugerzellen wurden unter Verwendung des Transfektionsreagenz Lipofektamin (Invitrogen) nach Herstellerprotokoll in 6 Well-Zellkulturplatten (Greiner) transfiziert. Zellmedien: Dulbecco’s MEM (Biochrom AG) mit 10 % FCS (PAA Laboratories) und 1 % Penicillin/Streptamycin (Biochrom AG). Fluoreszenz-aktivierter Zellsortierer (FACScan-Analyse) Die transfizierten und abgelösten Säugerzellen wurden nach 24 Stunden in PBS (Gibco) mit 3 % FCS aufgenommen und auf eine Zellzahl von 20000 Zellen/µl eingestellt. Von dieser Zellsuspension wurden pro Probe je 10 µl in eine 96 Well-Mikrotiterplatte (Greiner) pipettiert, anschließend wurden 25 µl des ersten Antikörpers (c=30 µg/µl in PBS/3% FCS) in jedes Well dazugegeben. Nach 30 Minuten bei 4 °C wurden die Proben mit PBS/ 3% FCS gewaBIOspektrum · 5/02 · 8. Jahrgang Abb 3: FACScan-Analyse. Rot : irrelevanter Antikörper Grün: relevanter Antikörper Es zeigt sich eine deutliche Verschiebung der grünen zur roten Kurve, d.h. ag113 wird auf der Säugerzelllinie exprimiert. viele Kolonien zu identifizieren, wohingegen auf der Negativplatte keine Kolonien vorhanden waren. Diskussion 1.3 Kontroll-Restriktion und Sequenzanalyse Nach der Plasmidpräparation wurde ein Kontrollverdau mit den Enzymen der Klonierungsschnittstellen durchgeführt. Die Sequenzanalyse von geVag113 Klon 1 ergab, dass die cDNA keine Mutationen enthält und korrekt über die entsprechenden Schnittstellen in den geV Vektor kloniert wurde. Die ag113 DNA konnte erfolgreich ohne Mutationen in den geV Vektor kloniert werden. Für die Bakterienanzucht konnte das LidBac-System erfolgreich eingesetzt werden. 2. Expressionsanalyse von geVag113 in Säugerzellen schen. Hierfür wurden pro Well 200 µl PBS/ 3% FCS zugegeben und die Mikrotiterplatte bei 480 x g und 4°C für 5 Minuten zentrifugiert. Der Überstand wurde durch Ausschlagen entfernt. Anschließend wurden in jedes Well 25 µl des RPE markierten zweiten Antikörper (c=10 µg /µl in PBS/ 3% FCS) pipettiert. Nach einer Inkubationszeit von 30 Minuten bei 4°C wurde jede Probe wie oben beschrieben 2x gewaschen. Die Zellen wurden in 200 µl PBS 3% FCS resuspendiert und zum Messen in FACS-Röhrchen (Sarstedt) überführt. Ergebnisse 1. Klonierung einer Antigen kodierenden cDNA in den geV Vektor 1.1 Restriktionsverdau Die für ein Antigen kodierende cDNA (hier: ag 113), muss zunächst in den für genetische Immunisierungen optimierten geV-Vektor kloniert werden. Hierfür wurde die ag113 cDNA aus ihrem Ursprungsvektor pUrag113 mittels Verdau mit entsprechenden Restriktionsenzymen und elektrophoretischer Auftrennung isoliert. 1.2 Ligation und Transformation Der geV Vektor und die ag113 cDNA wurden nach den unter Material und Methoden aufgeführten Protokollen ligiert und transformiert. Auf der entsprechenden LBAmp-Platte waren Die Expressionsanalyse ist im Vorfeld der genetischen Immunisierung von großer Bedeutung. Im Verlauf der Immunisierung wird das Serum der Mäuse mehrere Male getestet. Dieser Serentest erfolgt mittels FACScanAnalyse auf Zellen, die mit der cDNA des Antigens transfiziert wurden. Ist das Serum positiv, werden die Maus B-Zellen mit SP210 Zellen fusioniert und anschließend die spezifischen Hybridome identifiziert. Ist das Serum negativ, wird die Immunisierung mit modifizierter Strategie weitergeführt. Aufgrund dieses Nachweisverfahrens wird vor dem Start einer Immunisierung die Exprimierbarkeit der Antigen-cDNA überprüft. Da die Exprimierbarkeit einer cDNA einen ganz entscheidenden Schritt im Verlauf des Produktionsprozesses darstellt, würde bei einem negativen Ergebnis eine Optimierung der Transfektion der Expression durchgeführt. Für die Expressionsanalyse von geV-ag113 wurden Säugerzellen chemisch transfiziert und die Expression im FACS analysiert. Abbildung 3 zeigt die sehr gute Exprimierbarkeit der geVag113 DNA. 1. Klonierung der ag113 DNA in den geV Vektor 2. Expressionsanalyse Es konnte gezeigt werden, dass sich die geV-ag113 DNA sehr gut in den verwendeten Säugerzellen exprimieren lässt (siehe Abb. 3). Daher sind keine weiteren Optimierungsschritte für eine verbesserte Transfektion/Expressionsstrategie nötig. Die DNA kann direkt zur genetischen Immunisierung eingesetzt werden. Literatur Tang DC, DeVit M, Johnston SA. Genetic immunization is a simple method for eliciting an immune response. Nature 1992; 356: 152–154 Korrespondenzadresse: Genovac AG Waltershofener Str. 17 D-79111 Freiburg Tel.: 0761/45636-0 [email protected] www.genovac.com











