Gram negative Resistenzkeime
Werbung
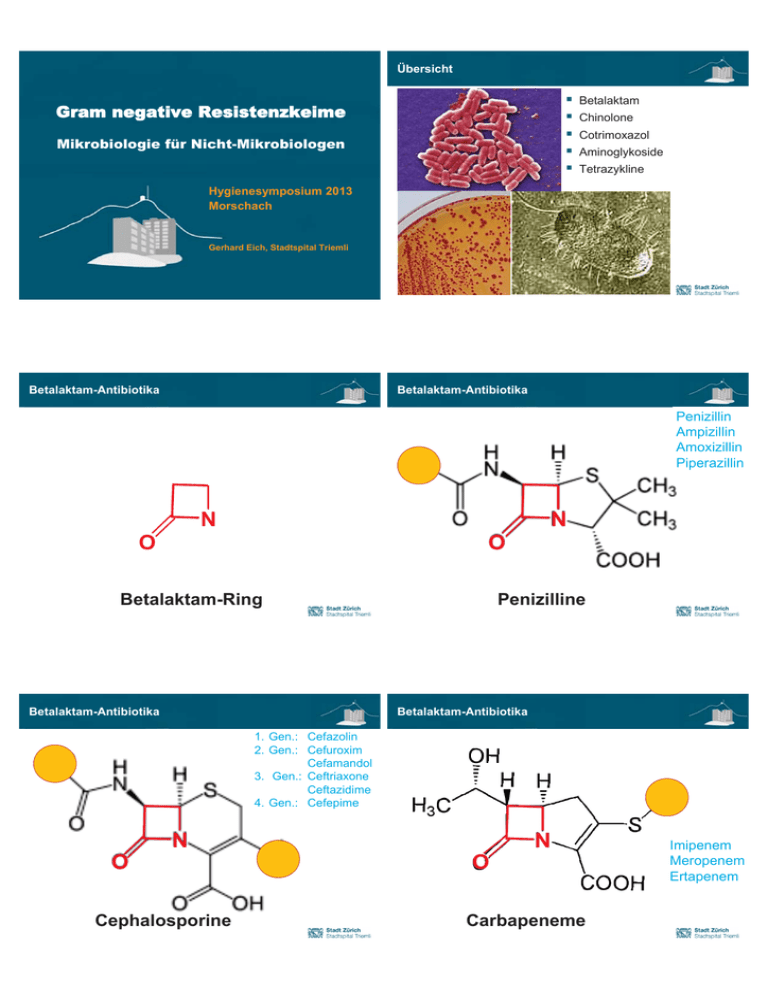
Übersicht Gram negative Resistenzkeime Mikrobiologie für Nicht-Mikrobiologen Betalaktam Chinolone Cotrimoxazol Aminoglykoside Tetrazykline Hygienesymposium 2013 Morschach Gerhard Eich, Stadtspital Triemli Betalaktam-Antibiotika Betalaktam-Antibiotika Penizillin Ampizillin Amoxizillin Piperazillin N N O O Betalaktam-Ring Betalaktam-Antibiotika Penizilline Betalaktam-Antibiotika 1. Gen.: Cefazolin 2. Gen.: Cefuroxim Cefamandol 3. Gen.: Ceftriaxone Ceftazidime 4. Gen.: Cefepime N O Cephalosporine N O Carbapeneme Imipenem Meropenem Ertapenem Betalaktam-Antibiotika: Wirkungsweise Ser O Betalaktam-Antibiotika: Wirkungsweise H zytoplasmatische Membran E-Laktam-Antibiotika: Analoga zu D-Ala-D-Ala R O O D-Alanin-D-Alanin O N O O H S O N O O N O N Ser O H S N N Penicillin R zytoplasmatische Membran O Resistenzmechanismen : Affinität zum PBP Betalaktam-Antibiotikum H Penicillin Binding Protein PBP R O O Resistenzmechanismen: Porine & Efflux-Pumpen Betalaktam-Antibiotikum erworben: Penizillin-R bei Pneumokokken Streptokokken Gram pos Bakterien Äussere Membran Zellwand Zellmembran intrinsisch: Cephalosporin-R bei Enterokokken PBP PBP` Gram neg Bakterien Resistenzmechanismen : Betalaktamasen Resistenzmechanismen gegen Betalaktamasen Betalaktamasen = Enzyme, die Betalaktam-Antibiotika zerstören Porine PBP H2O N O O O H Betalaktamase N H „klassische BLA der Enterobakterien Einteilung der Betalaktamasen TEM-1, TEM-2, SHV-1 Klassische Betalaktamasen Resistenzen ampC-Betalaktamasen Amoxizillin Extended-spectrum Betalaktamasen (ESBL) Amoxi/Clav 2 Gen Ceph Expression der Betalaktamase Chromosomale Betalaktamasen K1 (K.oxytoca) Eigenschaften: Carbapenemasen Gen auf einem Plasmid Hemmung durch BLA-Inhibitoren keine Wirkung gegen • 3. & 4. Gen Ceph (Ceftriaxone, Ceftazidime, Cefepime) • Carbapeneme 15 ampC-Betalaktamase Derepression von ampC Inaktiviert Cephalosporine 1.-3. Gen. und (Cephamyxine , zB Cefoxitin) nicht aktiv gegen Cefepime (4. Gen Ceph) und Carbapeneme Wichtig: Gen für ampC vorhanden bei • Enterobacter cloacae, aerogenes • Citobacter freundii • Serratia marcescens • Morganella morganii Zusätzliches Problem: • E.coli • Klebsiella spp. • Proteus spp. R ampC R R* ampC ampC auf Plasmiden bei: Mutation ampC: Selektion von Resistenz unter Therapie Resistenzmuster GNS Wildtyp Bei Wildtyp: keine Expression des Gens Æ Empfindlichkeit gegen 3. Gen. Ceph. I E. coli, P. mirabilis, Salmonella sp., Shigella sp. Mutation in 1 von 100‘000 Bakterien Æ permanente Produktion von ampC auf hohem Niveau (Hyperproduktion, Derepression) II Klebsiella sp. Citrobacter koseri Klassische Betalaktamase E. cloacae, E. aerogenes, ampC C. freundii, Serratia sp., Morganella morganii III Selektion von resistenten Bakterien vor allem bei Infektionen mit hoher Bakterienzahl • Intraabdominal • Osteomyelitis Risiko besteht vor allem bei: • Enterobacter cloacae, aerogenes • Citrobacter freundii Antibiogramm Antibiotikum Extended-spectrum Betalaktamasen ESBL I II III III TEM1,2 SHV1 ampC ampC derep. Amoxizillin E R R R Amoxi/Clav E E R R Pip/Tazo E E E R Ceph 3. Gen E E E R Ceph 4. Gen E E E E-I Cephamyxin E E E R Carbapenem E E E E ESBL-Diagnostik empfindlich auf BetalaktamAntibiotika Screening: verminderte Empfindlichkeit gg 3. Gen Ceph Empfindlich gegen Cephamyxine (Cefoxitin) Bestätigung: Hemmung durch Inhibitoren • Champagner-Korken • ESBL-Etest Inaktivieren alle Penizilline und Cephalosporine Inaktiv gegen Carbapeneme (und Cephamyxine) 500 verschiedene ESBL beschrieben: • Mutationen von TEM oder SHV oder erworben von anderen Bakterien • Bezeichnungen: TEM-x; SHV-x; OXA-x; CTX-M-x ¾ CTX-M-15 ist der häufigste Typ in der CH • Unterschiedliche Aktivität gegen die verschiedenen Cephalosporine Gen auf einem Plasmid Æ leichte Verbreitung auf andere Bakterien Hemmung durch Betalaktamase-Inhibitoren (zB Clavulansäure) ESBL: Champagner-Korken ESBL-Etest Antibiogramm ESBL: Wildtyp-Antibiogramm + ESBL 12 1.0 Probleme der Diagnostik von Betalaktamasen ESBL-Produktion auf niedrigem Niveau Kombination von Resistenzmechanismen: • ampC + ESBL • ESBL + Effluxpumpen Æ R gg Ertapenem Ähnliche Resistenzphänomene • ESBL • Dereprimierte ampC • Überproduktion von chromosomalen Betalaktamasen: K1 Æ K. oxytoca Chromosomale Betalaktamase K1 (K. oxytoca) Antibiotikum K1 High-level Amoxizillin R Amoxi/Clav R Pip/Tazo R Antibiotikum I od II ESBL ampC ampC + ESBL ampC derep. ampC derep. + ESBL Amoxizillin E/R R R R R R Amoxi/Clav E E-R R R R R Pip/Tazo E E-R E R R R Ceph 3. Gen E E-R E R R R Ceph 4. Gen E E-R E I-R E-I E-R Cephamyxin E E E R R R Carbapenem E E E E E E Antibiogramm ESBL: Wildtyp-Antibiogramm + ESBL Antibiotikum I II I + II ESBL ampC ampC + ESBL ampC derep. ampC derep. + ESBL Amoxizillin E R R R R R R Amoxi/Clav E E E-R R R R R Pip/Tazo E E E-R E R R R Ceph 3. Gen E E E-R E R R R Ceph 4. Gen E E E-R E I-R E-I E-R Cephamyxin E E E R R R R Carbapenem E E E E E E E Carbapenemasen Hydrolysieren Carbapeneme Verschiedene Untergruppen Natürliches Vorkommen: Ceph 3. Gen Ceftriaxone R Ceftazidime E Ceph 4. Gen E Carbapenem E • Gen auf Chromosomen • B. cereus, S.maltophilia, Aeromonas spp. Seit 80er Jahren auftreten von neuen Carbapenemasen: • z.T. auf Plasmiden • Vorkommen in Ps. aeruginosa & Enterobacteriaceae • 3 verschiedene Klassen Klasse A Serine Carbapenemase Metallo-Carbapenemase Vorkommen (weltweit): Vorkommen (v.a. Asien): MHKImipenem d4Pg/ml Untergruppen MHKImipenem: Eigenschaften: Zn++ an aktivem Zentrum • E. cloacae, S. marcescens, Klebsiella spp. • Chromosomal: SME, NMC, IMI • Plasmid: KPC, GES • Inhibition durch EDTA • Inhibition durch Clavulan-Säure • Hydrolyse von Aztreonam Summary Betalaktamasen bei GNS Enterobakteriaceae: • Bekannt: ¾ Klassisch TEM-1, TEM-2, SHV-1 ¾ ampC ¾ ESBL ¾ K1 (K.oxytoca • Neu: Carbapenemasen: ¾ Serine-Carbapenemasen (zB KPC) ¾ Metallo-Betalaktamasen Pseudomonas aeruginosa: A. baumanii: • Bekannt: Metallo-Betalaktamasen • Neu: KPC • Neu: Metallo-Betalaktamasen • Ursprünglich: B.cereus, Aeromonas spp., S. malt. • Neu: Ps. aeruginsa & Enterobacteriaceae • In Ps. aeruginosa: d8Pg/ml bis >128Pg/ml • In Enterobacteriaceae: 0.5Pg/ml Eigenschaften: • Inhibition durch EDTA • Inhibition durch Clavulan-Säure • Hydrolyse von Aztreonam











