Entwicklung einer neuen Technikwissenschaft
Werbung

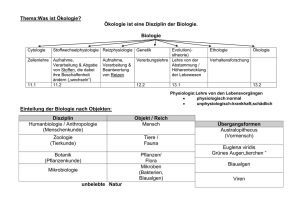
PdN BIOLOGIE in der Schule / BIOTECHNOLOGIE – SYNTHETISCHE BIOLOGIE HEFT 3 / 64. JAHRGANG / 2015 Entwicklung einer neuen Technikwissenschaft Von der Molekulargenetik zur Synthetischen Biologie* A. Pühler In diesem Beitrag wird das Forschungsgebiet Synthetische Biologie als neue Technikwissenschaft vorgestellt. Die Wurzeln der Synthetischen Biologie sowie Grundprinzipien und Einsatzfelder werden beschrieben. Die Bedeutung der Synthetischen Biologie für die zukünftige Biotechnologie wird herausgearbeitet. Stichwörter: Synthetische Biologie, Biotechnologie, Technikwissenschaft, Gentechnik 1 Einleitung – Das Forschungsgebiet Synthetische Biologie Der vorliegende Artikel beschäftigt sich mit der Entwicklung einer neuen Technikwissenschaft, die auf Elementen der Synthetischen Biologie aufbaut. Zunächst ist vorgesehen, den Begriff „Synthetische Biologie“ zu erläutern und deren Wurzeln zu benennen. Ein besonderes Augenmerk wird dann auf die mediale Geburtsstunde der Synthetischen Biologie gerichtet, bevor einige ausgewählte Aspekte präsentiert werden. Dabei wird zunächst als Erfolg der Synthetischen Biologie die Produktion des Antimalariamittels Artemisinin beschrieben. Anschließend wird auf allgemeine Grundprinzipien und Vorgehensweisen der Synthetischen Biologie eingegangen. Weitere Aspekte betreffen dann die Einbeziehung der Synthetischen Biologie in die universitäre Lehre sowie die Erfolge Bielefelder Studierender im Rahmen des weltweiten sogenannten iGEM-Wettbewerbs. Schließlich wird auch der Frage nachgegangen, ob das deutsche Gentechnikgesetz zur Überwachung der Aktivitäten auf dem Gebiet der Synthetischen Biologie geeignet ist. Das Herzstück des Artikels behandelt dann die Synthetische Biologie als neue Technikwissenschaft, wobei besonders ihre Bedeutung für eine zukünftige Biotechnologie herausgearbeitet werden soll. 2 Was versteckt sich nun hinter dem Begriff „Synthetische Biologie“? Leider fehlt bis heute eine prägnante Definition. Ein brauchbarer Ansatz ist in einer Studie mit dem Titel „Synthetische Biologie“ enthalten, die im Juli 2009 von den drei Organisationen Deutsche Akademie 4 der Technikwissenschaften, Nationale Akademie der Wissenschaften Leopoldina und der Deutschen Forschungsgemeinschaft herausgegeben wurde [1]. Diese Studie verfolgt den Zweck, das Forschungsgebiet Synthetische Biologie möglichst umfassend der Öffentlichkeit vorzustellen und der Politik Empfehlungen für den Umgang mit diesem Forschungsgebiet zu geben. In der genannten Studie wird Synthetische Biologie nun wie folgt definiert: Die Synthetische Biologie basiert auf den Erkenntnissen der molekularen Biologie, der Entschlüsselung kompletter Genome, der ganzheitlichen Betrachtung biologischer Systeme und dem technologischen Fortschritt bei der Synthese und Analyse von Nukleinsäuren. Sie führt ein weites Spektrum an naturwissenschaftlichen Disziplinen zusammen und verfolgt da- bei ingenieurwissenschaftliche Prinzipien. Das spezifische Merkmal der Synthetischen Biologie ist, dass sie biologische Systeme wesentlich verändert und gegebenenfalls mit chemisch synthetisierten Komponenten zu neuen Einheiten kombiniert. Dabei können Eigenschaften entstehen, wie sie in natürlich vorkommenden Organismen bisher nicht bekannt sind. Diese Definition greift also die Wurzeln der Synthetischen Biologie auf, weist auf den Einsatz von ingenieurwissenschaftlichen Prinzipien hin und endet mit der Feststellung, dass biologische Systeme mit neuen Eigenschaften konstruiert werden können, die so in der Natur nicht vorkommen. Damit sind die Kernpunkte der Synthetischen Biologie genannt: Synthetische Biologie nutzt Eigenschaften natürlich vorkommender Zellen, führt diese in geeigneten Wirtssystemen mittels geno- Abb. 1: Bilder von DNA-Sequenzierautomaten sowie eines Ausdrucks des Kapillarsequenzierers. Dargestellt sind drei unterschiedliche Sequenzierautomaten. Das Gerät von Applied Biosystems gehört der ersten Sequenziergeneration an. In der unteren Bildhälfte ist der Ausdruck eines Sequenzierlaufs dieses Geräts wiedergegeben. Die Interpretation der vier eingefärbten Kurven führt zur Festlegung der Basensequenz. Im weiteren werden noch 2 weitere Sequenierautomaten der zweiten Sequenziergeneration gezeigt. BIOTECHNOLOGIE – SYNTHETISCHE BIOLOGIE / PdN BIOLOGIE in der Schule HEFT 3 / 64. JAHRGANG / 2015 mischer Programmierung zusammen und konstruiert so neuartige Zellen. Start DMTO O B1 O 3 Die Wurzeln der Synthetischen Biologie Die Aufklärung der DNA-Struktur durch Watson und Crick im Jahre 1953 lässt sich als eine der frühesten Wurzeln der Synthetischen Biologie identifizieren [2]. Die damals postulierte DNA-Doppelhelix hat die Molekulargenetik begründet und viele Anstöße zu bahnbrechenden Erkenntnissen geliefert. So wurde der genetische Code in den 60er Jahren des letzten Jahrhunderts komplett aufgeklärt [3]. Dieser grundsätzlich für alle Lebewesen geltende genetische Code gibt Auskunft darüber, wie der Bauplan von Proteinen als genetische Information im DNA-Molekül gespeichert wird. Dazu liegen molekularbiologisch folgende Erkenntnisse vor. Proteine bestehen aus einer linearen Kette von 20 unterschiedlichen Aminosäuren. DNA-Einzelstränge hingegen stellen eine lineare Kette von vier unterschiedlichen Nukleotiden dar. Der genetische Code gibt nun Auskunft darüber, wie jede einzelne Aminosäure in den Proteinen durch Basentripletts genetisch kodiert wird. Kurz zusammengefasst bedeutet dies, dass die Abfolge von Aminosäuren in Proteinen durch die Abfolge der Basentripletts in den Einzelsträngen der DNA-Doppelhelix niedergelegt ist. Ein Durchbruch auf dem Weg zur Synthetischen Biologie ist dann mit der Entwicklung der Gentechnik verbunden. Diese Entwicklung wurde im Jahr 1973 zunächst bei Bakterien als Plasmid-basierte Technologie in Californien, USA, aus der Taufe gehoben [4]. Bereits die ersten Publikationen legten die Leistungsfähigkeit dieser Technologie offen, da Erbinformationen aus beliebigen Organismen in Bakterien eingeführt und dort stabil vererbt werden können. Kurz darauf konnten schon neben Bakterien auch eukaryotische Mikroorganismen wie Hefen und Pilze und schließlich auch mehrzellige Organismen aus dem Pflanzen- und Tierreich gentechnisch bearbeitet werden. Die Etablierung der Gentechnik, die praktisch Gentransfer zwischen beliebigen Organismen ermöglicht, löste eine weitreichende gesellschaftliche Diskussion über deren Gefahrenpotenziale aus. Um mögliche Gefahren durch Gentechnik von Mensch und Umwelt abzuwenden, wurden Richtlinien zum Umgang mit gentechnisch veränderten Organismen entwickelt, die in Deutschland Eingang in das Gentechnikgesetz fanden [5]. DMTO O O B2 O O NC O P N Detritylierung HO O DMTO Step 1 Aktivierung und Kopplung B1 O O O B2 O NC O P O O O O O O nächster Zyklus B1 Step 2 Schneiden O O O B1 O O O Step 3 Oxidation HO Abspaltung des fertigen Oligonukleotids HO O NC B2 DMTO O O O O O P O O O O O P O B2 O NC O O B1 Step 4 Detritylierung B2 O NC O P O O O O B1 B1 O O O Cl HO Cl OH O Cl Entfernung des Schutzes HO O B2 O O P O O O B1 HO Abb. 2: Ablauf der chemischen DNA-Synthese. Die chemische DNA-Synthese besteht aus den vier gezeigten Schritten. Pro Zyklus verlängern diese vier Syntheseschritte einen gegebenen DNA-Strang um ein Nukleotid. Der Prozess kann automatisiert werden und liefert DNA-Fragmente von bis zu 100 Nukleotiden Länge. Als eine weitere Wurzel der Synthetischen Biologie etablierte sich anschließend die DNA-Sequenzierung. Nachdem der genetische Code entschlüsselt vorlag, wurde intensiv an Verfahren zur Sequenzierung von DNA-Einzelsträngen gearbeitet. Im Jahr 1977 konnten schließlich zwei Sequenziertechnologien in der Zeitschrift PNAS vorgestellt werden, nämlich die chemische Degradationsmethode von Maxam und Gilbert [6] und die Kettenabbruchmethode von Sanger et al. [7]. Da die Sanger-Methode automatisiert werden konnte, setzte sich diese schließlich durch und führte zur Konstruktion von Sequenzierautomaten, mit denen zunächst kleinere Genome von Bakterien, dann aber auch größere Genome wie das menschliche Genom, sequenziert werden konnten. In der Zwischenzeit haben sich Sequenziergeräte der zweiten Generation durchgesetzt, die hoch parallel viele DNAEinzelstränge gleichzeitig bearbeiten können, wodurch das Sequenziergeschehen eine ungeahnte Beschleunigung erfuhr. Häufig verwendete Sequenzierautomaten der ersten und zweiten Generation werden in Abbildung 1 präsentiert. Als eine weitere Technologie, die für die Synthetische Biologie von großer Bedeutung ist, muss die chemische DNA-Synthese genannt werden [8]. In biologischen Systemen gilt die Molekularbiologie der DNA-Synthese als aufgeklärt. An einem vorhandenen DNA-Einzelstrang als Matrize wird der komplementäre DNA-Strang enzymatisch synthetisiert. Diese biologische DNA-Replikation kann bei der chemischen DNA-Synthese allerdings nicht genutzt werden, denn hier liegt als Blaupause eine vorgegebene Basensequenz vor, die bei der Synthese eines DNA-Einzelstrangs verwirklicht werden muss. Die chemische DNA-Synthese beruht daher auf einem komplett neuen Ansatz, der sich insbesondere der Methoden der Schutzgruppenchemie bedient (Abb. 2). Die Technologie ließ sich automatisieren, so dass heute mit entsprechenden Maschinen DNA-Einzelstränge bis zu einer Länge von rund 100 Basen hergestellt werden können. 5 PdN BIOLOGIE in der Schule / BIOTECHNOLOGIE – SYNTHETISCHE BIOLOGIE Abb. 3: Herstellung einer synthetischen Mycoplasma-mycoides-Zelle durch Einführung des chemisch synthetisierten Mycoplasma-mycoides-Genoms nach Mycoplasma capricolum (verändert nach [17]). Die geschilderten Erkenntnisse und Technologien spielen bei der Entwicklung der Synthetischen Biologie die entscheidende Rolle und werden deshalb als Wurzeln dieses Forschungsfeldes betrachtet. Wie die Synthetische Biologie schließlich in das Bewusstsein der Öffentlichkeit gelangte, wird im nächsten Kapitel geschildert. 4 Die mediale Geburtsstunde der Synthetischen Biologie Als mediale Geburtsstunde der Synthetischen Biologie wird allgemein das Frühjahr 2010 gesehen. Anlass dazu war eine Publikation der Arbeitsgruppe um Craig Venter in der Zeitschrift Science, die die Konstruktion einer bakteriellen Zelle beschreibt, die von einem chemisch synthetisierten Genom gesteuert wird [9]. Craig Venter gelang mit dieser Publikation ein weiterer Paukenschlag. Er wurde ja ursprünglich durch seinen unkonventio6 nellen DNA-Sequenzierungsansatz, die menschliche Genomsequenz durch Parallelschaltung von vielen Sequenziergeräten zu entschlüsseln, international bekannt [10]. In einem zweiten Großprojekt widmete er sich der Analyse mikrobieller Gemeinschaften aus dem marinen Bereich, was erneut den Einsatz von Sequenzierautomaten im industriellen Maßstab erforderte [11]. Das jüngste Großprojekt beinhaltet nun die chemische Synthese eines gesamten bakteriellen Genoms. Er wählte dazu das tierpathogene Bakterium Mycoplasma mycoides, das als Erreger der Lungenseuche bekannt ist. Dieses Bakterium bot sich aufgrund einer geringen Genomgröße von 1,08 Millionen Basenpaaren für eine chemische Gesamtsynthese an. Die Sequenz des Mycoplasma-mycoidesGenoms war bereits bekannt, sodass ein Ablaufplan zur chemischen Synthese dieses Genoms entworfen werden konnte. Der Umfang dieser Gesamtsynthese war HEFT 3 / 64. JAHRGANG / 2015 enorm, denn es musste aus DNA-Fragmenten mit einer Größe von rund 100 Basen das über 1 Million Basenpaare große Gesamtgenom zusammengesetzt werden. Diese Aufgabe meisterte die CraigVenter-Gruppe, wobei das chemisch synthetisierte bakterielle Genom schrittweise als künstliches Chromosom in einer Hefezelle aufgebaut wurde. Mit der Fertigstellung des chemisch synthetisierten Mycoplasma-mycoidesGenoms war zunächst aber nur der erste Schritt getan. Das chemisch synthetisierte Genom musste noch in eine Bakterienzelle transplantiert und dort zum Arbeiten gebracht werden. Auch dieser Schritt verlangte Entwicklungsarbeit, denn es gab dazu noch kein etabliertes Verfahren. Die Craig-Venter-Gruppe wählte für den Transplantationsschritt das Bakterium Mycoides capricolum. Die Transplantation verlief erfolgreich. Das chemisch synthetisierte Mycoplasma-mycoides-Genom etablierte sich in der Mycoplasma-capricolum-Zelle und wandelte diese in eine Mycoplasmamycoides-Zelle um. Die Sensation war damit perfekt. Die Craig-Venter-Gruppe hat mit diesem Verfahren Geschichte geschrieben und die erste Bakterienzelle mit einem chemisch synthetisierten Genom realisiert. Als Ergebnis liegt eine synthetische Zelle vor, die von einem chemisch synthetisierten Genom gesteuert wird. Weltweit wurde dieses Ergebnis von den Medien als großer Durchbruch gefeiert und die Craig-Venter-Publikation als die Geburtsstunde der Synthetischen Biologie bezeichnet. Ohne die Bedeutung dieses Erfolgs herunterzuspielen, ist anzumerken, dass die Craig-Venter-Gruppe nur einen Ausschnitt aus dem Gesamtansatz der Synthetischen Biologie bearbeitet hat. Sie hat gezeigt, dass die chemische DNASynthese ein wertvolles Werkzeug bei der Herstellung von DNA-Fragmenten darstellt, die nach Transfer in eine geeignete Zelle biologisch aktiv werden können. In Abbildung 3 wird das bahnbrechende Experiment der Craig-Venter-Gruppe in seinen Grundzügen dargestellt. Ein weiterer Kommentar soll den medialen Hype um die Craig-Venter-Publikation beleuchten. Craig Venter trug dazu nicht unwesentlich bei, denn die Überschrift der Publikation „Creation of a Bacterial Cell Controlled by a Chemically Synthesized Genome“ sprach von der Erschaffung einer synthetischen Zelle. Diese Wortwahl ist jedoch unangebracht, denn es handelt sich nicht um die Erschaffung einer synthetischen Zelle, sondern lediglich um den Nachbau einer in der Natur BIOTECHNOLOGIE – SYNTHETISCHE BIOLOGIE / PdN BIOLOGIE in der Schule HEFT 3 / 64. JAHRGANG / 2015 vorkommenden bakteriellen Spezies. Die Craig-Venter-Gruppe nutzte für die chemische Synthese des bakteriellen Genoms die Genomsequenz von Mycoplasma mycoides und anschließend für den Transplantationsschritt das Empfängerbakterium Mycoplasma capricolum. Damit wurde auf in der Natur vorhandene Informationen und Strukturen zurückgegriffen, also Vorhandenes nachgebaut und nicht etwas Neues erschaffen [12]. Mit dieser Bewertung des Craig-Venter-Experiments nähert man sich der Frage, wie sich Synthetische Biologie und Künstliches Leben voneinander abgrenzen. Synthetische Biologie konstruiert lebende Systeme unter Verwendung von biologischen Komponenten. Künstliches Leben hingegen erschafft Systeme, die Grundeigenschaften des Lebens aufweisen, ohne dass dazu biologische Komponenten verwendet werden. Nach dem heutigen Stand der Wissenschaft liegt die Erschaffung von künstlichem Leben jenseits aller Vorstellungskraft. Die Begriffe Synthetische Biologie und Künstliches Leben grenzen sich also scharf voneinander ab und sollten im allgemeinen Sprachgebrauch nicht vermengt werden. 5 Aspekte der Synthetischen Biologie Heutzutage wird Synthetische Biologie oft mit einer komplexeren Gentech- nik gleichgesetzt. Man transferiert nicht mehr nur einzelne Gene, sondern konzentriert sich auf größere Gencluster. Solche Gencluster können für synthetische Stoffwechselwege kodieren, aber auch als synthetische Genschalter aktiv sein. Das bekannteste Beispiel für synthetische Stoffwechselwege stellt die Biosynthese der Antimalaria-Substanz Artemisinin dar. Bis heute wird Artemisinin auf klassische Weise durch Extraktion aus getrockneten Pflanzen des einjährigen Gelben Beifuß gewonnen. Allerdings ist die ausreichende landwirtschaftliche Produktion von Pflanzenmaterial nicht gewährleistet, so dass ein Projekt ins Leben gerufen wurde, Artemisinin mit Hilfe der Synthetischen Biologie zu produzieren. Dazu soll das Darmbakterium Escherichia coli oder die Hefe Saccharomyces cerevisiae mit einem synthetischen Gencluser ausgerüstet werden, das die Biosynthese von Artemisinin vermittelt [13, 14]. Die Arbeiten sind bereits weit gediehen. Die synthetischen Mikroorganismen werden in Fermenterkulturen gezogen und produzieren die medizinisch wertvolle Substanz Artemisinin. Dieses prominente Projekt wurde übrigens von einer Reihe von interessanten Kooperationspartnern vorangetrieben. Neben der Nichtregierungsorganisation „One World Health“ und der Bill-Gates- Mevalonatweg (Anfang) Acetylcholin atoB HMGS IHMGR Mevalonatweg (Ende) Mervalonat MK PMK MPD iDi Stiftung sind zwei Industrieunternehmen, nämlich Amyris und Sanofi-Aventis an der Durchführung beteiligt. Dieses Projekt zeigt beispielhaft, welche Rolle die Synthetische Biologie in Zukunft auf dem biotechnologischen Sektor spielen kann. Der Stoffwechselweg zur Herstellung von Artemisinin mittels Synthetischer Biologie wird in Abbildung 4 präsentiert. Ein weiterer Aspekt betrifft die Entwicklung von Minimalzellen, die ein sogenanntes Minimalgenom enthalten. Das Minimalgenom soll per Definition nur essentielle Gene enthalten, die eine Zelle für das Überleben unter vorgegebenen Fermentationsbedingungen benötigt. Damit können aus einem Genom all die Gene entfernt werden, die einer Zelle das Überleben in einer komplexen Umwelt ermöglichen. Eine Minimalzelle mit einem reduzierten Genom ist also nur an definierte Fermentationsbedingungen angepasst. Man kann eine solche Minimalzelle auch als Chassis nutzen, das mit Genkassetten beladen gewünschte Substanzen produzieren kann. Minimalzellen spielen übrigens bei biologischen Sicherheitsfragen eine entscheidende Rolle. Zu den nicht essentiellen Genen eines Mikroorganismus zählen z. B. alle Gene, die dazu beitragen, Stresssituationen in einer komplexen Umwelt zu bewältigen. Werden nun Synthase iapA FPP ADS Amorphadien HydroxylaseEinheit CPR p450 Artemisinsäure H O H3C H3C SCoA HOOC OH H OPP OH H H H O O Artemisinsäure O O H H O O Artemisin Abb. 4: Darstellung des Biosynthesewegs zur Herstellung des Antimalariamittels Artemisinin. Der Biosyntheseweg zur Herstellung von Artemisininsäure geht von Acetyl-CoA aus. Zunächst wird der Mevalonat-Stoffwechselweg der Bierhefe Saccharomyces cerevisiae genutzt, gefolgt von drei Biosyntheseschritten aus der Pflanze Artemisia annua. Artemisininsäure wird dann chemisch zu Artemisinin umgewandelt. 7 PdN BIOLOGIE in der Schule / BIOTECHNOLOGIE – SYNTHETISCHE BIOLOGIE diese Stress-Gene aus dem mikrobiellen Genom entfernt, dann ist ein solcher Mikroorganismus für ein Überleben in einer komplexen Umwelt wenig geeignet. Eine synthetische Zelle, die als Ausgangspunkt eine Minimalzelle nutzt, besitzt damit ein intrinsisches Sicherheitssystem. Die Verwendung von Minimalzellen bei der Konstruktion von synthetischen Zellen ist aus Sicht der biologischen Sicherheit also ein wichtiges Argument. Synthetische Biologie als ein sich neu entwickelnder Wissenschaftszweig benötigt wissenschaftlichen Nachwuchs. Universitäten sind gefordert, vernünftige Ausbildungsprogramme ins Leben zu rufen. Ein Ansatzpunkt ist sicherlich die Einrichtung von Graduiertenschulen, die sich mit Fragen der Synthetischen Biologie beschäftigen. Solche Graduiertenschulen liefern gut ausgebildete Nachwuchswissenschaftler, die dann für Leitungsaufgaben in Akademia und Industrie zur Verfügung stehen. Man sollte aber bereits frühzeitig darüber nachdenken, wie man Synthetische Biologie auch in die universitäre Grundausbildung, also in Bachelorund Masterprogramme integrieren kann. Eine interessante Variante, Studierende an diesen neuen Wissenschaftszweig heran zu führen, wird mit dem iGEM-Wettbewerb realisiert. Beim iGEM-Wettbewerb (international genetically engineered machine competition) handelt es sich um den bedeutendsten internationalen Wettbewerb für Studierende auf dem Gebiet der Synthetischen Biologie [15]. Dieser Wettbewerb wird von dem berühmten Massachusetts Institute of Technology in Boston, USA, durchgeführt. Er wurde 2003 ins Leben gerufen und entwickelte eine ungeahnte Anziehungskraft. In der Zwischenzeit nehmen mehr als 200 studentische Gruppen aus allen Erdteilen an diesem Wettbewerb teil und ringen um eine der begehrten Goldmedaillen. Aufgabe des Wettbewerbs ist es, eine relevante Forschungsfrage mit Mitteln der Synthetischen Biologie zu lösen und dabei sogenannte Biobausteine (Biobricks) zu entwickeln, die von den iGEM-Organisatoren gesammelt und anderen iGEM-Teams wieder zur Verfügung gestellt werden. Die entwickelten Biobausteine funktionieren als synthetische Genschalter, kodieren aber auch für synthetische Stoffwechselwege. Ein Spezifikum des iGEM-Wettbewerbs ist es, dass die beteiligten Gruppen selbst großen Einsatz und viel Eigeninitiative entwickeln müssen. Diese Initiative beginnt bei der Auswahl des Forschungsthemas und dessen Bearbeitung und führt hin bis zur Finanzierung des Projekts und zur Kommunikation mit der Öffentlichkeit. Es ist verblüffend, mit welchem Enthusiasmus sich iGEM-Gruppen den vielfältigen Aufgaben stellen und bereits frühzeitig ein vertieftes Wissen auf dem Gebiet der Synthetischen Biologie erwerben. Am Centrum für Biotechnologie der Universität Bielefeld hat sich bereits im Jahr 2010 das erste iGEM-Team von Studierenden aus Masterstudiengängen formiert. Seitdem haben Bielefelder iGemTeams kontinuierlich an dem Wettbewerb teilgenommen. Als Themen wurde die Konstruktion von Biosensoren für Capsaicin und Bisphenol A aufgegriffen. Im Abb. 5: Das Bielefelder iGEM-Team des Jahres 2014 nach erfolgreicher Vorstellung ihres Projektes beim internationalen Wettbewerbs-Finale in Boston, USA. Die Stellwand nennt das im Wettbewerb vorgestellte Projekt „The Transformers – From Carbondioxide to Biofuel“. Foto: Justin Knight/iGEM Foundation 8 HEFT 3 / 64. JAHRGANG / 2015 Weiteren erfolgte die Entwicklung eines zellfreien Filtersystems für Mikrokontaminanten in Abwasser sowie der mikrobiellen Stromproduktion in einer Brennstoffzelle. Im Wettbewerb 2010 beteiligte sich das Bielefelder iGEM-Team mit dem Thema „Bakterielle Produktion von Biokraftstoff durch CO2-Fixierung“. Die Beiträge der Bielefelder iGEM-Teams wurden jedesmal prämiert. Besonders im Jahr 2013 wurde das Team in der Vorauswahl Europasieger und anschließend Vizeweltmeister. Abbildung 5 zeigt das Bielefelder iGEM-Team des Jahres 2014 mit einem Hinweis auf das behandelte Thema. Es wurde bereits darauf hingewiesen, dass gentechnische Verfahren in Deutschland dem Gentechnik-Gesetz unterliegen. Da die Synthetische Biologie als eine komplexere Gentechnik verstanden wird, muss die Frage beantwortet werden, ob das Gentechnikgesetz auch bei synthetisch erzeugten Organismen angewendet werden kann. Zur Beantwortung dieser Frage ist von Bedeutung, dass experimentell hergestellte synthetische Zellen zur Zeit ausnahmslos in großer Nähe zu ihren Ausgangszellen liegen, und dass dadurch das Prinzip der Risikoabschätzung greift. Das geltende Gentechnikgesetz kann daher für Fragen der biologischen Sicherheit von synthetischen Zellen herangezogen werden. Damit benötigt das Gebiet der Synthetischen Biologie augenblicklich keine neuen Regulierungsvorschriften. Diese Überlegungen begründen auch, warum in Deutschland von einem Moratorium, also von einem Forschungsverbot auf dem Gebiet der Synthetischen Biologie Abstand genommen wird. Diese Sichtweise wird auch in der eingangs erwähnten Studie zur Synthetischen Biologie vertreten. An Stelle eines Moratoriums wird in dieser Stellungnahme der Politik ein begleitendes Monitoring empfohlen. Die Politik hat diese Empfehlung umgesetzt und in der Zwischenzeit eine Monitoringstelle bei der Zentralen Kommission für Biologische Sicherheit eingerichtet [16]. Nicht verschwiegen werden soll, dass die Synthetische Biologie natürlich auch Potenziale beinhaltet, die bei der Entwicklung von biologischen Waffen genutzt werden können. Ein einfaches Szenario ist z. B. die chemische Synthese von pathogenen Virengenomen oder auch von Genen, die die Gefährlichkeit von pathogenen Mikroorganismen erhöhen. Da die chemische DNA-Synthese augenblicklich aber weltweit in Händen von einigen Firmen liegt, kann darauf geachtet werden, HEFT 3 / 64. JAHRGANG / 2015 BIOTECHNOLOGIE – SYNTHETISCHE BIOLOGIE / PdN BIOLOGIE in der Schule dass keine DNA-Fragmente aus dem sicherheitsrelevanten Bereich synthetisiert werden. Allerdings liegen keine Patentrezepte vor, mit denen die Synthese von problematischen DNA-Fragmenten unterbunden werden kann. 7 Synthetische Biologie als neue Technikwissenschaft Die Definition des Begriffs Synthetische Biologie beinhaltet den Hinweis, dass dieses Fachgebiet ingenieurwissenschaftliche Prinzipien nutzt. Diese Aussage bedarf weiterer Erläuterungen, wobei geklärt werden muss, welche besonderen ingenieurwissenschaftlichen Prinzipien zur Anwendung kommen. Hierzu soll als Beispiel die technische Entwicklung eines neuen Flugzeugtyps herangezogen werden. Zunächst werden am Reißbrett oder am Computer erste Pläne skizziert. Diese Pläne dienen dazu, die Flugeigenschaften des neuen Flugzeugs zu ermitteln, wobei Methoden eingesetzt werden, die mittels Simulation Voraussagen über das Flugverhalten des neuen Flugzeugs ermöglichen. Erst wenn diese Simulationen erfolgreich abgeschlossen sind, wird man daran gehen, einen ersten Prototyp zu bauen. Ein solcher Prototyp lässt sich dann unter realen Bedingungen experimentell auf seine Flugeigenschaften testen. Die geschilderte Vorgehensweise gilt nun auch für die Synthetische Biologie. Zunächst muss festgelegt werden, welche Eigenschaften die zu konstruierende synthetische Zelle besitzen soll. Dazu ist der Bauplan für die synthetische Zelle zu entwerfen, d. h. auf genomischer Ebene müssen die für die gewünschten Eigenschaften zuständigen Genfragmente zusammengestellt werden. Als nächstes wird dann in silico geprüft, ob der entworfene Bauplan zu einer lebensfähigen synthetischen Zelle mit den gewünschten Eigenschaften führt. Dazu benötigt man ebenfalls Modelle, die das System ‚synthetische Zellen‘ beschreiben und Simulationsschritte erlauben. Fallen diese Prüfungen zur Zufriedenheit aus, dann wird man auch hier den Prototyp einer synthetischen Zelle verwirklichen, also den genetischen Bauplan der synthetischen Zelle mittels genomischer Programmierung umsetzen. Diese analoge Vorgehensweise sowohl beim Bau eines neuen Flugzeugs als auch bei der Konstruktion einer synthetischen Zelle macht deutlich, warum man der Synthetischen Biologie ingenieurmäßige Prinzipien zuschreibt. Die ingenieurmäßige Vorgehensweise bei der Konstruktion von synthetischen Abb. 6: Präsentation der vier omics-Technologien. Die vier omics-Technologien Genomik, Transkriptomik, Proteomik und Metabolomik werden durch typische Geräte und die damit erzeugten Ergebnisse dargestellt. In (A) ist ein DNA-Sequenzierautomat und als typisches Ergebnis das zirkuläre Genom eines Bakteriums dargestellt. Teil (B) zeigt eine Maschine zur Herstellung von Mikroarrays und deren Einsatz bei der Messung von Transkriptionsvorgängen. Teil (C) zeigt ein spezielles Massenspektrometer, das zur Identifizierung von Proteinen eingesetzt wird. In Teil (D) ist erneut ein Massenspektrometer abgebildet, mit dem Metabolite entsprechend ihrer Masse und Quantität detektiert werden können. Zellen wirft die Frage auf, wie das Gesamtgeschehen in einer Zelle modellhaft erfasst werden kann. Dazu wurde im Vorfeld der Synthetischen Biologie bereits die Systembiologie auf Basis von omicsDaten entwickelt. Omics-Daten, die das Geschehen in einer Zelle ganzheitlich beschreiben, werden mit den omics-Technologien Genomik, Transkriptomik, Proteomik und Metabolomik gewonnen. In Abbildung 6 werden die vier grundlegenden omics-Technologien anhand der eingesetzten Apparaturen dargestellt. Mittels omics-Technologien werden dabei hochparallel alle Gene, alle Transkripte, die meisten Proteine und viele Metabolite einer Zelle erfasst, wobei die identifizierten Transkripte, Proteine und Metabolite jeweils vom Zustand der Zelle und seiner Umgebung abhängen und dadurch zeitlich stark variieren können. Omics-Daten entwerfen ein ganzheitliches Bild des zellulären Geschehens. Die Systembiologie versucht nun auf diesen omics-Daten aufbauend das Gesamtgeschehen in einer Zelle modellhaft zu erfassen. So wird versucht die Genregulation in einer Zelle anhand der Transkripte und den Stofffluss über das Auftreten von Metaboliten zu modellieren. Solche Modelle erleichtern anschließend die Entwicklung von genomischen Bauplänen für synthetische Zellen und spielen auch eine wesentliche Rolle, wenn erste Prototypen von synthetischen Zellen detailliert analysiert werden. Die obige Darstellung zeichnet den Weg vor, der der Synthetischen Biologie den Übergang von einer biologischen Wissenschaft zu einer Technikwissenschaft ermöglicht. Allerdings ist der Weg noch weit. Die Systembiologie ist über Anfangserfolge noch nicht hinaus gekommen und die modellhafte Erfassung des gesamten zellulären Geschehens aus omics-Daten scheint eine Jahrhundertaufgabe zu sein. Die Gründe hierfür sind offensichtlich, denn das zelluläre Geschehen ist hochkomplex und kann augenblicklich nur in Teilen erfasst werden. Trotz all dem wird der Synthetischen Biologie eine große Zukunft vorausgesagt, insbesondere wenn man ihr Zusammenspiel mit der Biotechnologie in Betracht zieht. Die Biotechnologie nutzt ja das Synthesepotenzial von lebenden Zellen zur Produktion von bestimmten Substanzen. In Zukunft lässt sich nun dieses Synthesepotenzial mittels Synthetischer Biologie ausweiten, womit die Produktion neuartiger Substanzen ermöglicht wird, die sich heute noch einer biotechnologischen Produktion entziehen. Die zukünftige Vorstellung einer biotechnologischen Produktion mittels Synthetischer Biologie geht von der Idee aus, dass vorhandene Chassis-Zellen mit ausgewählten Genkassetten zur Produktion von neuartigen Substanzen beladen werden. Mit dieser Vision wird der Synthetischen Biologie eine bedeutende Rolle bei der zukünftigen Entwicklung der Biotechnologie zugeschrieben. Synthetische Biologie wird in Zukunft als Technikwissenschaft also besonders für die Fortentwicklung der Biotechnologie eine herausgehobene Rolle spielen. ■ Anmerkung * Der vorliegende Artikel baut auf einem Vortrag auf, den der Autor im Rahmen einer 9 PdN BIOLOGIE in der Schule / BIOTECHNOLOGIE – SYNTHETISCHE BIOLOGIE Schülerakademie am 7. Juli 2014 am Centrum für Biotechnologie der Universität Bielefeld gehalten hat. Literatur [1] Synthetische Biologie; Synthetic Biology: Stellungnahme. Standpunkte. (2009), Hrsg. Deutsche Forschungsgemeinschaft (DFG), acatech – Deutsche Akademie der Technikwissenschaften, Deutsche Akademie der Naturforscher Leopoldina, WILEY-VCH. [2] Watson, J., Crick, F. (1953): Molecular structure of Nucleic Acids: A Structure for Deoxyribose Nucleic Acid; Nature 171, Nr. 4356, 737–738. [3] Nirenberg, M. W.: The Genetic Code, Nobel Lecture, December 12, 1968. [4] Cohen, S. N., Chang, A. C. Y., Boyer, H. W., Helling, R. B. (1973): Construction of Biologically Functional Bacterial Plasmids In Vitro; Proc. Nat. Acad. Sci. USA 70 (11), 3240–3244. [5] Gesetz zur Regelung der Gentechnik (Gentechnikgesetz – GenTG); http://www.gesetzeim-internet.de/gentg/index.html [6] Maxam, A. M., Gilbert, W. (1977): A new method for sequencing DNA, Proc. Natl. Acad. Sci. USA. 74 (2), 560–564. [7] Sanger, F., Nicklen, S., Coulson, A. R. (1977): DNA sequencing with chain-terminating inhibitors, Proc. Natl. Acad. Sci. USA. 74 (12), 5463–5467. [8] Reese, C. B. (2005): Oligo- and poly-nucleotides: 50 years of chemical synthesis; Org. Biomol. Chem. 3, 3851–3868. [9] Gibson, D. G. et al. (2010): Creation of a bacterial cell by a chemically synthesized genome; Science 329 No. 5987, 52–56. [10] Venter, J. C., et al. (2001): The sequence of the human genome. Science 291 No. 5507, 1304–1451. [11] Venter, J. C., et al. (2004): Environmental Genome Shotgun Sequencing of the Sargasso Sea. Science 304 No. 5667, 66–74. HEFT 3 / 64. JAHRGANG / 2015 [12] Pühler, A. (2010): Leben aus der Retorte; Biospektrum 05.10, 16. Jahrgang. [13] Martin, V. J. J., Pitera, D. J., Withers, S. T., Newmann, J. D., Keasling, J. D.: (2003): engineering a mevalonate pathway in Escherichia coli for production of terpenoids; Nat. Biotechnol. 21, 796–802. [14] Ro, D. K., et al. (2006): Production of the antimalarial drug precursor artemisinic acid in engineered yeast; Nature 440, 940–943. [15] http://igem.org/ [16] Monitoring der Synthetischen Biologie in Deutschland (2012): 1. Zwischenbericht der Zentralen Kommission für die Biologische Sicherheit; Bundesamt für Verbraucherschutz und Lebensmittelsicherheit. [17] Itaya, M. (2010): A synthetic DNA transplant. Nature Biotechnology 28, 687–689. Anschrift des Verfassers Prof. Dr. A. Pühler, Centrum für Biotechnologie, Universität Bielefeld, 33594 Bielefeld Lösungen zu den Aufgaben des Arbeitsblattes Zu 1.: Der Zusammenbau des Mycoplasma-mycoidesGenoms begann mit der Synthese von relativ kleinen DNAAbschnitten (Oligonukleotide). Die Oligonukleotide wurden dann in Bakterien der Art Escherichia coli vermehrt. Das Verknüpfen der Fragmente erfolgte nun schrittweise in Hefezellen. Dabei durften die Bruchstücke nicht wahllos aneinander gekettet werden, sondern die richtige Reihenfolge musste beachtet werden. Durch die überlappenden Abschnitte konnten sich aufgrund der Basenpaarung die Fragmente in der vorgesehenen Reihenfolge finden. Zu 2.: Im Wesentlichen handelt es sich um biochemische Methoden, zum Beispiel • Proteomik (Bestimmung der Proteinzusammensetzung in Zellen v. a. durch 2D-Gelelektrophorese) • DNA-Sequenzierung (Ablesen der Nukleotid-Abfolge; man unterscheidet die klassischen Methoden wie z. B. die Didesoxymethode nach Sanger, und die modernen Sequenzierungsmethoden der 2. Generation wie z. B. die Hybridisierungsmethode) • Genexpressionsanalyse (Untersuchung der Transkriptionsaktivität einzelner Gene) • Mikroarrays (z. B. Nachweis der mRNA-Menge bestimmter Zielgene). Zu 3.: Durch Antibiotika werden Bakterienzellen abgetötet. Mycoplasmen werden zum Beispiel durch Tetrazyklin zerstört. Wenn dagegen in das Bakteriengenom das Tetrazyklin-Gen eingebaut ist, wird ein Enzym gebildet, welches das Antibiotikum unwirksam macht. Eine Bakterienzelle, die dieses Gen enthält, überlebt also auf tetrazyklinhaltigen Nährböden. So können Bakterienzellen, die ein Genom mit oder ohne dieses Gen aufweisen, unterschieden werden. Zu 4.: Der intrazelluläre Raum ist durch sehr konstante chemische und physikalische Faktoren gekennzeichnet. Parasiti10 sche Bakterien wie die Mycoplasmen, die intrazellülär in ihren Wirtsorganismen und damit unter gleichbleibenden Umweltbedingungen leben, haben im Lauf der Evolution einen großen Teil der Gene, die sonst zur Auseinandersetzung mit den Umweltbedingungen nötig wären, verloren. Nicht notwendige Gene unterliegen nicht dem Selektionsdruck, mutieren relativ rasch und werden im Lauf von relativ wenigen Generationen abgebaut. Zu 5.: Unter Transformation versteht man die Übertragung von blanker DNA auf lebende Bakterien. Die synthetische DNA wird in der Bakterienzelle neben dem vorhandenen Lineom ebenfalls als Lineom eingebaut. Bei der nächsten Zellteilung erhält jede Tochterzelle eines der beiden Lineome. Wenn in das in-vitro synthetisierte Lineom zum Beispiel ein Tetrazyklin-Gen eingebaut wurde, ist eine gezielte Auswahl (Selektion) der Bakterien mit dem neu synthetisierten Lineom möglich. Durch Verwendung eines Nährbodens mit Tetrazyklin sterben die Bakterien mit dem ursprünglichen Genom ab, während die Bakterien mit der neu synthetisierten DNA überleben und sich vermehren. Zu 6.: Die Medien haben mit dieser Einschätzung gewaltig übertrieben. Natürlich ist die wissenschaftliche Arbeit, die sich über Jahre hinzog und von Hunderten von Wissenschaftlern geleistet wurde, enorm. Es wurde aber kein neues Leben erschaffen. Die vorhandenen Stoffwechselkapazitäten der Mycoplasma-Zellen sowie die notwendigen Baumaterialien wurden genutzt. Die seit der Entstehung des Lebens auf der Erde in Milliarden Jahren entwickelten biochemischen Produktionsmöglichkeiten wurden im Labor nur nachvollzogen, wobei auf vorhandene biochemische Materialien zurückgegriffen wurde. Das Arbeitsblatt wurde erstellt durch Dr. Wolfgang Jungbauer, Wilhelm-Röntgen-Str. 3, 91710 Gunzenhausen, E-Mail: [email protected] PdN BIOLOGIE in der Schule / BIOTECHNOLOGIE – SYNTHETISCHE BIOLOGIE 3 / 64. JAHRGANG / 2015 Arbeitsblatt „Und am achten Tag erschuf Craig Venter künstliches Leben“ (Zitat Cinthia Brisenõ in www.spiegel.de/wissenschaft/natur/erster-kuenstlicher-organismus-/ sie-sollen-tun-was-wir-wollen-a-696057.html) Kein Biogenetiker unserer Zeit ist so umstritten wie der Abschnitt 1 Design der zu synthetisierenden DNA: US-Amerikaner John Craig Venter. Mit seinem im Jahr Festlegung der Basensequenz mit wesent2001 erzielten Erfolg der Entschlüsselung des menschlichen Teilen des Genoms von Mycoplasma mycoides ssp. capri, dazu einige lichen Erbgutes in seinen privaten Forschungslaweitere, für die Synthese wichtige Gene bors – und zwar schneller als die staatlich geförderten sowie von Markersequenzen zur Identifizierung der neuen DNA Forschungsinstitute (HUGO, Human-Genom-Projekt) – zog er das Interesse der internationalen Medien auf Abschnitt 2 sich. Als ihm dann noch weitere spektakuläre Erfolge In-vitro-Synthese von Oligonukleotiden als kürzeren gelangen, wurde er schon als zukünftiger Nobelpreis„Bausteinen“ der gewünschten DNA träger gefeiert. Aber seine auf Grund der Profitorientierung zwiespältige Arbeit, gefördert durch Sponsorengelder, stieß auf viel Misstrauen. 2010 folgte der Aufbau von kürzeren Fragmenten und Vervielfältigung in Coli-Bakterien (1-kbp-Fragmente) nächste Paukenschlag: Craig Venter verkündete die Erschaffung künstlichen Lebens. Zusammenbau der Fragmente und Klonierung Die wissenschaftliche Leistung von Craig Venter und in Hefezellen (10-kbp-Fragmnente, 100 kbpFragmente, schließlich komplettes Genom) seinem Team ist dabei unumstritten. Zum ersten Mal gelang es, eine künstliche DNA in die Zelle einer BakAbschnitt 3 terienart zu übertragen, worauf diese Zelle nach AnFreisetzen der vervielfältigten synthetischen weisung der synthetischen DNA aktiv wurde. Das Ziel DNA aus den Hefezellen Transformation der synthetisierten DNA in war also die Konstruktion eines neuen BakterienstamZellen der Bakterienart Mycoplasma capricolum mes mit Hilfe einer am Computer entworfenen und Selektion der transformierten Bakterien und dann im Labor synthetisierten DNA. Anzucht auf Nährböden Als Vorbild diente das als Erreger einer LungenNach 30 Teilungsschritten Überprüfung der Funktion der synthetisierten DNA krankheit bei Huftieren bekannte Bakterium Mycoplasma mycoides. Die Verwendung dieser Bakterienart war sehr vorteilhaft, nämlich ein sehr kleines Genom von nur knapp mehr als einer Million Basenpaaren sowie das Fehlen einer Zellwand. Besorgnis löste dagegen die Tatsache aus, dass es sich um parasitär lebende Bakterien handelte, wodurch eventuell nicht kontrollierbare Probleme entstehen könnten. Die Forscher orientierten sich an dem damals bereits vollständig bekannten Genom von Mycoplasma mycoides ssp. capri. Die einzelnen Arbeitsabschnitte gliederten sich wie in der Abbildung dargestellt. Ergebnis: Es ist ein neuer Bakterienstamm mit den vorherbestimmten Eigenschaften entstanden (z. B. Blaufärbung der veränderten Zellen auf bestimmten Nährböden). Der neue Stamm heißt Mycoplasma mycoides JCVI syn1.0 (die Abkürzung JCVI steht für „John Craig Venter Institute“ in San Diego (USA) Aufgaben 1. Die von der Craig-Venter-Gruppe verwendeten DNA-Streifen („Fragmente“) enthielten jeweils einen zusätzlichen, überlappenden Abschnitt von 80 bp (Basenpaaren). Erklären Sie dies. 2. Zwischen den Genomen von Mycoplasma mycoides ssp. capri und Mycoplasma mycoides JCVI syn.1.0 bestehen große Übereinstimmungen, aber auch etliche gravierende Unterschiede. Nennen sie einige Methoden, solche Unterschiede festzustellen und kennzeichnen Sie jeweils die Methode mit einer kurzen Defi nition. 3. In das chemisch synthetisierte Genom von Mycoplasma mycoides JCVI syn1.0 wurde von der Forschungsgruppe ein Gen für bakterielle Resistenz gegen das Antibiotikum Tetrazyklin eingebaut. Formulieren Sie eine begründete Hypothese für diesen Arbeitsschritt. 4. Das Fehlen einer Zellwand bei Mycoplasma mycoides kann als Angepasstheit an den Parasitismus interpretiert werden. Parasitisch lebende Bakterien weisen nämlich im Vergleich zu nicht parasitären Arten ein deutlich kleineres Genom auf. Begründen Sie diesen Sachverhalt. 5. Nach der Transformation einer Mycoplasma-mycoides-Zelle befinden sich kurzfristig in dem Bakterium zwei Genome nebeneinander. Bei der nächsten Zellteilung erfolgt dann eine Entmischung. Im Laborexperiment besteht damit die Möglichkeit zur Selektion und gezielten Weitervermehrung von Zellen, die das synthetische Genom enthalten. Begründen Sie diese Aussagen. 6. In den Medien wurde der Erfolg der Craig-Venter-Gruppe als bahnbrechendes wissenschaftliches Ereignis gefeiert. Bewerten Sie die Richtigkeit des in der Überschrift dieses Arbeitsblattes verwendeten Zitats. Stellen Sie Pro- und Contra-Argumente begründend gegenüber. 11