Biologie 11. Klasse – 2. KA 1. Transportmechanismen
Werbung

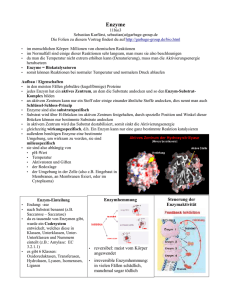
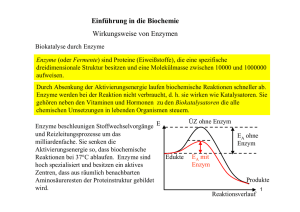
Biologie 11. Klasse – 2. KA 1. Transportmechanismen durch die Biomembran 1.1. Passiver Transport Erfolgt immer in Richtung des Konzentrationsgefälles Keine chemische Energie wird verbraucht Antrieb für diese Art von Transport ist der Konzentrationsunterschied → je größer das Konzentrationsgefälle, desto schneller die Diffusion 1.1.1. Einfache Diffusion Lipophile und sehr kleine ungeladene Moleküle wie Gase und Wasser (mit Einschränkungen) → können Membran direkt passieren Diffundieren durch Lipiddoppelschicht → ermöglicht durch die ständige Bewegung der Lipidmoleküle der Biomembran 1.1.2. Erleichterte Diffusion Phospholipide → behindern Diffusion hydrophiler Teilchen Spezifische Membranproteine erleichtern das Passieren der Zellmembran (Tunnelproteine oder Trägerproteine/Carrier) Tunnelproteine →durchspannen Membran und bilden einen Kanal, durch den die Teilchen diffundieren können Polare Aminosäuren im Inneren des Kanals → kleine polare oder geladene Teilchen wie Ionen können über Kanäle in die Zelle gelangen Wassermoleküle → Aquaporine (hydrophile Tunnelproteine) Manche Kanäle → öffnen Pforte auf ein bestimmtes Signal hin (Spannungsänderung oder Anlagerung eines Moleküls, z. B. Hormon) Carrier → nehmen das zu transportierende Molekül auf und geben es auf der anderen Membranseite wieder ab Jeder Carrier transportiert nur einen bestimmten Molekül oder nahe verwandte Moleküle (auf bestimmte Moleküle spezialisiert → haben für sie eine Bindungsstelle) Nach Verbindung des Carries mit dem Substrat → Änderung der Konformation (3D Form) Durch diese Umlagerung wird das betreffende Molekül durch die Membran geschleust und auf der anderen Seite freigesetzt Manche Carrier → Bindungstelle für zwei verschiedene Teilchen; sie verändern ihre Konformation für einen Durchlass nur wenn beide Stellen besetzt sind → Cotransport verläuft entweder in gleicher (Symport) oder entgegen gesetzter Richtung (Antiport) 1.2. Aktiver Transport Ionen oder Moleküle können durch eine Zellmembran in entgegengesetzter Richtung zum Konzentrationsgefälle transportiert werden (niedrige Konz. zu höh. Konz.) Energie wird verbraucht (ATP bindet an eine Bindungsstelle des Proteins und aktiviert es) POR DIANA X. F. 1.2.1. Primär aktiver Transport Transportprotein ähnelt einer Pumpe → es bindet das Molekül und transportiert es unter Energieaufwand durch die Membran Dabei ändert sich die Konformation des Transportproteins Bekanntes Beispiel: Natrium-Kalium-Pumple (entgegengesetzt gerichteter Transport von 3 Na+-Ionen und 2 K+-Ionen unter Spaltung von einem ATP-Molekül) 1.2.2. Sekundär aktiver Transport Cotransport von zwei Molekülen/Ionen → Na+-Ionen werden entlang ihres Konzentrationsgefälles transportiert, wodurch ein anderes Molekül/Ion entgegen seines Konzentrationsgefälles transportiert wird Konzentrationsgefälle an Na+-Ionen genutzt, welches durch die Natrium-Kalium-Pumpe erzeugt wird → das zweite Molekül/Ion kann in die gleiche oder entgegengesetzte Richtung wie Na+-Ionen transportiert werden ohne Verbrauch von ATP Energie wird indirekt verbraucht → damit der Konzentrationsgradient der Na+-Ionen aufrecht erhhalten bleibt, mussen diese entgegen das Konzentrationsgefälles transportiert werden (primär aktiver Transport) 2. Proteine Proteine übernehmen viele verschiedene Funktionen in unserem Körper, z. B. Enzyme, Hormone, Antikörper (Immunabwehr) und Strukturproteine (z. B. Haare, ...) 2.1. Aufbau der Aminosäuren Aminogruppe (H2N) Carboxylgruppe (COOH) Organischer Rest: unpolar (z. B. Alanin); polar (z. B. Serin); sauer (z. B. Asparaginsäure); basisch (z. B. Lysin) 2.2. Die Peptidbindung Proteine entstehen durch Verknüpfung der Aminosäuren-Bausteine. Dabei reagiert die Aminogruppe der einen Aminosäure mit der Carboxylgruppe der anderen Aminosäure unter Abspaltung von H2O. Die entstehende Bindung nennt man Peptidbindung (eine kovalente Bindung). Die Proteinkette (Polypeptidkette) besitzt immer ein Ende mit freier Aminogruppe und ein Ende mit freier Carboxylgruppe. POR DIANA X. F. 2.3. Die Primärstruktur AS können in beliebiger Reihenfolge in einem Protein auftreten Die Abfolge/Sequenz der einzelnen AS nennt man Primärstruktur 2.4. Die Sekundärstruktur Räumliche Struktur von Proteinen Ergibt sich durch die Ausbilbung von Wasserstoffbrücken zwischen der CO-Gruppe und der NH-Gruppe der Peptidbindungen (im Proteinrückgrad) Entweder α-Helix (schraubige Anordnung) oder β-Faltblatt 2.5. Die Tertiärstruktur Ausbildung der 3D Struktur des Proteins Ergibt sich durch Ausbildung von Bindungen/Wechselwirkungen zwischen den Seitenketten (Rest) der Aminosäuren der Peptidbindung Wechselwirkungen: Disulfidbrücken (kovalente Bindung, am stärksten); Ionenbindung; Wasserstoffbrücken; VdW-Wechselwirkungen 2.6. Die Quartärstruktur Zusammenlagerung mehrer Proteinketten zu einem Protein 3. Die Enzymatik Enzyme sind Biokatalysatoren. Bei den Stoffwechselvorgängen in Zellen handelt es sich um chemische Reaktionen. Diese Reaktionen laufen nur ab, wenn die benötigte Aktivierungsenergie überschritten wird. Enzyme wirken als Biokatalysatoren, d. h. sie beschleunigen bzw. ermöglichen chemische Reaktionen (auch schon z. B. bei Körpertemperatur), indem sie die Aktivierungsenergie herabsetzen. (Energiediagramm S. 65) 3.1. Die enzymatische Reaktion Wichtig: das Enzym geht unverändert aus der Reaktion hervor → kann neues Substrat binden Der aktive Zentrum: taschenförmige Einbuchtung, in die das Substrat eingelagert wird. Hier findet die chemische Reaktion statt Schlüssel-Schloss-Modell: das Substrat und das Enzym passen wie Schlüssel und Schloss zueinander (räumliche Struktur) Schlüssel-Schloss-Prinzip → Enzyme sind substratspezifisch POR DIANA X. F. Enzyme sind auch wirkungsspezifisch → sie katalysieren nur eine von vielen möglichen Reaktionen. 3.2. Cofaktoren Binden an Enzyme Sind für die Aktivität bestimmter Enzyme unerlöslich Enzyme: entweder reine Proteinenenzyme oder Proteidenenzyme. Proteidenenzyme: Apoenzym + Cofaktor = Holoenzyme Cofaktoren: entweder Metallionen (Spurenelemente) oder Coenzyme (z. B. Vitamin B) Die Coenzyme transportieren Molekülgruppen an das Substrat (z. B. Wasserstoff-, Phosphatgruppen). Sie reagieren mit dem Substrat wenn sie locker gebunden sind. Wenn sie fest am Enzym gebunden sind reagieren sie nicht. Metallionen: stabilisieren als Ladungsträger das Enzym, dienen als Bindungsstelle von Substrat und Enzym 3.3. Denaturierung Bei hohen Temperaturen → Tertiärstruktur wird verändert und zerstört → Bindungskräfte (vor allem VdW-WW und H-Brücken) überwunden werden (die sind für die räumliche Struktur der Enzyme verantwortlich Passform des Enzymmoleküls für das Substrat verloren (aktives Zentrum → zerstört) Reste der Aminosäuren: unpolare: Ala, Val, Leu, Ile, Met, Pro, Phe basische: Lys, Arg, His POR DIANA X. F. polare: Ser, Thr, Cys, Tyr, Trp, Asn, Gln saure: Asp, Glu