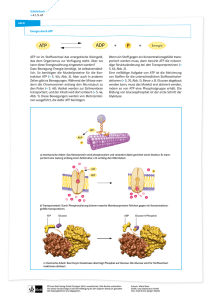
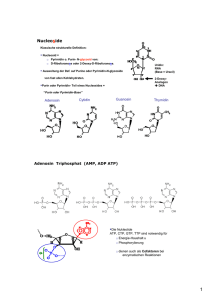
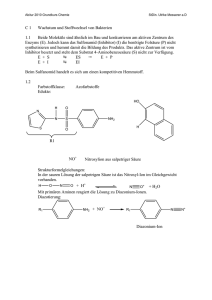
Zusammenfassung Biochemie WS 2003/04
Werbung

BIOCHEMIE Zusammenfassung von Ana Sesartic WS 2003/04 Die Zusammenfassung ist gegliedert nach Kapiteln der 5. deutschen Ausgabe des Buches Biochemie von J.M.Berg, J.L.Tymoczko und L.Stryer, Spektrum Verlag, 2003. Auf diese Ausgabe beziehen sich auch alle genannten Seitenzahlen und Kapitelnummern. Es werden nur die Kapitel und Themen berücksichtigt, die explizit als Prüfungsstoff angegeben wurden. Die Biochemie und die Revolution der Genomforschung (Kapitel 1, Seiten 3-18)...... 2 Struktur und Funktion der Proteine (Kapitel 3, Seiten 45-81) ..................................... 5 Erforschung der Proteine (Kapitel 4, Seiten 77-104) ................................................ 11 Enzyme: Grundlegende Konzepte und Kinetik (Kapitel 8, Seiten 210-241).............. 15 Katalytische Strategien (Kapitel 9, Seiten 249-268).................................................. 20 Lipide und Zellmembranen (Kapitel 12, Seiten 349-374).......................................... 23 Der Stoffwechsel: Konzepte und Grundmuster (Kapitel 14, Seiten 407-427) ........... 27 Kohlenhydrate (Kapitel 11, Seiten 323-334) ............................................................. 30 Glykolyse und Gluconeogenese (Kapitel 16, Seiten 465-491).................................. 33 Der Citratzyklus (Kapitel 17, Seiten 509-531) ........................................................... 38 Die oxidative Phosphorylierung (Kapitel 18, Seiten 535-570)................................... 43 © 2004 Ana Sesartic 1 Prolog: Die Biochemie und die Revolution der Genomforschung (Kapitel 1, Seiten 3-18) 1.1 DNA = DesoxyriboNucleicAcid Enger Zusammenhang zwischen Struktur und Funktion. DNA ist ein höchst wirksamer und robuster Informationsspeicher. 1.1.1 DNA ist ein lineares Polymer aus vier verschiedenen Monomeren (= ZuckerPhosphat-Einheit mit einer der 4 Basen am Zuckerbestandteil angehängt). Die Basen Adenin (A), Cytosin (C), Guanin (G) und Thymin (T) können entlang eines DNA-Stranges beliebig angeordnet sein. Die Basensequenz eines DNA-Stranges stellt die genetische Information dar. 1.1.2 Das DNA-Molekül ist eine Doppelhelix mit Zucker-Phosphat-Rückgrat aussen und Basenpaaren (AT bzw. GC) innen. Die Basen werden durch Wasserstoffbrücken zusammengehalten. Aufgrund der Basenpaarung legt die Sequenz in einem Strang die Sequenz des anderen genau fest. Bei der DNAReplikation kann jeder Strang als Matrize für die Neubildung des Partnerstrangs dienen. 1.1.3 RNA = RiboNucleicAcid RNA liegt meist als Einzelstrang vor. Als Zucker hat sie Ribose und anstelle von Thymin hat sie die Base Uracil (U). Aufgaben: Zwischenstufe im Informationsfluss von der DNA zu den Proteinen (Transkription von DNA in mRNA) Übersetzen der Information von mRNA Sequenz in Informationen, welche die Sequenz der Proteinbausteine bestimmt (Translation von mRNA in ein Protein) Funktioneller Bestandteil der Ribosomen (Translationsmaschinen) Transkription der DNA durch mRNA und anschliessende Translation der mRNA in Proteine durch Ribosomen. © 2004 Ana Sesartic 2 1.1.4 Wichtige Aufgabe der DNA-Sequenzen ist die Sequenzen der Proteine festzulegen. Genetischer Code: 3 Basen in der DNA Kette legen genau eine Aminosäure fest. Proteine sind lineare polymere die aus 20 verschiedenen Aminosäuren bestehen. Sie falten sich von selbst in eine genau definierte, vielgestaltige Raumstruktur, die ausschliesslich durch die Sequenz ihrer Aminosäuren festgelegt wird. 1.2 Alle Organismen bedienen sich der DNA und des gleichen genetischen Codes. Unter molekularen Gesichtspunkten sind die Lebewesen bemerkenswert einheitlich Alle stammen von einem gemeinsamen Vorfahren ab. Die Vielfalt entstand v.a. durch Anpassung vorhandener Biochemischer Komponenten an neue Aufgaben Eukarya: Eukaryonten (mit Zellkern) Bacteria: Prokaryonten (ohne Zellkern) Archaea: (Bakterienähnlich, aber deutlich verschieden) 1.3 Kovalente Bindungen: benachbarte Atome teilen sich ein Elektronenpaar. Um die Bindung aufzulösen braucht es viel Energie, wobei Mehrfachbindungen (z.B. C=O hat 732 kJ/mol) noch stärker sind als Einfachbindungen. Name of bond Covalent bond (C-C) Hydrogen bond Electrostatic interaction Van der Waals interaction Distance (Å) 1.54 1.5-2.6 3 1.5-3 Bond energy (kJ/mol) 356 4-13 6 2-4 Resonanzstrukturen: gleichbedeutende Strukturen (Mesomere) das man Resonanzstrukturen schreiben kann ist stabiler! Molekül für Reversible Wechselwirkungen zwischen Biomolekülen werden durch drei Arten nicht-kovalenter Bindungen vermittelt: Elektrostatische Bindungen Wasserstoffbrücken Van der Waals-Bindungen o Unspezifische, dipolinduzierte Wechselwirkungen o Grössenordnung: ca. 4 kJ/mol o Zum Vergleich: thermische Energie von Molekülen: ca. 2.5 kJ/mol 1.3.1 Elektrostatische Wechselwirkungen: Anziehungskräfte entgegengesetzt geladener Atome. Im Wasser haben sie Energiegehalt von 5.9 kJ/mol Wasserstoffbrücken: 2 Elektronegative Atome wie N oder O teilen sich ein Wasserstoffatom. Donorgruppe: H und Atom das H stark an sich bindet, Akzeptorgruppe: H weniger stark daran gebunden. © 2004 Ana Sesartic 3 Van-der-Waals-Wechselwirkungen: Verteilung der Elektronenladung um einen Atomkern ändert sich im Laufe der Zeit. Diese vorübergehende Asymmetrie der Elektronenladung führt zu VdW-Kräften. Bei Kontaktdistanz sind Energieverhältnisse am günstigsten. 1.3.2 Wasser ist ein polares Molekül, ist geknickt und hat deshalb asymmetrische Ladungsverteilung. Wasser ist wegen Wasserstoffbrücken sehr kohäsiv, und neigt zu Interaktionen. Wasser ist für polare Moleküle ein hervorragendes Lösungsmittel. Es schwächt die elektrostatischen Wechselwirkungen zwischen Ionen beträchtlich ab (Dielektrizitätskonstante = 80) durch Bildung einer gerichteten Hülle (Aquakomplex). Wechselwirkungen zwischen polaren Molekülen werden ebenfalls geschwächt, was aber in biologischen Systemen mit wasserfreien Mikroumgebungen umgangen wird. 1.3.3 1. Hauptsatz der Thermodynamik: Gesamtenergie eines Systems und seiner Umgebung ist konstant. Das Universum hat stets den gleichen Energieinhalt. Energie kann weder erschaffen noch zerstört werden, sie kann aber verschiedene Formen annehmen. Kinetische Energie: zufällige Molekülbewegung, Wärme Potentielle Energie: Wahrscheinlichkeit, dass Atome miteinander reagieren 2. Hauptsatz der Thermodynamik: Die Gesamtentropie eines Systems und seiner Umgebung nimmt bei spontan ablaufenden Prozessen stets zu. Enthalpie: Wärmegehalt eines Systems. Entropie: Mass für die Zufälligkeit oder Unordnung in einem System. Entropie nimmt nur dann zu, wenn: ∆G = ∆HSystem - T ∆SSystem < 0 Damit eine Reaktion spontan ablaufen kann, muss die Veränderung der freien Enthalpie negativ sein. © 2004 Ana Sesartic 4 1.3.4 Hydrophobe Wechselwirkung (a.k.a. Hydrophober Effekt): Wenn unpolare Moleküle sich in Wasser zusammenlagern, nimmt die Entropie zu, weil Wassermoleküle in die Hauptmenge des Wassers abgedrängt werden. Proteinfaltung: unpolare Aminosäuren neigen stark dazu, sich im Inneren eines gefalteten Proteinmoleküls aneinander zu lagern. Die daraus entstandene Entropiezunahme des Wassers, gleicht den Entropieverlust aus, der mit dem Faltungsprozess verbunden ist. Bei der Proteinfaltung bilden sich auch viele schwache Bindungen aus, wodurch Wärme an die Umgebung abgegeben wird. Struktur und Funktion der Proteine (Kapitel 3, Seiten 45-81) 3 Proteine sind lineare Polymere, die aus mesomeren Untereinheiten, den Aminosäuren, zusammengesetzt sind. Proteine enthalten eine grosse Vielfalt an funktionellen Gruppen, deren Reaktivität bedeutend ist für die Funktion von Enzymen (= Katalysatoren). Proteine können miteinander und mit anderen biologischen Makromolekülen interagieren und komplexe Zusammenschlüsse bilden. Manche Proteine sind relativ starr, während andere über eine begrenzte Flexibilität verfügen. 3.1 Eine α-Aminosäure besteht aus einem zentralen α-Kohlenstoff, an den eine Aminogruppe, eine Carboxylgruppe, ein Wasserstoffatom und ein charakteristischer "Rest" R (= Seitenkette) gebunden sind. Aminosäuren sind chiral (D und L Isomere). [Achirale Ausnahme: Glycin mit H als Rest] Nur L-Aminosäuren (fast alle mit SKonfiguration) sind Bestandteile von Proteinen. In neutraler Lösung liegen Aminosäuren als dipolare Ionen (Zwitterionen) vor, wobei die Aminogruppe © 2004 Ana Sesartic 5 protoniert und die Carboxylgruppe dissoziiert (pH-abhängig veränderbar). Die 20 Arten von Aminosäurenseitenketten unterscheiden sich in Grösse, Gestalt, Ladung, Fähigkeit zur Ausbildung von H-Brücken, hydrophobem Charakter und chemischer Reaktivität. Längere aliphatische Seitenketten sind hydrophob und stabilisieren dadurch die 3D Struktur wasserlöslicher Proteine. Aminosäuren mit aliphatischer Seitenkette Glycin (Gly, G) Abb.S.49 o R=H o Achiral (Ausnahme) Alanin (Ala, A) Abb.S.49 o R = CH3 (Methylgruppe) Valin (Val, V) Abb.S.49 Leucin (Leu, L) Abb.S.49 Isoleucin (Ile, I) Abb.S.49 o Zusätzliches Chiralitätszentrum Methionin (Met, M) Abb.S.49 o Thioethergruppe (-S-) o R = CH2CH2SCH3 Prolin (Pro, P) Abb.S.50 o Zyklische Struktur (beeinflusst stark den Aufbau eines Proteins) o Seitenkette bindet an α-Kohlenstoff und Aminogruppe Aminosäuren mit aromatischer Seitenkette Phenylalanin (Phe, F) Abb.S.50 o R = Phenylring o Leicht hydrophob Tyrosin (Tyr, Y) Abb.S.50 o Aromatischer Ring enthält reaktionsfreudige Hydroxylgruppe und kann wegen delokalisierten π–Elektronen sehr stark UV-Licht absorbieren o Weniger hydrophob Tryptophan (Trp, W) Abb.S.50 o Aromatischer Ring enthält reaktionsfreudige Hydroxylgruppe und kann wegen delokalisierten π–Elektronen sehr stark UV-Licht absorbieren o Weniger hydrophob Aminosäuren mit Hydroxylgruppen enthaltenden aliphatischen Seitenketten Serin (Ser, S) Abb.S.52 o Wegen Hydroxylgruppe hydrophiler und reaktiver als Ala und Val Threonin (Thr, T) Abb.S.52 o Wegen Hydroxylgruppe hydrophiler und reaktiver als Ala und Val o Zusätzliches Chiralitätszentrum Cystein (Cys, C) Abb.S.52 Enthält Sulfhydryl- (sehr reaktionsfreudig) oder Thiol-Gruppe (-SH-). Je 2 dieser Gruppen können sich zu einer Disulfidbrücke (= Oxidation der freien Schwefelgruppe; kovalente Bindung) vereinigen, welche eine wichtige Rolle bei der Stabilisierung von Proteinen spielt. Aminosäuren mit basischen Seitenketten Lysin (Lys, K) Abb.S.53 o Seitenkette sehr polar und somit hydrophil © 2004 Ana Sesartic 6 Endgruppe (primäre Aminogruppe) bei neutralem pH positiv geladen Arginin (Arg, R) Abb.S.53 o Seitenkette sehr polar und somit hydrophil o Endgruppe (Guanidiniumgruppe) bei neutralem pH positiv geladen Histidin (His, H) Abb.S.53 o Häufig im aktiven Zentrum eines Proteins, wo der Imidazolring im Verlauf enzymatischer Reaktionen Protonen je nach Bedarf binden oder freisetzen kann o Aminosäuren mit sauren Seitenketten Asparaginsäure (= Aspartat) Abb.S.54 Glutaminsäure (= Glutamat) Abb.S.54 Aminosäuren mit carboxamidhaltige Seitenketten Asparagin (Asn, N) Abb.S.54 o Ungeladenes Derivat von Aspartat Glutamin (Gln, Q) Abb.S.54 o Ungeladenes Derivat von Glutamat Sieben Aminosäuren haben leicht ionisierbare Seitenketten und sind in der Lage, Protonen abzugeben oder aufzunehmen, um Reaktionen zu ermöglichen oder Ionenverbindungen einzugehen. © 2004 Ana Sesartic 7 3.2 Proteine sind lineare Polymere (50-200 Aminosäuren lang), bei denen die αCarboxylgruppe einer Aminosäure mit der α-Aminogruppe einer zweiten Aminosäure über eine Peptidbindung verknüpft ist. Die Biosynthese von Peptidbindungen benötigt Energie. Peptidbindungen sind kinetisch stabil. Polypeptidkette: Reihe von über Peptidbindungen verknüpfter Aminosäuren (= Peptide; haben geringe Anzahl Aminosäuren). Polar. Das Aminoende ist der Beginn der Polypeptidkette (immer links !). Hauptkette sind die sich regelmässig wiederholenden Einheiten des Polypeptids, welche über hohes Potential zur Ausbildung von H-Brücken verfügen. Seitenketten sind variabel. Molekulargewicht der meisten Polypeptidketten liegt zwischen 5.5-220 kd (Kilo Dalton). Disulfidbrücken: entsteht durch Oxidation von zwei Cysteinresten. Solche Quervernetzungen findet man v.a. in extrazellulären Proteinen. © 2004 Ana Sesartic 8 3.2.1 Primärstruktur: Genetisch festgelegte Sequenz der Aminosäuren eines Proteins. Nucleotidsequenz der DNA spezifiziert eine komplementäre Nucleotidsequenz der RNA, welche die Aminosäuresequenz im Protein bestimmt, welche wiederum die 3D Struktur bestimmt. 3.2.2 Peptidbindung: (= C-N Bindung) ist im Prinzip planar und starr, weil ihr Doppelbindungscharakter die Rotation verhindert. Die anderen Bindungen sind drehbar und dadurch kommt die 3D Struktur zustande. Die Peptidbindung ist ungeladen und ermöglicht den verknüpften Aminosäurepolymeren sich zu dicht gepackten globulären Strukturen zusammenzulagern. Nahezu alle Peptidbindungen sind in trans-Konfiguration (α-C auf entgegengesetzten Seiten der Peptidbindung) geknüpft. CisVerknüpfung wäre durch sterische Kollisionen zwischen den Gruppen am α-C Atom gestört. Die Rotationsfreiheit um zwei der Bindungen jeder Aminosäure erlauben es Proteinen, sich auf verschiedene Weise zu falten. Die Starrheit der Peptideinheit und sterischer Platzmangel schränkt die Zahl der ihr möglichen Strukturen hinreichend ein, um die Proteinfaltung zu ermöglichen. 3.3 Polypeptidketten können sich zu regelmässigen Strukturen wie α-Helix, βFaltblatt, Kehren (β-turn) und Schleifen (Ω-loop) falten. 3.3.1 Wasserstoffbrücken der α-Helix: Die CO-Gruppe einer Aminosäure i bildet innerhalb der α-Helix eine H-Brücke mit der NH-Gruppe der Aminosäure i+4. Wasserstoffbrücken stabilisieren die Helix. Drehsinn einer Helix kann nach rechts oder links weisen. Grundsätzlich sind aber alle in Proteinen anzutreffenden α-Helices rechtsgewunden, da energetisch günstiger (weniger sterische Kollisionen). Zwei oder mehr Helices können sich zu einer Superhelix (coiled coil) umeinander-winden; einer sehr stabilen Struktur die sich u.a. in Myosin, Fibrin und Keratin findet. © 2004 Ana Sesartic 9 3.3.2 Die β-Faltblatt Struktur wird von H-Brücken zwischen den Strängen stabilisiert Paralleles β-Faltblatt: benachbarte Stränge verlaufen in gleiche Richtung Antiparalleles β-Faltblatt: Laufrichtung benachbarter β-Stränge ist entgegengesetzt β-Faltblatt ist vor allem in fettsäurebindenden Proteinen bedeutend. 3.3.3 Polypeptidketten können ihre Richtung umkehren, indem sie Kehren und Schleifen ausbilden. Kehren (Abb.S.66) und Schleifen liegen stets an der Oberfläche eines Proteins. Sekundärstrukturen Parallele und antiparallele β-Faltblatt-Struktur, wird von Wasserstoffbrücken zwischen den Strängen stabilisiert. α-Helices Biegungen (Kehren und Schleifen) Verteilung von α-Helices, β-Faltblatt, Kehren und Schleifen entlang einer Proteinkette. ≈ räumliche Anordnung von Aminosäureresten. 3.4 Tertiärstruktur: 3D Struktur einer ganzen Polypeptidkette. Wasserlösliche Proteine falten sich zu kompakten Strukturen mit einem unpolaren Kern. ≈ räumliche Beziehung von Aminosäureresten. Muster der Disulfidbrücken. Polypeptidkette faltet sich spontan so, dass die hydrophoben Seitenketten im Inneren verborgen sind und die polaren, geladenen Reste an der Oberfläche liegen (= ist thermodynamisch stabiler). amphipatisch: hydrophob im Inneren des Moleküls, polar auf der Oberfläche. "Ausnahme" Porin: Aussenseite hydrophob, Zentrum des Moleküls polar, bildet wassergefüllten Kanal. Domäne: kompakte globuläre Einheit, 30-400 Aminosäuren lang. 3.5 Quartärstruktur: räumliche Anordnung von Untereinheiten (einzelnen Polypeptidketten) und die Art ihrer Wechselwirkungen miteinander. 3.6 Aminosäuresequenz eines Proteins legt dessen 3D Struktur fest. Sequenz Konformation © 2004 Ana Sesartic 10 3.6.1 Aminosäuren zeigen unterschiedliche Neigungen zur Ausbildung von αHelices, β-Faltblatt und Kehren. Oftmals entscheidet das Umfeld über die endgültige Konformation. α-Helices: Glutamat, Alanin, Leucin β-Faltblatt: Valin und Isoleucin Kehren: Glycin, Asparagin, Prolin 3.6.2 Die Faltung von Proteinen ist ein hochkooperativer Vorgang. Proteine lassen sich durch Hitze oder chemische Substanzen denaturieren. Die Faltung / Entfaltung von Proteinen folgt einem Alles-oder-nichts Prinzip. Bedingungen, die nur einen Teil des Proteins in seiner Integrität stören, wirken sich mit grosser Wahrscheinlichkeit auf das gesamte Protein aus. Eine kooperative Faltung stellt sicher, dass sich teilweise gefaltete Strukturen, die mit Vorgängen innerhalb der Zelle wechselwirken könnten, nicht ansammeln können. 3.6.3 Die Proteinfaltung verläuft über eine fortschreitende Stabilisierung von Zwischenprodukten und nicht durch zufälliges Ausprobieren. Da die Stabilisierung relativ schwach ist, können korrekte Zwischenprodukte verloren gehen, v.a. solche die früh im Faltungsprozess entstehen. 3.6.4 Die Vorhersage der 3D Struktur aus den Aminosäuren ist sehr schwierig. 3.6.5 Durch Modifikation und Spaltung erhalten Proteine neue Eigenschaften. Das Anfügen von Zuckerresten macht ein Protein hydrophiler, das Anfügen einer Fettsäure an eine α-Aminogruppe oder eine Cysteinsulfhydrylgruppe macht ein Protein hydrophober. Ein Gen bestimmt eine Polypeptidkette (Protein) Erforschung der Proteine (Kapitel 4, Seiten 77-104) 4.0.1 Proteom: funktionelle Expression von Information des Genoms. Art, Funktion und Interaktion von Proteinen die zu einer funktionellen Einheit gehören. 4.1 Die Aufreinigung eines Proteins ist der erste Schritt zum Verständnis seiner Funktion. 4.1.1 Assay: Test auf irgendeine besondere Eigenschaft des Proteins, die hilft das zu suchende Protein zu erkennen. Spezifische Aktivität: Verhältnis der Enzymaktivität zur Proteinmenge im Enzymassay. Ziel der Aufreinigung ist die Maximierung der spezifischen Aktivität. 4.1.2 Damit ein Protein aufgereinigt werden kann, muss es aus der Zelle freigesetzt werden. Homogenisat (aufgebrochene Zellen) Differenzierte Zentrifugation (ergibt Fraktionen abnehmender Dichte) © 2004 Ana Sesartic 11 4.1.3 Proteine lassen sich entsprechend ihrer Grösse, Löslichkeit, Ladung und Bindungsaffinität aufreinigen. Aussalzen: Löslichkeit der meisten Proteine wird durch hohe Salzkonzentrationen herabgesetzt. Dialyse: Trennung der Proteine von kleinen Molekülen durch semipermeable Membran. Gelfiltrationschromatographie: Auftrennung nach Grösse. Grosse Moleküle passieren die Säule schneller und verlassen sie zuerst, weil ihnen nur ein kleineres Volumen zugänglich ist. Ionenaustauschchromatographie: Trennung aufgrund der Nettoladung. Affinitätschromatographie: Affinität der Proteine zu bestimmten chemischen Gruppen. Isolierung von Transkriptionsfaktoren. Hochdruckflüssigkeitschromatographie: Grösseres Auflösungsvermögen und schnelle Auftrennung. 4.1.4 Gelelektrophorese: Molekül mit einer Nettoladung wandert in einem elektrischen Feld. Proteine werden getrennt und anschliessend sichtbar gemacht. Kleine Proteine wandern rasch durchs Gel, Grosse bleiben oben, nahe der Probenauftragsstelle. Isoelektrische Fokussierung: Trennung aufgrund des relativen Gehalts an sauren und basischen Resten. Isoelektrische Punkt (pI) = pH bei dem Nettoladung eines Proteins null beträgt. Zweidimensionale Gelelektrophorese: Kombination aus isoelektrischer Fokussierung und SDS-Polyacrylamidgelelektrophorese. 4.1.5 Quantitative Auswertung der Aufreinigung: Gesamtprotein Gesamtaktivität Spezifische Aktivität Ausbeute Reinheitsgrad 4.1.6 Die Ultrazentrifugation eignet sich zur Trennung von Biomolekülen und zur Bestimmung des Molekulargewichts. Der Sedimentationskoeffizient beschreibt Verhalten des Teilchens unter Einfluss der Zentrifugalkraft. Sedimentationsgeschwindigkeit ist bestimmt durch Masse, Form, Dichte des Teilchens und Dichte der Lösung. Zonen-, Banden-, bzw. Dichtegradientzentrifugation: Auftrennen von Proteinen mit unterschiedlichen Sedimentationskoeffizienten. Sedimentationsgleichgewichtszentrifugation: Bestimmung von Molekulargewichten unter nicht-denaturierenden Bedingungen, bei denen die Quartärstruktur erhalten bleibt. 4.1.7 Die Masse eines Proteins kann durch Massenspektrometrie präzise bestimmt werden. Die Proteinprobe wird durch einen Laserstrahl ionisiert. Ein elektrisches Feld beschleunigt die gebildeten Ionen Richtung Detektor. Die leichtesten Ionen erreichen den Detektor zuerst. Ausserdem wird die Flugzeit gemessen. © 2004 Ana Sesartic 12 4.2 Der Abbau von Proteinen nach Edman: Markierung des N-Terminus mit einem chemischen Agens. Danach Entfernung einer Aminosäure nach der anderen. Somit Ermittlung der Aminosäuresequenz. Wenn die Sequenz bekannt ist, kann man nach dem Gen suchen, welches das Protein kodiert hat. Man kann Proteine spezifisch in kleine Peptide zerlegen, um die Analyse zu erleichtern. Denn die Reaktionen des Edman-Abbaus, v.a. der Abspaltungsschritt laufen nicht zu 100% ab. Somit setzen pro Reaktionsschritt nicht alle Peptide im Reaktionsgemisch das Aminosäurederivat frei. Diagonale Elektrophorese: Über Disulfidbrücken verknüpfte Peptide werden in horizontale Richtung elektrophoretisch aufgetrennt und anschliessend mit Perameisensäure behandelt. Dann folgt eine Elektrophorese in vertikaler Richtung. Somit wird die ursprüngliche Position der Disulfidbrücken bestimmt. 4.2.1 Die Sequenz eines bestimmten Proteins kann mit allen anderen bekannten Sequenzen verglichen werden, um mögliche wichtige Ähnlichkeiten aufzuspüren. Der Vergleich von Sequenzen des gleichen Proteins in verschiedenen Arten liefert Informationen über Evolutionswege. Aminosäureanalysen können sich wiederholende Sequenzen aufspüren. Viele Proteine enthalten Aminosäuresequenzen, die als Signale wirken und den Bestimmungsort eines Proteins festlegen oder seine Prozessierung steuern. Sequenzdaten liefern die Grundlage für die Erzeugung spezifischer Antikörper gegen ein bestimmtes Protein. Aminosäuresequenzen sind wertvoll zur Herstellung von spezifischen DNASonden, die das Gen erkennen, welches das entsprechende Protein kodiert. 4.2.2 Genom- und Proteomanalyse sind komplementäre Ansätze, um die strukturelle Grundlage der Proteinfunktion aufzudecken. 4.3 Die Immunologie liefert wichtige Methoden zur Untersuchung von Proteinen. 4.3.1 Gegen ein Protein lassen sich spezifische Antikörper (Immunoglobuline) herstellen. Ein Antikörper erkennt auf einem grossen Molekül eine spezielle Gruppe von Aminosäuren, die antigene Determinante (= Epitop). Zellen produzieren viele verschiedene Antikörper, die jeweils ein anderes Oberflächenmerkmal desselben Antigens (fremdes Protein, Polysaccharid oder Nucleinsäure) erkennen (polyklonale Antikörper). © 2004 Ana Sesartic 13 4.3.2 Monoklonale Antikörper sind identisch und werden von Klonen produziert, die jeweils aus einer antikörperproduzierenden Zelle hervorgehen. Sie erkennen nur ein einzelnes Epitop und sind leicht herzustellen, wenn man kurzlebige antikörperproduzierende Zellen mit unsterblichen Myelomzellen fusioniert. Monoklonale Antikörper werden auch in vielen Assays verwendet (z.B. Nachweis von Isoenzymen im Blut nach Herzinfarkt oder Untersuchung von Bluttransfusionen auf AIDS). 4.3.3 Mithilfe eines enzymgekoppelten immunoassays (ELISA) lassen sich Proteine nachweisen und quantifizieren. Dabei wird ein Enzym, dass ein farbloses Substrat in ein farbiges Produkt umsetzt, an einen spezifischen Antikörper gekoppelt, der an das gesuchte Protein bindet. Indirekter ELISA: Intensität der Farbentwicklung als Mass für Menge an Antikörpern Nachweis für Vorhandensein von Antikörpern "Sandwich"-ELISA: Farbreaktion proportional zur Menge des Antigens Nachweis und Quantifizierung von Antigenen. 4.3.4 Western-Blotting erlaubt den Nachweis von per Gelelektrophorese aufgetrennten Proteinen. Proteine werden von Gel auf Polymerschicht übertragen und mit einem radioaktiven Antikörper markiert. Auf dem Autoradiogramm erscheint dann eine Bande, die dem gesuchten Protein entspricht. 4.3.5 Mit Fluoreszenzstoffen lassen sich Proteine in Zellen sichtbar machen. Somit kann man die Lokalisation eines Proteins in der Zelle aufzeigen. Ein grün fluoreszierendes Protein GFP wird aus einer Qualle gewonnen. © 2004 Ana Sesartic 14 Enzyme: Grundlegende Konzepte und Kinetik (Kapitel 8, Seiten 210-241) 8.1 Enzyme sind leistungsstarke und hochspezifische (bezüglich Reaktion und Reaktand) Katalysatoren. Sie beschleunigen Reaktionen ums Millionenfache. Proteolytische Enzyme katalysieren die Hydrolyse von Peptidbindungen (und Esterbindungen). Spezifität eines Enzyms hängt von der Wechselwirkung des Substrats mit dem Enzym ab. Dies resultiert aus der 3D Struktur des Enzymproteins 8.1.1 Viele Enzyme benötigen für Ihre Aktivität Cofaktoren. Apoenzym + Cofaktor = Holoenzym Cofaktoren sind Metalle oder kleine organische Moleküle, welche sich oft von Vitaminen ableiten. Wenn fest gebunden, nennt man sie prosthetische Gruppe. 8.1.2 Enzyme können verschiedene Energieformen ineinander umwandeln. Z.B. bei der Photosynthese 8.1.3 Enzyme klassifiziert man anhand der Rektionstypen die sie katalysieren. Oxidoreduktasen Oxidation-Reduktion Transferasen Gruppenübertragung Hydrolasen Hydrolysereaktion (Übertragung funktioneller Gruppen auf Wasser) Peptidbindung! Lyasen Hinzufügen / Entfernen von Gruppen zur Bildung von Doppelbindungen Isomerasen Isomerisierung (intramolekulare Gruppenübertragung) Ligasen Ligation zweier Substrate auf Kosten der ATP-Hydrolyse 8.2 Die freie Enthalpie ist eine wichtige thermodynamische Funktion zum Verständnis von Enzymen. 8.2.1 Die Änderung der freien Enthalpie liefert Informationen über die Spontaneität einer Reaktion, aber nicht über ihre Geschwindigkeit. ∆G < 0 spontan ∆G = 0 Gleichgewicht ∆G > 0 NICHT spontan ∆G ist vom Weg (molekularem Mechanismus) der Umwandlung unabhängig. Es sagt nichts über die Geschwindigkeit der Reaktion aus. © 2004 Ana Sesartic 15 8.2.2 Änderung der freien Enthalpie ∆G = ∆G° + RT ln [C ][ D] [ A][ B] wenn A + B R C + D ∆G hängt von der Art der Reaktionspartner und ihren Konzentrationen ab. ∆G°': Änderung der freien Standardenthalpie bei pH7. ∆G° ' = − RT ln K ' = −2.303RT log K ' −∆G K ' = 10 5.71 8.2.3 Enzyme können nur die Reaktionsgeschwindigkeit, aber nicht das Reaktionsgleichgewicht verschieben. Einstellung des Gleichgewichts hängt nur vom Unterschied zwischen der freien Enthalpie der Edukte und Produkte ab. 8.3 Enzyme beschleunigen Reaktionen durch Erleichterung der Bildung von Übergangszuständen (senken der Aktivierungsenergie). Übergangszustand hat höhere freie Enthalpie als Substrat oder Produkt. Aktivierungsenergie ist Differenz zwischen der freien Enthalpie des Übergangszustands und der des Substrats. Geschwindigkeit der Reaktion ist proportional zur Konzentration des Substrats im Übergangszustand. Das Wesen der Katalyse besteht in der spezifischen Bindung des Übergangs-zustandes. Enzyme stabilisieren den Übergangszustand durch selektive Bindung. Dadurch senken sie die Aktivierungsenergie für eine Reaktion. 8.3.1 Die Bildung eines Enzym-Substrat-Komplexes (ES-Komplex) ist der erste Schritt bei der enzymatischen Katalyse. Aktives Zentrum: Region des Enzyms an das die Substrate gebunden werden. Beweise für Existenz von ES-Komplexen Enzymkatalysierte Reaktionen erreichen eine Maximalgeschwindigkeit Hinweis auf Bildung definierter ES-Komplexe Röntgenstrukturanalysen aktiver Zentren Spektroskopische Eigenschaften der ES-Komplexe im Unterschied zu Enzymen und Substraten 8.3.2 Gemeinsame Eigenschaften der aktiven Zentren: Das aktive Zentrum (AZ) ist eine 3D Spalte, die von vielen Gruppen aus verschiedenen Abschnitten der Aminosäuresequenz gebildet wird. Das AZ stellt nur einen relativ kleinen Teil des Gesamtenzyms dar. Aktive Zentren sind höhlen- oder spaltenförmig Substrate werden durch viele schwache Kräfte an das Enzym gebunden Die Bindungsspezifität ist von der definierten Anordnung der Atome im aktiven Zentrum abhängig. © 2004 Ana Sesartic 16 Induced Fit: Die aktiven Zentren einiger Enzyme haben erst nach der Bindung des Substrats eine dazu komplementäre Form. Katalytische Gruppen: Aminosäurereste die direkt am Trennen / Bilden von Bindungen teilnehmen. 8.4 Michaelis-Menten-Modell ist nur auf Steady State (Fliessgleichgewicht) anzuwenden. Bei vielen Enzymen variiert die Katalysegeschwindigkeit V0 (welche als Anzahl pro Sekunde entstehenden Mole des Produkts definiert ist) mit der Substratkonzentration. Bei der Katalyse muss ein spezifischer ESKomplex als Zwischenprodukt auftreten. k1 k2 E + S R ES → E + P k−1 Annahme eines Steady State: V0 = Vmax Bei [ S ] = K m ist V0 = Vmax 2 [S ] [S ] + Km ; Km = k−1 + k2 k1 . Km entspricht also der Substratkonzentration, bei der die Reaktionsgeschwindigkeit die Hälfte ihres Maximalwerts erreicht. 8.4.1 Die Bedeutung der Km und Vmax Werte Km: Dissoziationskonstante des ES-Komplexes, wenn k2 sehr klein gegenüber k-1 ist. Mass für die Stabilität des ES-Komplexes ≈ Substratkonzentration in der Zelle. KM = Vmax/2 Vmax: Anzahl von Substratmolekülen, die – bei vollständiger Sättigung des Enzyms mit Substrat – pro Zeiteinheit in das Produkt umgewandelt werden. Nie real messbar. Reaktion 1. Ordnung bei tiefer Konzentration. Wenn alle aktiven Zentren gebunden sind, ist Vmax erreicht (konstant) Reaktion 0. Ordnung. 8.4.2 Das kinetische Optimum der enzymatischen Katalyse: Kkat/KM-Kriterium = Mass katalytischer Effizienz. Geschwindigkeitskonstante für Wechselwirkung. kkat [ E ][ S ] Km Wenn [ S ] K m , hängt die enzymatische Geschwindigkeit von den Werten V0 = für Kkat/KM, [S] und [E]T ab. kkat kkat k1 < k1 = K m kkat + k−1 Die Bildungsgeschwindigkeit des ES-Komplexes kann nicht grösser werden als die Geschwindigkeit der diffusionskontrollierten Begegnung von Enzym und Substrat. Kinetische Perfektion: Katalysegeschwindigkeit wird nur durch die Geschwindigkeit beschränkt, mit der sie in der Lösung ihrem Substrat begegnen. (Z.B. Cholinesterase; Reaktion nur diffusionslimitiert) kkat Km = 108 bis 109 = obere Limite der diffusionslimitierten Geschwindigkeit. Circe-Effekt: Nutzung von Anziehungskräften, um ein Substrat in ein Zentrum zu dirigieren, in dem es eine Strukturumwandlung erfährt. 8.4.3 Die meisten biochemischen Reaktionen beinhalten mehrere Substrate. Sequenzielle Verdrängung: zuerst binden alle Substrate an das Enzym, ehe das Produkt gebildet wird. Es bildet sich ein ternärer Komplex des Enzyms © 2004 Ana Sesartic 17 und beider Substrate, welcher nach der Katalyse aus dem Enzym und den Produkten besteht. Viele Enzyme, die NAD+ oder NADH als Substrat haben reagieren nach dem geordneten sequenziellen Mechanismus, ein Beispiel für zufälligen sequenziellen Mechanismus ist die Bildung von Keratinphosphat. Doppelte Verdrängung (Pingpong – Reaktion): Produkt(e) werden freigesetzt noch bevor alle Substrate an das Enzym gebunden sind. substituiertes Enzym-Zwischenprodukt in dem das Enzym zeitweilig modifiziert ist. Bsp. Reaktion: Austausch von Aminogruppen zwischen Aminosäuren und α-Ketosäuren. 8.4.4 8.5 Allosterische Enzyme gehorchen nicht der Michaelis-Menten-Kinetik. Sie können ihre katalytischen Eigenschaften anpassen und haben eine sigmoide Kurve (= S-Kurve) kooperative Substratbindung: Bindung des Substrats an ein aktives Zentrum des Enzyms erleichtert die Substratbindung an die anderen aktiven Zentren. Enzyme können durch spezifische Moleküle gehemmt werden. Irreversibler Inhibitor: fest gebunden, dissoziiert nur sehr langsam vom Enzym weg. Z.B. Aspirin, Penicillin Reversibler Inhibitor: schnelle Dissoziation des Enzym-InhibitorKomplexes Kompetitive Hemmung: kompetitiver Inhibitor vermindert die Katalysegeschwindigkeit indem er den Anteil der Enzymmoleküle mit gebundenem Substrat verringert; KM grösser. Enzym kann entweder Substrat oder Inhibitor binden, aber nicht beide. Kann durch genug hohe Substratkonzentration überwunden werden. [Abb.8.17 S.232] Nichtkompetitive Hemmung: Reversibel. Inhibitor und Substrat können gleichzeitig an unterschiedlichen Bindungsstellen von demselben Enzymmolekül gebunden werden, aber Enzym funktioniert nicht, da durch Inhibitorbindung Enzymform verändert wird. Erniedrigung der Wechselzahl eines Enzyms. Lässt sich nicht durch Erhöhung der Substratkonzentration aufheben. [Abb.8.18 S.232] Gemischte Hemmung: Inhibitor beeinträchtigt Substratbindung und Wechselzahl eines Enzyms. 8.5.1 Kompetitive und nichtkompetititve Hemmung lassen sich kinetisch Unterscheiden. Kompetitiv: kann durch ausreichend hohe Substratkonzentration überwunden werden. Nicht-Kompetitiv: lässt sich nicht durch Erhöhung der Substratkonzentration ausschalten. 8.5.2 Irreversible Inhibitoren können zur Untersuchung des aktiven Zentrums verwendet werden. Drei Kategorien von Irreversiblen Inhibitoren sind: © 2004 Ana Sesartic 18 Gruppenspezifische Reagenzien: reagieren mit spezifischen R-Gruppen von Aminosäuren. Substratanaloga / Affinitätsmarker: Moleküle, die dem Substrat des Enzyms strukturell ähnlich sind. Ahmt das normale Substrat nach, indem es sich ans AZ bindet und modifiziert das Enzym kovalent, sodass das Enzym irreversibel inhibitiert wird. Selbstmordinhibitoren / mechanismusbasierte Inhibitoren: modifizierte Substrate, mit denen sich ein aktives Zentrum eines Enzyms am spezifischsten modifizieren lässt. Zwischenprodukt inaktiviert das Enzym durch kovalente Modifikation. 8.5.3 Analoga des Übergangszustands sind starke Enzyminhibitoren. Z.B. die Racemisierung von Prolin verläuft über einen Übergangszustand, in dem der tetraedrische α-Kohlenstoff durch den Verlust eines Protons trigonal-planar wird. Der α-Kohlenstoff dieses Inhibitors ist trigonal-planar, genau wie der des Übergangszustands. Selektive Bindung des Übergangszustands (Übergangszustand wird stabilisiert)! 8.5.4 Katalytische Antikörper (Abzyme) demonstrieren die Wichtigkeit der selektiven Bindung des Übergangszustands für die Enzymaktivität. Es sind Antikörper, die Übergangszustände (als Antigen) erkennen, und als Katalysatoren fungieren. Abzyme stabilisieren den Übergangszustand und sind dadurch katalytisch wirksam. Bedeutung der Übergangszustandsanaloga: Vermitteln Einblicke in den Katalysemechanismus Wirkungsvolle und spezifische Enzyminhibitoren Immunogene, die neuartige Katalysatoren erzeugen 8.5.5 Penicillin hemmt irreversibel die Transpeptidase, ein Schlüsselenzym der Zellwandsynthese in Bakterien. Das reaktive Zentrum des Penicillins ist die Peptidbindung in seinem β-Lactamring. Das Penicillin hemmt die quervernetzende Transpeptidase indem es mit ihr reagiert und einen stabilen inaktiven Komplex bildet. Diese Penicilloyl-Enzym-Komplex reagiert nicht weiter. 8.6 Vitamine sind organische Moleküle, die in kleinen Mengen in der Nahrung einiger höherer Tiere vorkommen müssen. Ihre biosynthetischen Reaktionswege sind komplex. Es ist darum effizienter sie zu sich zu nehmen, als Enzyme zu synthetisieren, die zu ihrem Aufbau nötig sind. © 2004 Ana Sesartic 19 8.6.1 Wasserlösliche Vitamine fungieren als Coenzyme, so z.B. Ascorbinsäure (Vitamin C) und Vitamin-B-Komplex. Die B-Vitamine sind Bestandteile von Coenzymen. Alle Vitamine (ausser C) müssen modifiziert werden, ehe sie ihre Funktion erfüllen können. Kollagen, das in Abwesenheit von Ascorbat synthetisiert wird, ist weniger stabil als das normale Protein. Die durch ungenügende Hydroxylierung des Kollagens gebildeten abnormalen Fasern tragen zu Hautläsionen und der Brüchigkeit der Blutgefässe bei, die man bei Skorbut beobachtet. Vitamin C Mangel führt also zu Skorbut. 8.6.2 Fettlösliche Vitamine sind an so unterschiedlichen Prozessen wie der Blutgerinnung und dem Sehvorgang beteiligt. Vitamin K: Blutgerinnung Vitamin A: Sehpigmente, Wachstum Vitamin D: Knochenbildung Vitamin E: Fruchtbarkeit Katalytische Strategien (Kapitel 9, Seiten 249-268) 9 Serinprotease: (Bsp.Chymotrypsin) führen Reaktion durch, die bei neutralem pH und ohne Katalysator unmessbar langsam abläuft. Carboanhydrasen: müssen hohe absolute Reaktionsgeschwindigkeit erzielen, um mit anderen schnellen physiologischen Prozessen Schritt halten zu können. Restriktionsendonucleasen: (Bsp.EcoRV) hohe Spezifizität. Nucleosidmonophosphat-Kinasen (NMP-Kinasen): übertragen eine Phosphorylgruppe vom ATP auf ein Nucleotid. 9.0.1 Einige grundlegende katalytische Mechanismen sind vielen Enzymen gemeinsam: Kovalente Katalyse: das aktive Zentrum enthält eine reaktive Gruppe, die im Verlauf der Katalyse kovalent verändert wird. (Bsp. Chymotrypsin) © 2004 Ana Sesartic 20 Allgemeine Säure-Base-Katalyse: ein anderes Molekül als Wasser ist Protonendonor/-akzeptor. Metallionenkatalyse: Metallion als elektrophiler Katalysator, Erzeuger nucleophiler Gruppe, Erhöher der Bindungsenergie durch Bindung ans Substrat (Bsp. NMP-Kinase) Katalyse durch Annäherung: zwei Substrate auf einer Bindungsoberfläche des Enzyms und dadurch Erhöhung der Reaktionsgeschwindigkeit. 9.1 Proteasen ermöglichen eine schwer durchführbare Reaktion. Sie spalten Proteine durch eine Hydrolysereaktion (= Addition eines Wassermoleküls an eine Peptidbindung). Resonanzstruktur verleiht der Peptidbindung Doppelbindungs-charakter. Enzym, dass die Bindung spalten will, muss also nucleophilen Angriff auf die Carbonylgruppe ermöglichen. 9.1.1 Chymotrypsin besitzt einen hochreaktiven Serinrest. Es spaltet Peptidbindungen an der Carboxylseite von Aminosäuren mit aromatischen oder grossvolumigen hydrophoben Seitenketten. 9.1.2 Die Chymotrypsinreaktion (Hydrolyse durch Chymotrypsin) erfolgt in zwei Schritten, die über ein kovalent gebundenes Zwischenprodukt miteinander verknüpft sind. 1) Phase des schnellen Anstiegs (burst) vor Erreichen des Gleichgewichts Acylierung Acyl-Enzym-Zwischenprodukt 2) Gleichgewichtsphase: Deacylierung freies Enzym 9.1.3 Serin ist Teil einer katalytischen Triade mit Histidin und Aspartat. Über Aufbau von H-Brücken durch Histidin und Aspartat wird Reaktivität des Serins erhöht. Sie wandelt Serin in eine starke nucleophile Gruppe um, machen es also viel saurer. Histidin positioniert Serinkette und polarisiert deren Hydroxygruppe. Dadurch wirkt der Rest als Basenkatalysator (Akzeptor für HIonen). Der Aspartatrest stützt die Orientierung des Histidins und macht es zu besseren Protonenakzeptor. © 2004 Ana Sesartic 21 Peptidhydrolyse durch Chymotrypsin: 1) Substratbindung 2) Nucleophiler Angriff verändert Geometrie in Umgebung des C-Atoms. Tetraedrisches Zwischenprodukt entstanden. Ladung stabilisiert durch Oxyaniontasche 3) Zwischenprodukt zerfällt und Acyl-Enzym entsteht 4) Aminogruppe löst sich vom Enzym. 5) Wasser ersetzt Aminogruppe 6) Estergruppe des Acyl-Enzyms wird hydrolysiert 7) Carbonsäureprodukt bildet sich und wird freigesetzt Oxyaniontasche stabilisiert das tetraedrische Zwischenprodukt durch Stabilisierung der negativen Ladung im Enzym. H-brücken verknüpfen Peptid-NHGruppen mit dem negativ geladenen Sauerstoffatom. Hydrophobe Tasche von Chymotrypsin (S1-Tasche) ist verantwortlich für die Substratspezifizität. Die Bindung einer geeigneten Seitenkette in diese Tasche positioniert die daran anschliessende Peptidbindung im aktiven Zentrum für die Spaltung. 9.1.4 Katalytische Triaden kommen auch bei anderen hydrolytischen Enzymen vor. Trypsin: spaltet Peptidbindungen nach Aminosäuren mit langen, positiv geladenen Seitenketten (Arginin und Lysin). Elastase: spaltet Peptidbindungen nach Aminosäuren mit kleinen Seitenketten. 9.1.5 Die katalytische Triade wurde mithilfe ortspezifischer Mutagenese genau untersucht. 9.1.6 Cystein-, Aspartat- und Metalloproteasen sind weitere wichtige Klassen von peptidspaltenden Enzymen. Bei jeder dieser drei Enzymklassen besitzt das aktive Zentrum Merkmale, welche die Aktivierung von Wasser oder einer anderen nucleophilen Gruppe sowie die Polarisierung der Peptidcarbonylgruppe und die anschliessende Stabilisierung eines tetraedrischen Zwischenprodukts ermöglichen. Cysteinproteasen: Cystinrest, der von einem Histidinrest aktiviert wird, übernimmt die Funktion der nucleophilen Gruppe Aspartatproteasen: Paar von Asparaginsäureresten im aktiven Zentrum wirken zusammen und ermöglichen es einem Wassermolekül eine Peptidbindung anzugreifen. Metalloproteasen: Aktives Zentrum enthält eine gebundenes Metallion. 9.1.7 Proteaseinhibitoren sind wichtige Medikamente. Z.B. bei der Behandlung von Bluthochdruck, AIDS, etc. 9.2 Carboanhydrasen machen schnelle Reaktionen noch schneller. Z.B. Beschleunigen die CO2-Hydratisierung und helfen das CO2 leichter und schneller in der Lunge aus der gelösten Form im Blut ins Gas umzuwandeln und auszuatmen. 9.2.1 Carboanhydrasen enthalten ein gebundenes Zink-Ion, das für die katalytische Aktivität essenziell ist. Metallionen sind reaktiv weil: positiv geladen, verschiedene stabile Oxidationszustände, Bildung von starken aber kinetisch instabilen Bindungen. © 2004 Ana Sesartic 22 9.2.2 Bei der Katalyse kommt es zur Aktivierung eines Wassermoleküls durch Zink. Wasser wird dabei reaktiver (saurer) gemacht, da es an Zn2+ Zentrum gebunden ist. Die Bindung eines Wassermoleküls an Zink begünstigt die Bildung eines Übergangszustands, dabei kommt es durch die Freisetzung des Protons und durch Zusammenbringen der beiden Reaktanden zur Bildung von Hydrogencarbonat. 9.2.3 Ein Protonen-Shuttle ermöglicht die schnelle Regeneration der aktiven Enzymform. Histidin zieht von dem zinkgebundenen Wassermolekül ein Proton ab. Dadurch entsteht ein nucleophiles Hydroxyion und ein protoniertes Histidin. Der Puffer entfernt aus dem Histidinrest ein Proton, sodass wieder die deprotonierte Form entsteht. 9.2.4 Durch konvergente Evolution sind bei verschiedenen Carboanhydrasen aktive Zentren auf der Basis von Zink entstanden. α-Carboanhydrasen: zu menschlichen Enzymen homolog; bei Tieren, einigen Bakterien und Algen; drei Histidinreste β-Carboanhydrasen: bei höheren Pflanzen und zahlreichen Bakterien. Ein Histidinrest γ-Carboanhydrasen: bei Archaea; mit linksgängiger β-Helix Lipide und Zellmembranen (Kapitel 12, Seiten 349-374) 12 Biologische Membran definiert die innere und äussere Seite einer Zelle. Es ist eine dynamische Struktur, in der Proteine (Transportsystem, verleiht selektive Permeabilität) in einem Meer von Lipiden (Permeabilitätsschranke) schwimmen. Beim Metabolismus baut die Zelle einen Protonengradienten über die Membran auf. Ionengradienten sind eine Form der Energiekonservierung. Membranen schützen die Zelle und sorgen für Komparmentierung und Aufbau eines Ionengradienten. 12.1 Gemeinsame Merkmale der Membranen Blattartige, 2 Moleküle dicke Strukturen, die geschlossene Grenzen bilden. Bestehen hauptsächlich aus Lipiden und Proteinen, enthalten auch Kohlenhydrate. Membranlipide haben hydrophilen und hydrophoben Anteil. Lipiddoppelschichten sind Barrieren für die Passage polarer Moleküle. Spezifische Proteine vermitteln spezielle Membranfunktionen: Pumpen, Kanäle, Rezeptoren, Energieüberträger und Enzyme. Sind nicht-kovalente Molekülanordnungen. Sind asymmetrisch (Innen- und Aussenseiten unterscheiden sich). © 2004 Ana Sesartic 23 Sind flüssige Strukturen ("2D Lösungen gerichteter Proteine und Lipide") Meist elektrisch polarisiert (Innenseite negativ, -60mV) 12.2 Fettsäuren sind die Hauptbestandteile der Lipide und zuständig für ihr hydrophobes Verhalten. 12.2.1 Fettsäuren: Kohlenwasserstoffketten (KW-Ketten), die mit einer Carboxylgruppe enden. ω-Kohlenstoffatom: Kohlenstoff der Methylgruppe am äusseren Ende der Kette 12.2.2 Fettsäuren variieren in Kettenlänge und Sättigungsgrad. Am häufigsten sind Fettsäuren mit 16 und 18 C-Atomen. Ungesättigte Fettsäuren besitzen Doppelbindungen und haben einen niedrigeren Schmelzpunkt als gesättigte. Kurze Ketten und Doppelbindungen erhöhen die Fluidität der Fettsäuren und ihrer Derivate. 12.3 Lipide sind wasserunlösliche Biomoleküle, die in organischen Lösungsmitteln wie Chloroform sehr gut löslich sind. Sie dienen als Brennstoffmoleküle, hochangereicherte Energiespeicher, Signalmoleküle und Membrankomponenten. Die drei Hauptgruppen von Membranlipiden sind: Phospholipide, Glykolipide und Cholesterin. 12.3.1 Phospholipide stellen den grössten Anteil der Membranlipide. Phosphoglyceride: Glycerinskelett an das 2 Fettsäureketten und ein phosphoryliertes Alkohol gebunden sind. Die wichtigsten Phosphoglyceride entstehen aus Phosphatidat durch Bildung einer Esterbindung zwischen der Phosphatgruppe des Phosphatidats und der Hydroxylgruppe eines von mehreren Alkoholen. [Abb.12.4 und 12.5 S.353f.] Sphingomyelin: Hat Sphingosinskelett. Von ihm leiten sich kohlenhydrathaltige Glykolipide ab. 12.3.2 Die Membranen der Archaea enthalten Etherlipide mit verzweigten Alkylketten. Diese Verbindungen sind viel stabiler, was den Archaea u.a. erlaubt in extremen Bedingungen zu überleben. 12.3.3 Glykolipide enthalten Kohlenhydrateinheiten, die sich immer auf der extrazellulären Seite der Membran befinden. In tierische Zellen leiten sie sich von Sphingosin ab. [Abb.S.355] 12.3.4 Cholesterin ist ein Lipid mit einem Steroidgerüst (4 verbundene KW-Ringe). Es beeinflusst die Fluidität der Membranen. 12.3.5 Ein Membranlipid ist ein amphiphatisches Molekül mit einem hydrophilen und einem hydrophobem Anteil. © 2004 Ana Sesartic 24 12.4 Phospholipide und Glykolipide bilden in wässrigen Medien leicht bimolekulare Schichten. Die Membranbildung ist eine Konsequenz der amphipathischen Natur dieser Moleküle. Die bevorzugte Struktur der meisten Phospho- und Glykolipide in wässrigen Medien ist nicht die Micelle, sondern die Lipiddoppelschicht. Dies ist ein Prozess der Selbstaggregation (selfassembly). V.a. hydrophobe Kräfte sind für die Bildung von Lipiddoppelschichten verantwortlich. Van-der-Waals Kräfte zwischen den KWSchwänzen begünstigen eine enge Packung der Ketten. Es bilden sich auch elektrostatische Bindungen und Wasserstoffbrücken zwischen den polaren Köpfen und den Wassermolekülen der Umgebung aus. Micelle (20nm) Doppelmembran (3nm) Liposom (50nm) Lipiddoppelschichten sind kooperative Strukturen: Bestrebt sich auszubreiten Neigen zum Zusammenschluss mit sich selbst Sind selbstreparierend 12.4.1 Aus Phospholipiden können Lipidvesikel (= Liposomen) entstehen, wässrige Kompartimente, die von einer Lipiddoppelschicht umgeben sind. Ionen oder Moleküle können im wässrigen Inneren von Liposomen eingeschlossen werden und in der Form in Zellen eingeschleust werden. Dies bietet eine Möglichkeit zur medizinischen Anwendung von Liposomen. 12.4.2 Lipiddoppelschichten sind für Ionen und die meisten polaren Moleküle nicht permeabel. Ausnahme: Wasser kann Membran durchqueren. Die Permeabilitätskoeffizienten (Fähigkeit, die Lipiddoppelschicht zu durchqueren) kleiner Moleküle stehen mit dem Verhältnis ihrer Löslichkeit in unpolaren Lösungsmitteln zu der in Wasser in Beziehung. 12.5 Proteine bewerkstelligen die meisten Prozesse an Membranen. Allgemein enthalten Membranen mit verschiedenen Funktionen unterschiedliche Proteine. Proteinkomponenten von Membranen lassen sich durch SDSPolyacrylamidgelelektrophorese identifizieren. Austauschprozesse werden durch Membranproteine vermittelt. Diese müssen auf der Aussenseite apolar (hydrophob) sein, um sich in der Membran einzunisten. 12.5.1 Proteine sind in der Lipiddoppelschicht unterschiedlich angeordnet. Integrale Membranproteine stehen in intensiver Wechselwirkung mit dem KW-Bereich der Lipiddoppelschicht. Periphere Membranproteine binden an die Oberfläche integraler Membranproteine, bzw. treten in Wechselwirkung mit den polaren Kopfgruppen der Lipide. © 2004 Ana Sesartic 25 12.5.2 Wechselwirkungen zwischen Proteinen und Membranen Proteine können die Membran mit (apolaren) α-Helices durchziehen (am häufigsten) Ein Kanalprotein kann aus β-Strängen gebildet werden (z.B. ein Porin). Innenseite hydrophil wässriger Kanal Eine teilweise Einbettung eines Proteins in die Membran kann zu seiner Bindung an die Membranoberfläche führen. 12.5.3 Kovalent gebundene hydrophobe Gruppen (Palmitoylgruppe, Farnesylgruppe, Glykosylphosphatidylinositol-(GPI-)Anker) verbinden Proteine mit Membranen. 12.5.4 Transmembranhelices können aus Aminosäuresequenzen exakt vorausgesagt werden. 12.6 Lipide und viele Membranproteine diffundieren schnell in der Membranebene. Ein Lipidmolekül kann so z.B. in einer Sekunde von einem Ende eines Bakteriums zum anderen gelangen. Einige Proteine sind fast so beweglich wie Lipide, andere sind praktisch immobil. 12.6.1 Flüssigmosaikmodell erlaubt laterale Bewegung in der Membran, aber keinen Wechsel der Membranseite. Membranen sind 2D Lösungen gerichteter Lipide und globulärer Proteine. Proteine machen keine Flip-Flops! Daher kann die Membranasymmetrie über lange Zeiträume erhalten bleiben. Laterale Diffusion: Bewegung entlang der Membranebene. Ist wesentlich schneller als die transversale (Flip-Flop). Transversale Diffusion (Flip-Flop): Wanderung eines Moleküls von einer Membranoberfläche zur anderen. 12.6.2 Fluidität der Membran wird durch Fettsäurezusammensetzung und Cholesteringehalt geregelt. Kurze Ketten und Doppelbindungen (ungesättigte Fettsäuren) erhöhen die Fluidität (senken den Schmelzpunkt). Übergangstemperatur fest flüssig (Tm) hängt von der Länge der Fettsäureketten und ihrem Sättigungsgrad ab. Tm sinkt, wenn eine cis-Doppelbindung einen Knick in der KW-Kette verursacht. Bakterien regulieren die Fluidität ihrer Membranen durch Variation der Doppelbindungszahl und der Länge der Fettsäureketten. Bei Tieren ist Cholesterin der Hauptregulator der Membranfluidität. 12.6.3 Alle biologischen Membranen sind asymmetrisch (innere und äussere Ober-flächen haben unterschiedliche Bestandteile und Enzymaktivitäten). © 2004 Ana Sesartic 26 Bsp.: Na+-K+-Pumpe. Membranen werden stets durch Erweiterung bereits vorhandener Membranen synthetisiert. 12.7 Eukaryontenzellen enthalten Kompartimente, die von inneren Membranen umgeben sind. Periplasma: Region zwischen zwei Membranen (enthält die Zellwand). Eukaryontenzellen (mit Ausnahme von Pflanzenzellen) besitzen keine Zellwände, ihre Zellmembranen bestehen aus einer einzigen Lipiddoppelschicht. In Eukaryontenzellen finden sich Doppelmembranen bei folgenden Organellen: ER, Mitochondrien, Zellkern. Bei Bakterien mit zwei Membranen enthält die äussere Porine, die für alle frei diffundierbar sind. Die Innere Membran enthält selektive Membranproteine. 12.7.1 Proteine werden durch Signalsequenzen zu spezifischen Kompartimenten gelenkt. 12.7.2 Membranknospung (budding) und –fusion bestimmen viele wichtige biologische Prozesse. Dadurch können z.B. einige Kompartimente Material austauschen: Rezeptorvermittelte Endocytose ist wichtig beim Cholesterinstoffwechsel o LDL bindet an Rezeptor o Einstülpung des Komplexes Vesikel o Vesikel verschmilzt mit Lysosom, dass den Abbau des LDL und die Freisetzung des Cholesterins bewirkt. Neurotransmitterfreisetzung: Synaptische Vesikel verschmelzen mit Plasmamembran und setzen Neurotransmitter in den Spalt frei. Der Stoffwechsel: Konzepte und Grundmuster (Kapitel 14, Seiten 407-427) 14.0.1 Zellen wandeln verschiedene Formen von Energie ineinander um. Energie dient zur: Ausführung mechanischer Arbeit Aktiven Transport von Molekülen Synthese von Makromolekülen und anderen Biomolekülen aus einfachen Vorstufen Phototrophe Organismen wandeln Lichtenergie in chemische Energie um. Chemotrophe Organismen erhalten chemische Energie (welche in mechanische Energie umgewandelt werden kann) durch Oxidation von Nährstoffen, die durch phototrophe erzeugt wurden. 14.1 Der Metabolismus = Stoffwechsel = Umwandlung von Molekülen, besteht aus vielen gekoppelten Reaktionen. Katabolismus: Umwandlung von Brennstoffen in zelluläre Energie. Brennstoffe (Kohlenhydrate, Fette) © 2004 Ana Sesartic Katabolismus → CO2 + H 2O + nutzbare Energie 27 Anabolismus: Synthese; benötigt Energie Nutzbare Energie + kleine Moleküle Anabolismus → komplexe Moleküle Amphibolisch: Stoffwechselwege, die je nach Energiebedingungen in der Zelle anabolisch oder katabolisch sein können. 14.1.1 Eine thermodynamisch ungünstige Reaktion kann durch eine günstige Reaktion, die mit ihr gekoppelt ist, angetrieben werden. Die Gesamtänderung der freien Enthalpie einer chemisch gekoppelten Serie von Reaktionen entspricht der Summe der Änderungen der freien Enthalpie der Einzelschritte. Metabolische Stoffwechselwege entstehen durch enzymatisch katalysierte Reaktionen, die derart gekoppelt sind, dass die freie Enthalpie des Stoffwechselweges insgesamt negativ ist. 14.1.2 Adenosintriphosphat (ATP) ist ein Energiereiches Molekül, weil seine Triphosphat-Einheit zwei PhosphorsäureanhydridBindungen enthält. Der ATPADP-Zyklus ist der fundamentale Mechanismus des Energieaus-tausches in biologischen Systemen. Zwei wichtige Elektronenüberträger, NAD+ und FAD, sind Derivate des ATP. 14.1.3 Die ATP-Hydrolyse treibt den Metabolismus indem sie das Gleichgewicht gekoppelter Reaktionen verschiebt. Die Hydrolyse von n ATP-Molekülen ändert das Gleichgewichtsverhältnis einer gekoppelten Reaktion (oder Reaktionsfolge) um einen Faktor von 108n. Eine thermodynamisch ungünstige Reaktionsfolge kann also durch Kopplung mit der Hydrolyse einer ausreichenden Anzahl ATP-Moleküle in eine thermodynamisch günstige umgewandelt werden. 14.1.4 Die strukturelle Grundlage für das hohe Phosphorylgruppenübertragungspotenzial des ATP: Resonanzstabilisierung Elektrostatische Abstossung (bei Hydrolyse des ATP vermindert) Stabilisierung aufgrund Hydratation Aus diesen drei Gründen entsteht bei der Hydrolyse der Phosphorylsäureanhydridbindungen viel freie Enthalpie. 14.1.5 Das Phosphorylgruppenübertragungspotenzial ist eine wichtige Form der Energieumwandlung in der Zelle. Kreatinphosphat + ADP + H+ 14.2 Kreatin − Kinase U ATP + Kreatin Die Oxidation von Kohlenstoffverbindungen ist für die Zelle eine wichtige Energiequelle. ATP dient als unmittelbarer Donor freier Enthalpie. © 2004 Ana Sesartic 28 14.2.1 Verbindungen mit hohem Phosphorylgruppenübertragungspotenzial können die Kohlenstoffoxidation an die ATP-Synthese koppeln. Die Oxidationsenergie wird anfänglich in einer energiereichen Phosphatverbindung eingefangen und dann zur Bildung von ATP genutzt. 14.2.2 Ionengradienten über eine Membran sind eine wichtige Form zellulärer Energie, die an die ATPSynthese gekoppelt werden kann. Protonengradienten, aufgebaut aus Energie aus Oxidation von Brennstoffen, können die ATPSynthese antreiben. (= oxidative Phosphorylierung). 14.2.3 Die einzelnen Abschnitte der Energiegewinnung aus Nahrungsstoffen: 1) Abbau grosser Moleküle (Polymer) zu kleineren Einheiten (Monomer) 2) Abbau kleiner Moleküle (Monomer) zu einfachen Einheiten (Oxidation zu Acetyl-CoA), die zentrale Rolle im Stoffwechsel spielen. 3) ATP entsteht aus der vollständigen Oxidation der Acetylgruppe des Acetyl-CoA. 14.3.1 Aktivierte Carrier sind charakteristisch für den modularen Aufbau und die Wirtschaftlichkeit des Stoffwechsels. ATP ist ein aktivierter Carrier von Phosphorylgruppen, da die Phosphorylgruppenübertragung von ATP exergonisch ist. 1) Aktivierte Elektronen-Carrier für die Brennstoffoxidation Pyridinnucleotide oder Flavine. Bei der Oxidation eines Substrats nimmt der Nicotinamidring des NAD+ ein Wasserstoffion und zwei Elektronen auf, was einem Hydridion entspricht. Flavinadeninnucleotid (FAD) nimmt zwei Protonen und zwei Elektronen auf. 2) Ein aktivierter Elektronen-Carrier für reduktive Biosynthesen NADPH wird fast ausschliesslich für reduktive Biosynthesen verwendet, NADH dagegen in erster Linie zur Erzeugung von ATP. 3) Ein aktivierter Carrier von C2-Fragmenten Coenzym A (CoA) überträgt Acylgruppen. Acetyl-CoA hat ein hohes Acetylgruppenübertragungspotenzial, da der Transfer der Acetylgruppe exergonisch ist. Die kinetische Stabilität der Carrier in Abwesenheit von spezifischen Katalysatoren ist eine Voraussetzung für ihre biologische Funktion, da sie Enzymen ermöglicht, den Fluss von freier Enthalpie und Reduktionsäquivalenten zu kontrollieren. 14.3.2 Schlüsselreaktionen wiederholen sich im Stoffwechsel. 1) Redox Elektronenübertragung 2) Ligation (mit ATP Spaltung) Bildung kovalenter Bindungen (z.B. C-C) 3) Isomerisierung Umordnung von Atomen zur Isomerbildung 4) Gruppentransfer Übertragung einer funktionellen Gruppe von einem Molekül auf ein anderes 5) Hydrolyse Bindungsspaltung durch Addition von Wasser. Z.B. Verdauung von Proteinen = Hydrolyse der Peptidbindung 6) Addition oder Abspaltung funktioneller Gruppen an bzw. zur Ausbildung von Doppelbindungen; katalysiert durch Lyasen. Z.B. ein Schritt der Glykolyse © 2004 Ana Sesartic 29 Wichtige Reaktionen des Stoffwechsels: Citratzyklus Fettsäureabbau Fettsäuresynthese Lysinabbau 14.3.3 Stoffwechselprozesse werden reguliert durch: 1) Enzymmenge (durch Transkriptionsrate der Gene angepasst) 2) Enzymaktivität (durch reversible allosterische Kontrolle (Rückkopplungshemmung) und reversible kovalente Modifikation; Hormone koordinieren metabolische Beziehungen) 3) Verfügbarkeit von Substraten (Kontrolle des Substratflusses) a. Aufbauende und abbauende Stoffwechselwege sind fast immer getrennt b. Kompartimentierung trennt entgegengesetzte Reaktionen voneinander 4) Energiezustand a. Hohe ATP Konzentration hemmt ATP-erzeugende (katabole) Stoffwechselwege und fördert ATP-verbrauchende (anabole) Stoffwechselwege b. Die Energieladung einer Zelle ist gepuffert 1 [ ATP ] + [ ADP ] 2 Energieladung = [ ATP ] + [ ADP ] + [ AMP] [ ATP ] Phosphorylierungspotenzial = [ ADP][ Pi ] 14.3.4 ADP ist ein entwicklungsgeschichtlich altes Stoffwechselmolekül. Es findet sich wieder als Baustein in ATP, NADH, FAD und CoA. Kohlenhydrate (Kapitel 11, Seiten 323-334) 11 Hauptfunktionen der Kohlenhydrate Energiespeicher, Brennstoffe und Metaboliten Teile des Grundgerüsts von RNA und DNA (Ribose / Desoxyribose) Strukturelemente in Zellwänden von Bakterien und Pflanzen (Polysaccharide Cellulose, Chitin) Mit Proteinen und Lipiden verbunden (Glykoproteine) 11.1 Monosaccharide sind Aldehyde oder Ketone mit vielen Hydroxylgruppen (C-H2O)n ; Aldose (ROCH = Aldehyd), Ketose (ROCR' = Keton) D-Zucker: letztes OH zeigt nach rechts L-Zucker: letztes OH zeigt nach links © 2004 Ana Sesartic 30 Diastereomere: Isomere, die nicht wie Enantiomere Bild und Spiegelbild zueinander sind. Epimere: Kohlenhydrate, deren Konfiguration sich nur an einem asymmetrischen Zentrum unterscheidet. [Chiral: 4 verschiedene Substituenten] Ketosen haben ein asymmetrisches Zentrum weniger als Aldosen mit der gleichen Anzahl C-Atome. 11.1.1 Pentosen und Hexosen zyklisieren zu Furanose- und Pyranoseringen. Beim Ringschluss gibt es bei beiden ein chirales Zentrum mehr. Halbacetal = Aldehyd + Alkohol Halbketal = Keton + Alkohol Bildung der Pyranoseformen: D-Glucose in Kettenform zyklisiert, wenn C-5-Hydroxylgruppe die C-1Aldehydgruppe unter Bildung eines intramolekularen Halbacetals angreift. Dabei können α- und β-Anomere entstehen. Bildung der Furanoseformen: Offenkettige Fructose zyklisiert, wenn die C-5-Hydroxylgruppe die C-2Ketogruppe unter Bildung eines intramolekularen Halbketals angreift. Dabei können α- und β-Anomere (hemiacetal) entstehen. α: Hydroxylgruppe am C-1 unterhalb der Ringebene β: Hydroxylgruppe am C-1 oberhalb der Ringebene © 2004 Ana Sesartic 31 11.1.2 Axial: Bindung senkrecht zur Ringebene Substituenten behindern sich gegenseitig Äquatorial: Bindung parallel zur Ringebene Substituenten haben Platz Sesselkonformation der β-D-Glucopyranose ist stabiler als die Wannenform, da die sterische Hinderung geringer ist, wenn die axialen Positionen von HAtomen besetzt sind. Furanose Ringe könne sich zur Briefumschlag (envelope) Konfiguration falten. 11.1.3 Kohlenhydrate sind mit Alkoholen und Aminen durch glykosidische Bindungen zwischen anomerem C-Atom der Glucose und dem O-Atom der Hydroxylgruppe verknüpft. Es gibt O- und N-Glykosidische Bindungen. Der Ersatz von Hydroxylgruppen durch andere Substituenten führt zu modifizierten Monosacchariden. Man findet sie häufig auf Zelloberflächen. Lösungen mit Kupferionen (Fehling'sche Lösung) sind Nachweis für Glucose. 11.2 Komplexe Kohlenhydrate entstehen durch Verknüpfung von Monosacchariden. Oligosaccharide: entstehen durch Verknüpfung von mehreren Monosacchariden über O-Glykosidische Bindungen. 11.2.1 Disaccharide Saccharose: Rohrzucker, besteht aus Glucose und Fructose Lactose: Milchzucker, besteht aus Galactose und Glucose Maltose: zwei Glucosen mit α-1,4-glykosidische Bindung Enzyme Saccharase, Lactase und Maltase befinden sich auf der äusseren Oberfläche der Epithelzellen des Dünndarms 11.2.2 Polysaccharide: grosse polymere Oligosaccharide. Man nennt sie Homopolymere, wenn sie aus gleichen Monosaccharideinheiten bestehen. Glykogen ist die Speicherform der Glucose und das häufigsten Homopolymer tierischer Zellen. Glykogen und Stärke sind mobilisierbare Glucosespeicher. Stärke ist der Nährstoffreservoir der Pflanzen, ein Glucose Polymer bestehend aus aneinander gehängter Maltose (α-1,4). Hydrolysiert durch αAmylase, das von den Speichelzellen und vom Pankreas sezerniert wird. Amylose: unverzweigt, α-1,4-Bindung Amylopektin: verzweigt, eine α-1,6 auf 30 α-1,4-Bindungen [Dextran ist ein Glucosepolymer. Ein mikrobielles Polysaccharid, das von einigen Bakterien durch das Enzym Dextransaccharase mit Saccharose als Ausgangsmaterial synthetisiert wird.] [Chitin ist ein N-Acetylglucosaminpolymer (β-1,4-). Pilze haben es in ihren Zellwänden, bei Insekten bildet es das Exoskelett und bei Pflanzen ist es in Blättern und Samen vorhanden.] 11.2.3 Cellulose, das wichtigste strukturbildende Polymer der Pflanzen und die am häufigsten auftretende organische Verbindung in der Biosphäre, besteht aus linearen Ketten von Glucoseeinheiten, verknüpft durch β-1,4-Bindungen, welche die Bildung sehr langer gerader Ketten ermöglichen. Ketten sind durch H-Brücken stabilisiert. © 2004 Ana Sesartic 32 11.2.4 Glykosaminoglykane sind anionische Polysaccharidketten aus repetitiven Disaccharideinheiten, die ein Derivat eines Aminozuckers enthalten. Mindestens ein Kohlehydrat im Disaccharid besitzt eine negativ geladene Carboxylat- oder Sulfatgruppe. Sie sind gewöhnlich an Proteine gebunden und bilden Proteoglykane, welche als Gleitmittel dienen, Strukturbestandteile des Bindegewebes sind, Adhäsion von Zellen vermitteln und Zellproliferation stimulieren. 11.2.5 Für die Oligosaccharidsynthese sind Glykosyltransferasen verantwortlich. Sie katalysieren die Bildung der glykosidischen Bindungen. Monosaccharidnucleotide sind häufig Zwischenprodukte. Das anzufügende Kohlenhydrat stammt von einem Monosaccharidnucleotid. Beispiel für Sinn der Glykosyltransferasen: AB0-Blutgruppensystem, abhängig davon, was für ein Zucker an Erythrocyten-Oberfläche hängt. 0-Antigen: inaktive Glykosyltransferase A-Antigen: N-Acetylgalactosamin angehängt B-Antigen: Galactose angehängt Glykolyse und Gluconeogenese (Kapitel 16, Seiten 465491) 16 Glykolyse: Folge von anaeroben Reaktionen, in denen ein Molekül Glucose zu 2 Molekülen Pyruvat umgewandelt wird und gleichzeitig 2 Moleküle ATP entstehen. Gluconeogenese: Synthetisierung von Glucose aus Vorstufen, die keine Kohlenhydrate sind. 16.0.1 Glucose ist für die meisten Organismen ein wichtiger Brennstoff. 16.0.2 Gärung (Fermentation) erzeugt ATP in Abwesenheit von Sauerstoff. Organische Verbindungen fungieren sowohl als e—Donoren als auch als Akzeptoren. NADH muss wieder in NAD+ umgewandelt werden. Darum wird Pyruvat weiterverarbeitet. Ein Teil der Glucose oxidiert, danach gibt es eine Reduktion. Erzeugt nur geringe Energiemengen. Skelettmuskel erzeugt Milchsäure, wenn der Energiebedarf den Sauerstofftransport übersteigt. Obligate Anaerobier: können bei Anwesenheit des hochreaktiven O2 nicht überleben. (z.B. einige Bakterien Clostridium tetani) © 2004 Ana Sesartic 33 Fakultative Anaerobier: können mit und ohne O2 leben. Z.B. Bewohner der Gezeitenzone (Muschel Mytilus) zeigen lebensraumabhängige Anaerobiose. 16.1 Stufen der Glykolyse. Glykolyse findet im Cytosol statt. 1) Glucose (Phosphorylierung, Isomerisierung, Phosphorylierung) Fructose-1,6-Biphosphat. Glucose wird in Zelle eingefangen und in eine Verbindung überführt, die sich leicht in phosphorylierte C3-Einheiten spalten lässt. 2) Fructose-1,6-Biphosphat (C6-Kohlenhydrat) wird von Aldolase in zwei C3Fragmente gespalten, die leicht ineinander umwandelbar sind. 3) ATP wird erzeugt wenn die C3-Fragmente zu Pyruvat oxidiert werden. 1) Glucose wird eingefangen und destabilisiert 2) Durch Spaltung der C6-Einheit Fructose werden ineinander umwandelbare C3-Moleküle erzeugt 3) ATP wird erzeugt © 2004 Ana Sesartic 34 16.1.1 Die Hexokinase fängt Glucose in der Zelle ein und beginnt die Glykolyse. Stufe 1: Phosphorylierung der Glucose durch Hexonkinase Hexokinase – und alle anderen Kinasen – ist nur aktiv, wenn Mg2+ Ionen vorhanden sind. Das divalente Metallion bildet einen Komplex mit dem ATP. Induced Fit Die Substratinduzierte Schliessung einer Spalte ist eine allgemeine Eigenschaft von Kinasen. 16.1.2 Bildung von Fructose-1,6-Biphosphat aus Glucose-6-Phosphat durch Isomerisierung und Phosphorylierung. G-6-P → F-6-P : Isomerisierung; Umwandlung einer Aldose in eine Ketose ATP F-6-P → F-1,6-BP : Phosphorylierung; Katalysiert durch Phosphofructokinase (PFK) 16.1.3 Stufe 2: Die Aldolase spaltet das C6-Kohlenhydrat in zwei C3-Fragmente. 16.1.4 Die Triosephosphat-Isomerase (TIM) gewinnt ein C3-Fragment zurück. Isomerisierung einer Ketose zu einer Aldose verläuft über ein EndiolZwischenprodukt. TIM ist ein Beispiel für ein kinetisch perfektes Enzym. Der geschwindigkeitsbestimmende Schritt ist die diffusionskontrollierte Begegnung zwischen Enzym und Substrat. Katalytischer Mechanismus der TIM: Glutamat 165 überträgt mithilfe von Histidin 95 ein Proton zwischen CAtomen. Histidin wechselt dabei zwischen der neutralen und der relativ seltenen negativ geladenen Form. Die letztere wird durch Wechselwirkungen mit anderen Teilen des Enzyms stabilisiert. 16.1.5 Energieumwandlung: Über ein Thioester-Zwischenprodukt (kovalent ans Enzym gebunden) sind Phosphorylierung und Oxidation des Glycerinaldehyd3-Phosphats miteinander gekoppelt. Oxidation einer Aldehyd- zu einer Carboxylgruppe durch NAD+ (begünstigt) Phosphorylierung: Vereinigung einer Carboxylgruppe mit Orthophosphat zu einem Acylphosphat (ungünstig) 16.1.6 Stufe 3: Die Bildung von ATP aus 1,3-Biphosphatglycerat. 1) Ein Aldehyd wird zu einer Carbonsäure oxidiert 2) NAD+ wird zu NADH reduziert 3) ATP entsteht aus Pi und ADP auf Kosten der C-Oxidation 16.1.7 Die Erzeugung eines weiteren ATP und die Bildung von Pyruvat: 1) Umlagerung durch Phosphoglycerat-Mutase Mutase: ein Enzym, das den intramolekularen Transfer einer chemischen Gruppe katalysiert. 2) Bildung eines Enol Das hohe Phosphorylgruppen-Übertragungspotenzial des Phosphoenolpyruvats beruht auf der treibenden Kraft der nachfolgenden Enol-KetonUmwandlung © 2004 Ana Sesartic 35 16.1.8 Der Energiegewinn aus der Umwandlung von Glucose in Pyruvat: Bei der Überführung von Glucose in zwei Moleküle Pyruvat werden zwei Moleküle ATP gewonnen. Glukose + 2 Pi + 2 ADP + 2 NAD + → 2 Pyruvat + 2 ATP + 2 NADH + 2 H + + 2 H 2O 16.1.9 Die Aufrechterhaltung des Redoxgleichgewichts: Die unterschiedliche Verwertung des Pyruvats. In Reaktionen, an denen NADH beteiligt ist, können Ethanol oder Lactat entstehen. Alternativ kann eine C2-Einheit aus dem Pyruvat an das CoA gebunden werden, wobei Acetyl-CoA entsteht. [siehe S.479] 1) In der Hefe entsteht aus Pyruvat Ethanol I Decarboxylierung des Pyruvats II Acetaldehyd durch NADH zu Ethanol reduziert. Dieser Prozess regeneriert NAD+. Alkoholische Gärung: Umwandlung von Glucose in Ethanol. Keine Netto-Oxidations-Reduktions-Reaktion. 2) Milchsäuregärung = Lactat entsteht in verschiedenen Mikroorganismen aus Pyruvat Die Regenerierung des NAD+ bei der Reduktion von Pyruvat zu Lactat oder Ethanol hält den kontinuierlichen Ablauf der Glykolyse unter anaeroben Bedingungen aufrecht. Dabei wird nur ein kleiner Teil der in Glucose enthaltenen Energie gewonnen. 3) Acetyl-CoA entsteht in den Mitochondrien durch oxidative Decarboxylierung aus Pyruvat. Bei aeroben Bedingungen entsteht über den Citratzyklus und die Atmungskette am meisten Energie. 16.1.10 Die NAD+-Bindungsstelle ist bei vielen Dehydrogenasen sehr ähnlich. Rossmann-Falte, gebildet durch die nicotinamid- und adeninbindende Hälften. Das NAD+-Molekül lagert sich in ausgestreckter Konformation an. © 2004 Ana Sesartic 36 16.1.11 Ein grossteil der aufgenommenen Fructose wird in der Leber über den Fructose-1-Phosphat Weg metabolisiert. Da Leber und Muskulatur mehr Glucose als Fructose phosphorylieren, erhält das Fettgewebe mehr Fructose als Glucose. Für die Galactose gibt es keine katabolen Stoffwechselwege, sie wird deshalb in einen Metaboliten der Glucose umgewandelt. Die Umwandlung von UDP-Glucose in UDP-Galactose ist zur Synthese der Galactoseeinheiten in komplexen Polysacchariden und Glykoproteinen notwendig, wenn der Anteil der Galactose in der Nahrung für diesen Bedarf nicht ausreicht. 16.1.12 Viele Erwachsene vertragen keine Milch, weil ihnen die Lactase fehlt. Weil sie Lactose nicht abbauen können, vergären Mikroorganismen im Dickdarm diese und erzeugen dabei auch Methan und H2-£Gas, was zu Blähungen führt. Die ausserdem entstehende Milchsäure ist osmotisch aktiv und führt (wie auch die unverdaute Lactose) zum Wassereinstrom in den Darm, was Durchfall als Folge hat. Lactoseintoleranz 16.1.13 Wenn die Transferase fehlt (Galactosämie) ist Galactose stark toxisch. Z.B. Katarakte (Trübung der Augenlinse = "grauer Star") entstehen, wenn die Transferase in der Augenlinse nicht aktiv ist. 16.2 Die Glykolyse wird streng kontrolliert. Im Stoffwechsel stellen Enzyme, die weitgehend irreversible Reaktionen katalysieren, potenzielle Kontrollpunkte dar. 16.2.1 Die Phosphofructokinase (PFK) ist das Schlüsselenzym bei der Kontrolle der Glykolyse in Säugetieren. Die Aktivität des Enzyms steigt an (Glykolyse wird angeregt), wenn der ATP/AMP-Quotient kleiner wird (die Energieladung der Zelle sinkt). PFK wird von Citrat gehemmt, denn hoher Citratspiegel bedeutet genügend Biosynthesevorstufen sind vorhanden und keine weitere Glucose muss abgebaut werden. Fructose-2,6-biphosphat (F-2,6-BP) ist ein allosterischer Aktivator der PFK, der das Konformationsgleichgewicht dieses tetrameren Enzyms von der T- zur R-Form verschiebt. 16.2.2 Ein reguliertes bifunktionelles Enzym (PFK2 und FBPase2) synthetisiert F2,6-BP und baut es ab. Dieses bifunktionelle Enzym besteht aus einer Nterminalen regulatorischen Domäne, gefolgt von einer Kinase- und einer Phosphatasedomäne. Feedforward-Stimulierung: Der Überschuss an F-6-P führt zu höheren Konzentrationen von F-2,6-BP, was wiederum die PFK stimuliert. Aktivitäten von PFK2 oder FBPase2 werden durch Phosphorylierung eines einzigen Serinrestes reziprok kontrolliert. © 2004 Ana Sesartic 37 16.2.3 Hexokinase und Pyruvat-Kinase bestimmen ebenfalls die Geschwindigkeit der Glykolyse. Hexokinase wird von Glucose-6-Phosphat gehemmt, das sich anhäuft, wenn die PFK inaktiv ist. Schrittmacherreaktion (Commited Step): Phosphorylierung ovn F-6-P zu F-1,6-BP. PFK ist der wichtigste Kontrollpunkt der Glykolyse. Allgemein ist dasjenige Enzym, das die Schrittmacherreaktion einer Reaktionsfolge katalysiert, das wichtigste Kontrollelement dieses Stoffwechselweges. Pyruvat-Kinase wird von ATP und Alanin allosterisch gehemmt und von F-1,6-BP aktiviert, hat also ihre maximale Aktivität, wenn die Energieladung niedrig ist und sich Glykolyse-Zwischenprodukte anhäufen. Die hormongesteuerte Phosphorylierung hat die gleiche Funktion wie bei dem bifunktionellem Enzym das den F-2,6-BP Spiegel kontrolliert: die Leber am Glucoseverbrauch zu hindern, wenn Gehirn und Muskel die Glucose dringender benötigen. 16.2.4 Eine Familie von Transportproteinen (GLUT1 bis GLUT5) ermöglicht es der Glucose in tierische Zellen zu gelangen, oder sie zu verlassen. Die Glucosetransporter sind Mitglied der major faciliator-(MF-)Superfamilie. 16.2.5 Krebs und Glykolyse: Die hypoxischen Bedingungen innerhalb eines soliden Tumors führen zur Aktivierung des hypoxie-induzierenden Transkriptions-faktors (HIF-1), der eine Anpassung des Stoffwechsels induziert (Vermehrung der Glykolyseenzyme) und angiogene Faktoren aktiviert, die das Wachstum neuer Blutgefässe stimulieren. Der Citratzyklus (Kapitel 17, Seiten 509-531) 17 Citratzyklus: Oxidation von Glucosederivaten (C2 Einheit) zu Kohlendioxid. Dadurch werden energiereiche Elektronen gewonnen in Form von reduzierten Cofaktoren NADH und FADH2. Sie übertragen Energie auf O2. Bereitstellung von Bausteinen (Ausgangsverbindungen) für Biosynthese. 17.0.1 Citratzyklus im Überblick: Gewinnung von Elektronen hoher Energie in Form von reduzierte Cofaktoren NADH und FADH2 aus Brennstoffen. © 2004 Ana Sesartic 38 Zellatmung: Citratzyklus ist 1.Stufe der Zellatmung, in der e- hoher Energie aus dem als Brennstoff zugeführtem Kohlenstoff abgetrennt werden. Diese ereduzieren O2 und erzeugen einen Protonengradienten (erzeugt durch Fluss der e- durch eine Reihe von Membran-Proteinen), der zur ATP-Synthese verwendet wird. Die Reduktion des O2 und die ATP Synthese stellen die oxidative Phosphorylierung dar. Oxidation von Substrat ist entkoppelt von der Energiegewinnung durch oxidative Phosphorylierung (Atmungskette). Die meisten Brennstoffmoleküle werden als Acetyl-CoA in den Zyklus eingeschleust. Glykolyse und Citratzyklus sind durch die oxidative Decarboxylierung von Pyruvat unter der Bildung von Acetyl-CoA miteinander verbunden. Diese Reaktion sowie die Reaktionen des Citratzyklus laufen bei Eukaryonten innerhalb der Mitochondrien ab, im Gegensatz zur Glykolyse, die im Cytosol stattfindet. 17.1 Der Citratzyklus oxidiert Einheiten aus zwei Kohlenstoffatomen. Acetyl-CoA ist der Brennstoff für den Citratzyklus. Es entsteht beim Abbau von Glykogen, Fetten und vielen Aminosäuren. 17.1.1 Die Entstehung des Acetyl-CoA aus Pyruvat. Das in der Glykolyse produzierte Pyruvat wird in Acetyl-CoA umgewandelt, den Brennstoff des Citratzyklus. In der mitochondrialen Matrix wird das Pyruvat durch den Pyruvat-DehydrogenaseKomplex (multienzym Komplex aus 3 Enzymen) unter Bildung von AcetylCoA oxidativ decarboxyliert. (irreversible Reaktion) Umwandlung von Pyruvat in Acetyl-CoA: 1) Decarboxylierung 2) Oxidation 3) Übertragung der entstandenen Acetylgruppe auf CoA gekoppelte Reaktionen 17.1.2 Durch flexible Bindungen kann sich das Liponamid zwischen verschiedenen Zentren bewegen. Pyruvat-Dehydrogenase-Komplex: Die strukturelle Integration dreier verschiedener Enzyme ermöglicht die koordinierte Katalyse einer komplexen Reaktion. Die Nähe der einzelnen Enzyme erhöht die Gesamtgeschwindigkeit und vermindert Nebenreaktionen. 17.1.3 Die Citrat-Synthase bildet Citrat aus Oxalacetat und Acetyl-CoA. Kondensation von Oxalacetat (C4) und Acetyl-CoA (C2) unter Bildung von Citrat (C6), das zu Isozitrat (C6) isomerisiert wird. (Aldolkondensation: C4 + C2 = C6) Oxalacetat induziert eine grosse Strukturveränderung der CitratSynthase, und schafft so eine Bindungsstelle für das Acetyl-CoA. Dieses induced fit verhindert unerwünschte Nebenreaktionen. © 2004 Ana Sesartic 39 17.1.4 Citrat wird zu Isocitrat isomerisiert, durch eine Dehydratisierung und anschliessende Hydratisierung. 17.1.5 Isocitrat wird durch Oxidation und Decarboxylierung in α-Ketoglutarat überführt. Die Geschwindigkeit der α-Ketoglutarat-Bildung ist mit entscheidend für die Gesamtgeschwindigkeit des Zyklus. 17.1.6 Succinyl-CoA entsteht durch oxidative Decarboxylierung von α-Ketoglutarat, wobei das zweite CO2 Molekül entsteht. 17.1.7 Die Thioesterbindung im Succinyl-CoA wird durch anorganisches Phosphat unter Bildung von Succinat gespalten, wobei gleichzeitig eine Verbindung mit hohem Phosphorylgruppen-Übertragungspotenzial in Form eines GTP entsteht. 17.1.8 Regenerierung von Oxalacetat durch Oxidation von Succinat. Das Succinat wird zu Fumarat (C4) oxidiert, das dann zu Malat (C4) hydratisiert wird. Schliesslich wird das Malat oxidiert, um Oxalacetat (C4) zu regenerieren. Succinat-Dehydrogenase ist direkt mit der Atmungskette verbunden, dem Bindeglied zwischen Citratzyklus und ATP-Synthese. 17.1.9 Stöchiometrie des Citratzyklus Acetyl − CoA + 3 NAD + + FAD + GDP + Pi + 2 H 2O → 2CO2 + 3 NADH + FADH 2 + GTP + 2 H + + CoA 1) 2 C-Atome treten aus dem Acetyl-CoA in den Kreislauf ein und 2 C-Atome verlassen ihn in Form von CO2. Die aufeinanderfolgenden Decarboxylierungen werden durch die Isocitrat-Dehydrogenase und die α-Ketoglutarat-Dehydrogenase katalysiert. 2) In den vier Redox-Reaktionen des Zyklus werden 3 Elektronenpaare auf NAD+ und ein Paar auf FAD übertragen. Diese reduziertren e- Carrier werden in der Atmungskette unter Bildung von ca. 9 Molekülen ATP oxidiert. 3) Ein Molekül mit hohem Phosphorylgruppen-Übertragungspotenzial (GTP) entsteht bei der Spaltung der energiereichen Thioesterbindung im Succinyl-CoA. Daher entstehen insgesamt 10 Moleküle mit hohem Phosphorylgruppen-Übertragungspotenzial aus jeder C2 Einheit, die vollständig zu H2O und CO2 oxidiert wird. 4) 2 Moleküle Wasser werden verbraucht (1 bei Synthese des Citrats, 1 bei Hydratisierung des Fumarats). © 2004 Ana Sesartic 40 Molekularer Sauerstoff ist nicht direkt am Citratzyklus beteiligt, er verläuft aber nur unter aeroben Bedingungen, da NAD+ und FAD in den Mitochondrien nur durch e- Übertragung auf molekularen Sauerstoff regeneriert werden können. Im Gegensatz dazu kann die Glykolyse sowohl unter aeroben als auch unter anaeroben Bedingungen stattfinden. 17.2.1 Die Regulation des Pryuvat-Dehydrogenase-Komplexes erfolgt allo-sterisch und durch reversible Phosphorylierung. Die irreversible Entstehung von Acetyl-CoA aus Pyruvat ist ein wichtiger Kontrollpunkt für den Eintritt des aus der Glucose stammenden Pyruvats in den Citratzyklus. Die Aktivität des Pyruvat-Dehydrogenase-Komplexes wird durch reversible Phosphory-lierung gesteuert. Die e- Akzeptoren werden regeneriert, wenn NADH und FADH2 in der Atmungskette ihre Elektronen auf O2 übertragen, wobei ATP entsteht. Eine hohe Energieladung und grosse Mengen von Biosynthese-zwischen-produkten führen zur Abschaltung der Pyruvat-Dehydrogenase. © 2004 Ana Sesartic 41 17.2.2 Der Citratzyklus wird an verschiedenen Stellen kontrolliert. 17.2.3 Eine hohe Energieladung vermindert die Aktivitäten der IsocitratDehydrogenase und der α-Ketoglutarat-Dehydrogenase. Diese Mechanismen ergänzen einander und führen zu einer verlangsamten Acetyl-CoA-Bildung wenn die Energieladung der Zelle hoch ist und genügend Biosynthesevorstufen vorliegen. In vielen Bakterien ist auch die Synthese von Citrat aus Oxalacetat ein wichtiger Kontrollpunkt. 17.3 Der Citratzyklus liefert zahlreiche Biosynthesevorstufen, wie Nucleotidbasen, Protein und Hämgruppen. © 2004 Ana Sesartic 42 17.3.1 Wenn die Zwischenprodukte für Biosynthese abgezweigt werden, müssen sie wieder aufgefüllt werden durch anapletorische Reaktionen. Ein Beispiel einer solchen Reaktion wäre die Synthese des Oxalacetats durch Carboxylierung von Pyruvat. Pyruvat + CO2 + ATP + H 2O → Oxalacetat + ADP + Pi + 2 H + 17.3.2 Die Entgleisung des Pyruvatstoffwechsels ist die Ursache von Beriberi (Vitaminmangel [B1=Thiamin] Krankheit), sowie von Quecksilber- und Arsenitvergiftungen. 17.4 Der Glyoxylatzyklus ermöglicht es Pflanzen und Bakterien, mit Acetat zu wachsen (metabolitische Vielseitigkeit). Er umgeht die beiden Decarboxylierungsschritte des Citratzyklus, ausserdem werden 2 Moleküle Acetyl-CoA pro Durchgang in den Glyoxylatzyklus eingeschleust. In Pflanzen findet er in den Glykosomen statt. Problem: wenn man sich nur aus Substraten ernährt welche nur Acetyl-CoA ergeben (z.B. Fette): Kreislauf kann nicht aufgefüllt werden. Man kann keine Biosynthese machen, sondern nur Energie gewinnen. Lösung: (bei Pflanzen) Der Glykoxylatzyklus füllt die Zwischenprodukte wieder auf. Die oxidative Phosphorylierung (Kapitel 18, Seiten 535570) 18 Oxidative Phosphorylierung: Bei der Übertragung von e- von NADH und FADH2 über eine Reihe von e—Carriern auf O2 entsteht ATP, wenn Protonen durch einen Enzymkomplex in die Mitochondrienmatrix zurückfliessen. Die Oxidation von Brennstoffen und die Phosphorylierung von ADP sind durch eine Protonengradienten an der Inneren Mitochondrienmembran gekoppelt. 18.1.1 Die oxidative Phosphorylierung findet bei Eukaryonten in den Mitochondrien (inneren Mitochondrienmembran) statt. 18.1.1 Mitochondrien sind von einer Doppelmembran umschlossen. Oxidative Phosphorylierung findet in der inneren Mitochondrienmembran statt. Die meisten Reaktionen des Citratzyklus und der Fettsäureoxidation laufen in der Matrix ab. Äussere Membran enthält Mitochondrienporine, ein VDAC (voltage dependent anion channel). © 2004 Ana Sesartic 43 Bei Prokaryonten sind die Protonenpumpen und der Komplex zur ATPSynthese in der Cytoplasmamembran lokalisiert. 18.1.2 Mitochondrien sind das Resultat eines endosymbiotischen Ereignisses. Sie stammen von freilebenden Bakterien ab, die eine Symbiose mit einer anderen Zelle eingingen. 18.2 Die oxidative Phosphorylierung hängt vom Elektronentransfer ab. 18.2.1 Bei der oxidativen Phosphorylierung wird das Elektronenübertragungspotenzial des NADH oder des FADH2 in das Phosphorylgruppenübertragungspotenzial des ATP umgewandelt. Ein starkes Reduktionsmittel (NADH) gibt leicht e- ab und besitzt daher ein negatives Reduktionspotenzial während ein starkes Oxidationsmittel (O2) e- aufnimmt und ein positives Reduktions-potenzial besitzt. 18.2.2 Eine Potenzialdifferenz von 1,14V zwischen NADH und O2 treibt die Elektronentransportkette an und begünstigt die Bildung eines Protonengradienten. 18.2.3 Elektronen können zwischen Gruppen übertragen werden, die nicht in Kontakt stehen. Mithilfe einer aus Proteinen bestehenden Umgebung nimmt Geschwindigkeit des Elektronentransfers zu. Die e- -Transfer Geschwindigkeit nimmt bei höherer Antriebskraft der Reaktion zu, erreicht Maximus und vermindert sich dann. Elektronen-Carrier in den Proteinen der Elektronentransportkette sind Flavine, Eisen-Schwefel-Cluster, Chinone, Hämgruppen und Kupferionen. Bei Chinonen sind Elektronen-Transfer-Reaktionen mit der Protonenbindung und –freisetzung gekoppelt. 18.3 Die Atmungskette besteht aus vier Komplexen: drei Protonenpumpen und einer direkten Verbindung zum Citratzyklus. Der Elektronenfluss innerhalb der Transmembrankomplexe (NADH-Q-Oxioreduktase, Succinat-Q-Reduktase, Q-Cytochrom-c-Oxioreduktase, Cytochrom-c-Oxidase) bewirkt einen Protonentransport durch die innere Mitochondrienmembran. 18.3.1 Am Anfang der Atmungskette werden e- mit hohem Potenzial vom NADH auf die NADH-Q-Oxioreduktase übertragen. Diese Oxioreduktase enthält auch Fe-SZentren. Die e- tauchen dann wieder im QH2 auf, der reduzierten Form des Ubiquinons (Q). Durch den Fluss von 2e- durch die NADH-Q-Oxioreduktase vom NADH zum QH2 werden vier Protonen aus der Matrix des Mitochondriums gepumpt. Die Reduktion von Q zu QH2 führt zur Aufnahme von 2 Protonen aus der Matrix. (Siehe auch Abb.18.14, S.546) 18.3.2 Über Ubichinol treten e- vom FADH2 der Flavoproteine in die Atmungskette ein. Die Succinat-Dehydrogenase des Citratzyklus ist ein Teil des Succinat-QReduktase-Komplexes, der e- vom FADH2 auf Q unter Bildung von QH2 überträgt. Enzyme die e- vom FADH2 auf Q übertragen sind keine Protonenpumpen; deshalb wird bei der Oxidation von FADH2 weniger ATP gebildet. © 2004 Ana Sesartic 44 18.3.3 Die e- fliessen vom Ubichinol über die Q-Cytochrom-c-Oxioreduktase zum Cytochrom c. Cytochrom c ist ein beweglicher e- Carrier und überträgt die eauf die Cytochrom-c-Oxidase. Die Aufgabe der Q-Cytochrom-c-Oxidoreduktase besteht darin, die Elektronenübertragung von QH2 auf das oxidierte Cytochrom-c, ein wasserlösliches Protein, zu katalysieren und gleichzeitig Protonen aus der Mitochondrienmatrix zu pumpen. Ausserdem beinhaltet sie ein Rieske-Zentrum: 2Fe2S-Zentrum; eines der beiden Eisenionen ist mit zwei Histidinresten koordiniert. Diese Koordination stabilisiert das Zentrum in seiner reduzierten Form und erhöht sein Reduktionspotenzial. 18.3.4 Transmembrantransport von Protonen – der Q-Zyklus: Von den 2e- eines gebundenen QH2 wird eines auf Cytochrom c und das andere auf ein gebundenes Q unter Bildung des Semichinonradikalions Qübertragen. Das neu entstandene Q dissoziiert und wird durch ein zweites QH2 ersetzt, das ebenfalls seine e- abgibt: ein e- an ein zweites Cytochrom c, das andere an Q- zur Reduktion zu QH2. Dieser zweite e- Transfer führt zur Aufnahme von zwei Protonen aus der Matrix. Die prosthetischen Gruppen sind in ihrer oxidierten Form blau, in ihrer reduzierten Form rot dargestellt. 18.3.5 Die Cytochrom-c-Oxidase enthält die Cytochrome a und a3 sowie zwei Kupferionen und katalysiert die Reduktion von molekularem Sauerstoff zu Wasser. © 2004 Ana Sesartic 45 Ein Hämeisen und ein Kupferion in dieser Oxidase übertragen die e- dann auf O2, den letzten Akzeptor, wobei Wasser entsteht. Zusammen bilden Häm a3 und CuB das aktive Zentrum, in dem O2 zu H2O reduziert wird. Cytochrom-c-Oxidase pumpt im Verlauf eines jeden Reaktionszyklus weitere 4 Protonen aus der Matrix zur cytoplasmatischen Seite der Membran, sodass insgesamt 8 Protonen aus der Matrix entfernt werden. Der Katalysator darf keine tw. Reduzierten Zwischenprodukte freisetzen, denn die Übertragung eines e- auf O2 führt zu einem Superoxidanion, die Übertragung von 2e- zu Peroxid; beides für die Zelle toxische Verbindungen. 18.3.6 Das Superoxidradikal und andere toxische Derivate des O2 werden durch Schutzenzyme abgefangen. Superoxid-Dimutase fängt Superoxidradikale ab, indem es die Umwandlung zweier dieser Radikale in ein Wasserstoff-peroxid und molekularen Sauerstoff katalysiert. Das von der SuperoxidDimutase und bei anderen Prozessen gebildete Wasserstoffperoxid wird von der Katalase vernichtet. 18.3.7 Die Konformation des Cytochrom c blieb im wesentlichen mehr als 1 Mrd. Jahre konstant. 18.4 Ein Protonengradient treibt die ATP-Synthese an. Chemiosmotische Hypothese: Beim e- Transfer durch die Atmungskette werden Protonen aus der Matrix auf die Cytosolseite der inneren Mitochondrienmembran gepumpt. Der pH Gradient und das Membranpotenzial erzeugen eine protonenmotorische Kraft, die zum Antrieb der ATP-Synthese dient. 18.4.1 Die ATP-Synthase besteht aus einer protonenleitenden und einer katalytischen Einheit. Die γ-Untereinheit unterbricht die Symmetrie des α3 β3 – Hexamers: die 3 β-Untereinheiten unterscheiden sich aufgrund ihrer Wechselwirkung mit einer jeweils anderen Oberfläche von γ. F0 enthält den Protonenkanal des Komplexes. Rotor: beweglich, besteht aus c-Ring und γε-Stiel Stator: fester Rest des Moleküls Die Rotation der γ-Untereinheit induziert Strukturveränderungen in der β-Untereinheit, die zur Synthese und Freisetzung des ATP durch das Enzym führen. Der Protonenfluss durch die ATPSynthase liefert die Kraft für die Rotation. Protonenmotorische Kraft: pH Gradient (Matrixseite basisch) und Membranpotenzial (Matrixseite negativ). Protonenfluss zurück zur Matrixseite durch die ATP-Synthase treibt die ATP-Synthese an. © 2004 Ana Sesartic 46 18.4.2 Der Protonenfluss durch die ATP-Synthase führt zur Freisetzung von fest gebundenem ATP: der Mechanismus des Bindungswechsels. Die Rotation der γ-Untereinheit kann die 3 β-Untereinheiten umwandeln. Die Untereinheit in der T-Form (tight; fest) enthält neu synthetisiertes ATP, das aber nicht abgegeben werden kann. Die T-Form wird in eine O-Form (offen) umgewandelt, die ATP freisetzen kann. Sie bindet dann ADP und Pi und ein neuer Zyklus beginnt. Enzym gebundenes ATP bildet sich schnell auch in Abwesenheit einer protonenmotorischen Kraft. 18.4.3 Der kleinste molekulare Motor der Welt: die Rotationskatalyse. Die γUntereinheit rotiert in 120° Schritten, wobei jeder Schritt der Hydrolyse eines ATP-Moleküls entspricht, die die Rotation antreibt. 18.4.4 Der Protonenfluss rund um den c-Ring treibt die ATP-Synthese an. Die Protonenbewegung durch die Membran treibt die Rotation des c-Ringes an. Ein Proton tritt vom Intermembranraum in den cytosolischen Halbkanal ein und neutralisiert die Ladung eines Aspartatrestes in einer c-Untereinheit. Damit kann sich der c-Ring im Uhrzeigersinn um eine c-Einheit weiterdrehen und bewegt dabei einen Asparaginsäurerest aus der Membran in den Matrixhalbkanal. Dieses Proton kann sich in die Matrix bewegen und versetzt das System in den ursprünglichen Zustand. © 2004 Ana Sesartic 47 18.4.5 ATP-Synthase und G-Proteine besitzen mehrere gemeinsame Eigenschaften. 18.5 Viele Shuttle-Systeme ermöglichen den Transport durch mitochondriale Membranen. 18.5.1 Die e- des cytosolischen NADH gelangen durch Shuttle-Systeme in die Mitochondrien. Glycerin-3-Phosphat-Shuttle: e- des NADH gelangen in die mitochondriale e- Transportkette, wenn sie zur Reduktion von Dihydroxyacetonphosphat zu Glycerin-3-Phosphat verwendet werden. Das G-3-P wird durch einen e- Transfer zur prosthetischen FAD-Gruppe in der membrangebundenen Glycerin-3-Phosphat-Dehydrogenase reoxidiert. Die folgende e- Übertragung auf FQ, wobei QH2 entsteht, ermöglicht diesen eden Eintritt in die Atmungskette. Im Herz und in der Leber gelangen die e- des cytosolischen NADH über den Malat-Aspartat-Shuttle zu dem 2 Membran-Carrier und 4 Enzyme gehören, in die Mitochondrien. Dieser Shuttle ist im Gegensatz zum G-3-P Shuttle leicht reversibel. NADH kann nur dann über den Malat-Aspartat-Shuttle in die Mitochondrien gelangen, wenn das NADH/NAD+ Verhältnis im Cytosol höher ist als in der mitochondrialen Matrix. 18.5.2 Der Eintritt von ADP in die Mitochondrien ist mit dem Austritt von ATP durch eine ATP-ADP-Translokase gekoppelt, welche den Molekülen ermöglicht, die Permeabilitätsschranke zu überwinden. ADP gelangt nur in die mitochondriale Matrix, wenn ATP austritt, und umgekehrt. Bei einem positiven © 2004 Ana Sesartic 48 Membranpotenzial (Betrieb der Atmungskette) erfolgt die Umstülpung des Bindungszentrums von der Matrix- zur Cytosolseite schneller für ATP als für ADP, da ATP eine negative Ladung mehr besitzt. Das Membranpotenzial und daher auch die protonenmotorische Kraft wird beim Austausch von ATP gegen ADP vermindert, woraus sich ein Nettotransfer einer negativen Ladung aus der Matrix ergibt. 18.5.3 Die mitochondrialen Transporter für Metaboliten: Phosphat Carrier Dicarboxylat Carrier Tricarboxylat Carrier Pyruvat Carrier 18.6 Die Regulation der oxidativen Phosphorylierung wird hauptsächlich durch den ATP-Bedarf bestimmt. 18.6.1 Die vollständige Oxidation der Glucose zu CO2 ergibt etwa 30 ATP. © 2004 Ana Sesartic 49 18.6.2 Die Geschwindigkeit der oxidativen Phosphorylierung wird durch den ATP-Bedarf bestimmt. Normalerweise fliessen keine e- durch die e- Transportkette zum O2, wenn nicht gleichzeitig ADP zu ATP phosphoryliert wird. Atmungskontrolle: Regulation der Geschwindigkeit der oxidativen Phosphorylierung durch den ADP-Spiegel. e- fliessen von Brennstoffmolekülen zum O2 nur wenn ATP Bedarf vorliegt. NADH und FADH2 werden nur oxidiert, wenn gleichzeitig ADP zu ATP phosphoryliert wird. 18.6.3 Die oxidative Phosphorylierung kann an vielen Stellen gehemmt werden. Rotenon und Amytal blockieren die e- Übertragung innerhalb der NADHQ-Oxidoreduktase. Antimycin A blockiert den eFluss am Cytochrom bH in der Q-Cytochrom-cOxidase. Cyanid, Azid und Kohlenmonoxid blockieren e- Transfer in der Cytochrom-c-Oxidase. Die ATP-Synthase kann ebenfalls gehemmt werden. 18.6.4 Ein Kurzschluss im Protonengradient erzeugt Wärme. Das Entkopplungsprotein-1 (UCP-1) erzeugt Wärme, indem es den Fluss der Protonen in die Mitochondrien ohne ATP-Synthese vermittelt. Diese Methode der Wärmeerzeugung nützen winterschlafende Tiere, neugeborene Tiere (auch menschliche Babys) sowie kälteangepasste Tiere. 18.6.5 Mitochondrienkrankheiten: Leber'sche Optikusneuropathie (Form der Erblindung, die im mittleren Lebensalter auftritt). Die Häufung von Mutationen im Mitochondriengenom über Jahrzehnte hinweg können zum Altern, zu degenerativen Erkrankungen und zu Krebs beitragen. Da die mütterlich ererbten Mitochondrien in grosser Zahl vorhanden sind, gibt es viele Mutationsmöglichkeiten. Es gibt grosse Variationen in Art und Schwere der Krankheitssymptome, sowie ihrem Einsetzen. Defekte in der Zellatmung sind doppelt gefährlich: Energieerzeugung vermindert sich und die Wahrscheinlichkeit, dass reaktive Sauerstoffmoleküle anfallen steigt. 18.6.6 Mitochondrien spielen bei der Apoptose (programmierter Zelltod) eine Schlüsselrolle. In geschädigten Mitochondrien bildet sich die mitochondriale Permeabilitätstransitionspore. Cytochrom c aktiviert im Cytosol eine Kaskade von proteolytischen Enzymen (Caspasen), mit je besonderen Zielen "death by a thousand tiny cuts". © 2004 Ana Sesartic 50 18.6.7 Energieübertragung durch Protonengradienten: ein zentrales Prinzip der Bioenergetik. Protonengradienten sind eine zentrale, unwandelbare Form freier Enthalpie in biologischen Systemen. Aktiver Transport Elektronenpotenzial NADPH-Synthese ATP ~P Wärmeerzeugung Flagellenrotation © 2004 Ana Sesartic 51 ANHANG Allgemeines Schema für den Abbau von organischen Molekülen. © 2004 Ana Sesartic 52 Fruktose-1,6-diphosphat-Weg des Abbaus von Glukose (Glykolyse) © 2004 Ana Sesartic 53