Experimente zur katalytischen partiellen Oxidation niederer Alkane
Werbung

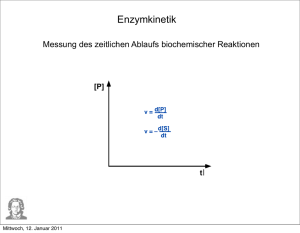
ENZYMKINETIK
1
Versuch K3
Enzymkinetik
1
Theoretische Grundlagen:
Enzyme gehören zur Klasse der Proteine (Eiweißstoffe). Die Monomereinheiten, aus denen
sie aufgebaut sind, sind Aminosäuren. Enzyme sind die Katalysatoren lebenswichtiger
Prozesse in der Zelle. Ihre zentrale Bedeutung lässt sich in dem Satz - Ohne Enzyme kein
Leben- zusammenfassen. Je nach dem Typus der biochemischen Reaktion, die katalysiert
wird, lassen sich die Enzyme unterteilen in:
-
Oxidoreduktasen: Redoxprozesse (z.B.: NAD+/NADH+H+)
-
Transferasen: Gruppenübertragungsreaktionen (z.B.: Methylgruppen)
-
Hydrolasen: Hydrolysen (z.B: Esterverseifung, Peptidspaltung)
-
Lyasen: nicht hydrolytische Bindungsspaltung(z.B.: C-C, =C=O)
-
Ligasen: Kopplung zweier Substrate (z.B.: Eiweißsynthese)
-
Isomerasen: Isomerisierungsreaktionen (z.B.: Keto-Enol-Umwandlung)
Enzymreaktionen folgen den Gesetzen der Reaktionskinetik, wenn man das Enzym als
stöchiometrische Größe mit in den Reaktionsablauf einbezieht. Um der Katalysatorfunktion
gerecht zu werden, muss man einen Kreisprozess formulieren:
E
S
+
EP
ES
E
+
P
Das Enzym (E) bildet mit dem Substrat (S) einen kurzlebigen Enzym-Substrat Komplex (ES),
der zu einem Enzymproduktkomplex (EP) umgesetzt wird. Diese bildet nach seinem Zerfall
das Produkt (P) und regeneriert dabei das Enzym. Michaelis und Menten formulierten 1913
einen Mechanismus, mit dem sich die Kinetik enzymatisch katalysierter Reaktionen einfacher
beschreiben lässt (Vernachlässigung der Bildung des Enzym-Produkt-Komplexes:
k2
k1
E
+
S
E
ES
k -1
k -2
+
P
2
ENZYMKINETIK
Dies ist ein Beispiel einer Folgereaktion. Unter der Voraussetzung, dass k2 << k1 ist, gilt die
Bodensteinsche Näherung für die Quasistationarität:
d [ES] / dt = k1 [E][S] - k-1[ES] - k2[ES] + k-2[E][P] = 0
Hier stellt [E] die Konzentration an freiem Enzym dar .
Durch die Annahme, dass zu Anfang [P]= 0 und weitere Umformungen, folgt schließlich die
Michaelis-Menten-Gleichung:
v = - d[S] / dt = k2[ES] = k2[Eo] [S] / {KM + [S]}
(I)
wobei [Eo ] = [E] + [ES] die gesamte Enzymkonzentration (Summe aus freiem und vom
Substrat gebundenem Enzym) darstellt.
KM ist die Michaelis-Menten-Konstante:
KM = {( [Eo] - [ES] ) [S] } / [ES]
Für [S] >> KM (Substratüberschuss) läuft die Reaktion mit maximaler Geschwindigkeit ab.
Die Michaelis-Menten-Gleichung wird zu:
vmax = k2 [E0]
Setzt man dies in Gleichung I ein so ergibt dies.
v = - d[S] / dt = d[P] / dt = vmax [S] / {KM + [S]}
(II)
Eine wichtige Beziehung erhält man für den Fall, dass v = vmax / 2 :
KM = [S]
(Welche Einheit hat also die Michaelis-Menten-Konstante?)
Hieraus ergibt sich praktisch die Möglichkeit zur Bestimmung von KM . In der Praxis geht
man allerdings so vor, dass man Gleichung II entweder in die Lineweaver-Burk- oder die
Eadie-Hofstee-Beziehung verwandelt und KM dann graphisch ermittelt. (siehe Lehrbuch !!!,
z.B. Moore-Hummel, Physikalische Chemie)
Zur Untersuchung enzymkatalysierter Methoden kommen je nach Reaktionstyp verschiedene
Methoden (pH-Wert, Leitfähigkeitsmessung, Sauerstoffbestimmung, Photometrie) zur
Anwendung. In diesem Versuch soll die Änderung der Extinktion (Absorption) des
ENZYMKINETIK
3
enzymkatalysierten Alkoholabbaus mittels der Photometrie untersucht werden. Ein
Absorptions photometer ist prinzipiell folgendermaßen aufgebaut:
Lichtquelle
Blende
Messküvette
Monochromator
(Gitter oder Prisma)
Photomultiplier
Verstärker und
Anzeige
Das Lambert-Beersche-Gesetz, in seiner für die Photometrie relevanten Form, sagt aus, dass
die Extinktion (dekadischer Logarithmus des Intensitätsverhältnis von eintretendem zu
austretendem Licht: I0/I ) vom molaren Extinktionskoeffizienten ε, der Dicke der Küvette d
(beide konstant und per Eichmessung bestimmbar) und der Konzentration c (z. B. Produkt das
gebildet wird und Licht einer bestimmten Wellenlänge absorbiert) abhängig ist.
E = log I0/I = ε c d
(III)
Bei konstanter Wellenlänge (ε=konst) und definierter Küvettenlänge d ist damit die
Extinktion proportional zur Konzentration des absorbierenden Stoffes. Dies ist das
Messprinzip der Photometrie, mit der Konzentrationen gemessen werden können. Gleichung
II lässt sich umformen in:
v = d[P] / dt = dc / dt = vmax [S] / ( KM + [S]) }
(IV)
Gleichung III nach c aufgelöst und in IV eingesetzt ergibt (ε d = konstant):
dE / dt = ε d { vmax [S] / ( KM + [S]) } = konst⋅v (mit konst= ε·⋅d)
4
ENZYMKINETIK
2
2.1
Experimenteller Teil:
Einfluss der Substratkonzentration auf die Enzymkinetik
Prinzip:
Die Peroxidase (POD) katalysiert unter Verbrauch von Wasserstoffperoxid die Oxidation von
2,2´´-Azino-di[3-ethylbenzthiazolinsulfonate (6)] (ABTS) zu einem grünen Farbstoff.
PO D
ABTS
+
H2O 2
grüner Farbstoff
+
H2O
Die Reaktion lässt sich aufgrund des charakteristischen Absorptionsmaximums des
Farbstoffes bei 405 nm photometrisch verfolgen. Bei überschüssiger Enzym (POD) soll der
Einfluss der Substratkonzentration (ABTS) auf die Reaktionsgeschwindigkeit untersucht
werden.
Material:
-
2,2´´-Azino-di[3-ethylbenzthiazolinsulfonate (6)] (ABTS)
-
Peroxidase-Lösung (0.2 mg Enzym . ml-1)
-
Wasserstoffperoxid -Lösung, 30%ig
-
PBS-Pufferlösung, pH 7.4-7.5 (0.8359 g Citronensäuremonohydrat, 1.068 Dinatriumhydrogenphosphat, 100 ml destilliertes Wasser)
Versuchsdurchführung:
Zu beachten:
- Den Deckel des Spektrometers bitte immer vorsichtig schließen, nie fallen lassen!
- Für Peroxidase, ABTS und Wasserstoffperoxid bitte separate Mikroliterspritzen nehmen
(entsprechend der Beschriftung)!!!
Vor Beginn des Versuchs soll das Spektrometer eingeschalten und auf 405 nm eingestellt
werden. Nach einer Viertelstunde hat der Temperaturausgleich stattgefunden. Nun soll durch
Drücken der Taste Mode auf Absorption gestellt werden. In einer Versuchsreihe soll eine
ENZYMKINETIK
5
Abhängigkeit der Reaktionsgeschwindigkeit von der Substratkonzentration für verschiedene
Substratkonzentrationen ermittelt werden. Bei jeder Messung muss der Deckel geschlossen
sein.
Zur Ausführung des Versuchs wird zunächst eine Lösung von 5 mg ABTS (SartoriusFeinwaage im anderen Praktikumsflügel beutzen!) in 1 ml Pufferlösung hergestellt. Diese
Lösung wird auch noch für den zweiten Versuchsteil benötigt. Das ABTS-Pulver nach dem
Ansetzen der Lösung unverzüglich verschließen und wieder in den Kühlschrank stellen. Nun
werden 10 µl Peroxidaselösung mit 90 µl Pufferlösung versetzt (Verdünnung 1:10). Auch
diese verdünnte Lösung wird im zweiten Versuchsteil nochmals gebraucht.
Es werden fünf Ansätze mit je 2.0 ml Pufferlösung, 2.5 µl verdünnter Peroxidaselösung und
2.5 µl Wasserstoffperoxid in 5 Küvetten gegeben . Nun wird die erste Küvette in das
Spektrometer gestellt und der Rührmotor eingeschaltet (Rührfisch in Küvette geben - dreht er
sich?). Jetzt nimmt man den Nullabgleich - bei geschlossenem Spektrometer - vor (Drücken
der correction-Taste). Nach Zugabe von 2 µl der ABTS-Lösung wird sofort die Stoppuhr
gestartet, der Deckel vorsichtig geschlossen und sofort alle 10 Sekunden die Extinktion
4 Minuten lang notiert. Die gleiche Prozedur (inklusive Nullabgleich!) wiederholt man mit
4 µl, 6 µl, 10 µl, 25 µl ABTS.
Auswertung:
Die Extinktionswerte sind für jede Substratkonzentration (Mw(ABTS) = 548.7 g/mol) in einem
Diagramm gegen die Zeit [s] aufzutragen (Michaelis-Menten-Graphik). Die Steigungen
(Änderung der Extinktion mit der Zeit) entsprechen den Reaktionsgeschwindigkeiten
(Gleichung III), die man in einem anderen Diagramm in Abhängigkeit von der
Substratkonzentration [mol/l] aufträgt. Mit Hilfe dieses Diagramms ermittelt man vmax /2 und
die Michaelis-Menten-Konstante. Außerdem erstelle man das Lineweaver-Burk-Diagramm,
um die gleichen Werte zu ermitteln. Hierfür wird die Michaelis-Menten-Gleichung
umgeformt in:
1/v = {KM / vmax [S]} + 1 / vmax
Nun trage man 1/v gegen 1 / [S] auf und ermittle vmax aus dem Ordinatenabschnitt und KM aus
dem Abszissenabschnitt.
6
ENZYMKINETIK
Zu beachten:
Die Ergebnisse sind nicht mit Literaturwerten vergleichbar, da es sich bei der eingesetzten
Peroxidase um ein Antikörperkonjugat handelt, so dass die Aktivität des Enzyms verändert ist.
2.2
Einfluss der Enzymkonzentration auf die Reaktionsgeschwindigkeit
Prinzip: (gleiche Reaktionsgleichung wie bei Versuch 1)
Die Reaktionsgeschwindigkeit derselben Reaktion wie oben soll - jetzt bei Substratüberschuss - in Abhängigkeit von der Enzymkonzentration (POD) untersucht werden.
Material:
wie bei Versuch 1
Ausführung:
Zunächst entnimmt man mit der entsprechenden Mikroliterspritze 10 µl aus der 1:10
verdünnten (!) Peroxidase-Lösung und mischt mit 1 ml PBS-Pufferlösung (pH 7.4) in einem
Reagenzglas. Man erhält also eine weitere Verdünnung um den Faktor 100. Nach dem
Ansetzen der Verdünnung beide Peroxidasespritzen gründlich mit dest. H2O waschen, um
eine Konzentrationsverfälschung zu vermeiden. In 5 Küvetten lege man jeweils 2.0 ml
Pufferlösung, 2.5 µl Wasserstoffperoxid und verschiedene Volumina der neuen PeroxidaseVerdünnung (0, 10, 20, 30, 40 und 100 µl) vor. Nun wird der Rührmotor gestartet und nach
Nullabgleich und der Zugabe von 25 µl der ABTS-Lösung (aus Versuch 1) die Extinktion
4 Minuten lang alle 10 s aufgenommen.
Zu beachten:
Nach Beendigung der Versuche bitte alle verwendeten Glasgeräte und Mikroliterspritzen mit
destilliertem Wasser spülen (Küvetten zuerst mit Küvettenspüli und anschließend destilliertem
Wasser) und zum Trocknen in die entspreechenden Vorrichtungen stellen (Küvettenständer,
Pipettengestell). Wasserstoffperoxid-Lösung in den Kühlschrank zurückstellen!
ENZYMKINETIK
7
Auswertung:
In einem Diagramm sind für jede Enzymkonzentration (Mw(POD) = 40.000) die gemessenen
Extinktionswerte gegen die Zeit aufzutragen. Die Steigungen (dE/dt) sind aus dem Diagramm
abzulesen. Die entsprechenden Enzymkonzentrationen sind in einem zweiten Diagramm
gegen die ermittelten Steigungen aufzutragen. Man erhält eine Eichkurve, die eine
Voraussage der zu erwartenden Extinktionsänderung bei einer bestimmten vorgelegten
Enzymkonzentration ermöglicht.
Die Steigung dieser Eichgerade ist graphisch zu ermitteln. Warum ergibt sich laut Theorie ein
linearer Verlauf der Eichkurve (s. Theorieteil!!) ? Welche Fehlerquellen gibt es für einen
nicht linearen Verlauf der Eichkurve (Stichwort: Gültigkeitsbereich des Lambert-Beerschen
Gesetz) ?
Stichworte:
Grundlagen der Biochemie:
Enzyme,
Aminosäuren,
Proteine,
(Primär-,
Sekundär-,
Tertiärstruktur,
Funktion),
Transskription, Translation
Grundlagen der Kinetik:
s. a. Grundlagen Versuch K1 und K2: (Definition der Reaktionsgeschwindigkeit, Reaktionsordnung, Halbwertszeit, Arrheniusgleichung, …), für diesen Versuch: Folgereaktionen,
Fließgleichgewicht, Quasistationarität, Beispiele enzymatischer Reaktionen, MichaelisMenten-Kinetik, graphische Darstellung und Herleitung der Michaelis-Menten-Gleichung,
Lineweaver-Burk, Eadie-Hofstee, Beeinflussung der Geschwindigkeit enzymatischer
Reaktionen (Substratkonzentration, Enzymkonzentration, Hemmung: irrreversibel, reversibel:
kompetitiv, nichtkompetitiv, antikompetitiv)
Spektroskopische Grundlagen:
Aufbau und Prinzip eines Absorptionsspektrometers, Photometrie, Lambert-Beersches
Gesetz, wie kommt Absorption zustande?
8
ENZYMKINETIK
Empfohlene Literatur :
Theoretische Grundlagen
¾ Michaelis-Menten (Kinetik); Physikalische Chemie, Moore-Hummel, 4. Auflage,
¾ Biochemische Grundlagen: Kurzes Lehrbuch der Biochemie, Karlson, 14. Auflage;
Organische Chemie, Beyer-Walter
¾ Atkins: Kinetik allgemein, Spektroskopie: Physikalische Chemie
¾ L. Stryer: Biochemie
Ausführung und Auswertung
¾ Hassinger-Wiebusch: Experimentelle Enzymologie
¾ Moore-Hummel: Physikalische Chemie, 4. Auflage




![E + S [ES] E + P Enzyme](http://s1.studylibde.com/store/data/011452180_1-cbdca6d5ea6d6e898aef4e3165d8acb4-300x300.png)