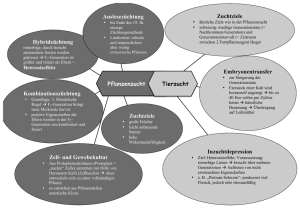
Kap 3 Gene und ihre Frequenzen Evolution “Moderne Synthese
Werbung

Evolution Kap 3 Gene und ihre Frequenzen “Moderne Synthese” “Moderne Synthese” Mechanismus: Selektion Einheit: Gen Makroevolution Mikroevolution (1890 - 1962) Arten, Gattungen,… Populationen, Individuen,… Lange Zeiträume Kurze Zeiträume (1892 - 1964) (1889 - 1988) R.A. Fisher (1930) The genetical theory of natural selection J.B.S Haldane (1932) The causes of evolution S. Wright (1931) Evolution in Mendelian populations Variation Morphologische Marker Gregor Mendel (1822 - 1884) Varianz: Mittelwert: V(x) = Vgenetic + Venvironmental Vgenetic = Vadd + Vdominance + Vepistasis R.A. Fisher Mendel’sche Genetik Morphologische Marker Locus Chromosom (Vater) Chromosom (Mutter) wild type Karyotyp eines Menschen yellow ebony Curly Molekulare Marker Gel-Elektrophorese • Allozyme (Iso-enzyme) • RFLP (restriction fragment length polymorphism) • RAPD (random amplified polymorphic DNA) • AFLP (amplified fragment length polymorphism) • Microsatellites • SNP (single nucleotide polymorphism) 90% der genetischen Variation, 1SNP/100-300 bp Variation in menschlichen Populationen Allozyme: Beispiel H = % der heterozygoten Loci unter allen Individuen Locus Enzym Acph Pgm-1 Pgm-2 Adk Pept-A Pept-C Pept-D Adn 6Pgdh Aph Amy Gpt Acid phosphotase Phosphoglucomutase-1 Phosphoglucomutase-2 Adenylate kinase Peptidase-A Peptidase-C Peptidase-D Adenosine deaminase 6-Phosphogluconate dehydrogenase Alkaline phosphatase (placental) Amylase (pancreatic) Glutamate-pyruvate transaminase (gemessen: 71 Loci bei Europäern) H 0.52 0,36 0.38 0,09 0,37 0,02 0.02 0.11 0.05 0.53 0.09 0.50 after Harris & Hopkinson (1972) J. Human Genet 36:9 (Ayala 1982, Tab.2.9) ADH: Alkoholdehydrogenase Mensch: ca. 90% aller asiatischen PazifikBewohner: Isoform ADH2*2 Vorteile: co-dominant, günstig, keine genomische Information Nachteile: Proteine stehen unter Selektion, wenig Variabilität Genetische Variation Vaterschaft Mutter Väter Mikrosatelliten Das Null-Modell Genetische Variation Es sei gegeben: Frequenzen pA, pa (pA + pa = 1) Diversität im Gen für Zystische Fibrose (Loss-of-function mutations) Eltern Weibchen Eier Gameten Männchen Spermien Die Gameten 1 Locus, 2 Allele: A,a Frequenzen pA, pa (pA + pa = 1) pA = 0.6 pa = 0.4 Genotypfrequenzen Allelfrequenzen Hardy-Weinberg Godfrey Harold Hardy Wilhelm Weinberg Genotypfrequenzen im Gleichgewicht: P = Genotyp AA = pA2 H = Genotyp Aa = 2 pA* pa Q = Genotyp aa = pa2 pA2 + 2 pA* pa + pa2= 1 Hardy-Weinberg HWE Hardy-Weinberg Beispiel: Schönbär (Panaxia dominnula) Bedingungen: • Sexuelle Fortpflanzung • Zufallspaarungen • Unendliche Populationsgrösse • Allele segregieren nach Mendel • Keine evolutiven Kräfte (Selektion, Drift, Genfluss, Mutation) HWE = Nullmodel Alle Abweichungen liegen an der Verletzung der Bedingungen Beobachtet χ2 Erwartet AA 1469 AB 138 2pApBN= BB 5 pB2N= pA2N= 1467.1 0.002 141.5 0.087 3.4 0.753 N = 1612 pA = (1469 + 69)/1612 = 0.954 pB = (69 + 5)/1612 χ2 = Σ(B-E)2/E χ2 = 0.842 N.S. χcrit2(1, 0.05)= 3.84 = 0.046 HWE Beispiel: Coelopa frigida Abweichungen vom HWE 3 Genotypen (ADH) in England Erwartet Beobachtet Frequenz Individuen BB 12 0.157 17.43 BD 64 0.478 53.06 DD 35 0.365 40.51 N = 111 1.000 111.00 2N = 222 Allele pB = (24 + 64)/222= 0.396 pD = (70 + 64)/222= 0.604 χ2 = 4.66; P = 0.03 • Kleine Populationsgrössen (->Drift) • Nichtzufällige Paarung • Selektion, Mutation, Migration Assortative Paarung Disassortative Paarungen Assortative Paarung Selbstinkompatabilität (SI) M F Pawlowsky (2003) Proc R Soc Lond B 270: 709-712 Inzucht Grosseltern A1A2 Inzucht Koeffizient F A3A4 F= Nicht-verwandt Eltern Nachkommen A1A3 H0 - H H0 ,mit H0 = 2pq H = beobachtete Heterozygosität A1A3 A1A4 Bereich: -1 ≤ F ≤ 1 A1A1 Inzucht “identical by descent” A1A1 Keine Inzucht ! “identical by state” Sewall Wright Inzucht, “selfing” P Eltern p2 A1A1 H Q 2pq q2 A1A2 Inzucht, Stammbaum AB AB A2A2 1/ 2 1/4 A1A1 1/2 A- 1/4 A1A2 Nachkommen A1A1 P’ A2A2 Q’ Selfing Eltern p2 A1A1 A- 1/ 2 1/ 2 1/ 2 B- B- 1/ 2 1/ 2 A2A2 H’ P 1/ 2 BB Inzucht H Q 2pq q2 A1A2 AA A2A2 Ht = H0 * (1-F)t 1/4 A1A1 Nachkommen 1/2 A1A2 A1A1 P’ 1/4 A2A2 A2A2 H’ F = (2pq - pq)/2pq =0.5 Q’ Wahlund-Effekt Inzucht, Fitnesseffekte p1 = 0.3, q1 = 0.7 p2 = 0.6, q2 = 0.4 P1 = 0.09 H1 = 0.42 Q1 = 0.49 P2 = 0.36 H1 = 0.48 Q1 = 0.16 Pz = 0.225 Hz = 0.45 Qz = 0.325 Pe = 0.2025 He = 0.495 Qe = 0.3025 Homozygotenüberschuss!!! F-Statistiken Strukturierte Population Gesamtpopulation T FST Subpopulation S FIT FIS Individuen I 1-FIT = (1-FIS) * (1-FST) pz = 0.45 qz = 0.55