Allgemeines zu Nukleinsäurenachweisen
Werbung

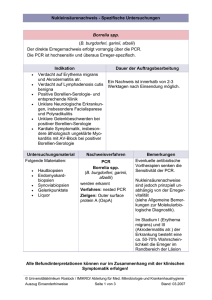
Nukleinsäurenachweis Allgemeine Bemerkungen zur Molekularbiologischen Diagnostik Allgemeine Angaben 1) Methodenbedingt ist der Nachweis nur einer oder weniger DNA-Zielsequenzen möglich. Damit erlaubt das Verfahren nur die Antwort auf sehr spezifische Fragestellungen ("Ist der Erreger X / das Gen Y im Material vorhanden?"), nicht aber auf allgemeine Fragestellungen "Enthält das Material pathogene Erreger?" oder "Weist der Patient Labormerkmale einer Infektion auf?". Besonderheiten des Molekularbiologischen Erregernachweises (PCR und mod. Verfahren) sind: sehr hohe Sensitivität und damit verbunden ein Kontaminationsrisiko die Sensitivitätsgrenze einer einfachen PCR liegt bei 5000 bis 50.000 Kopien der Ziel-DNA eine nested PCR kann eine Sensitivitätsgrenze von 10 Kopien der Ziel-DNA aufweisen Real-Time Verfahren mit fluoreszenzmarkierten Proben können eine Sensitivität von ~ 100 Kopien der Ziel-DNA aufweisen Wenn die Zielsequenz z.B. 10 mal im Genom eines Bakteriums vorhanden ist (z. B. TBC IS6110, 16S-rDNA-Gen der universellen eubakteriellen PCR), weist eine einfache PCR ~ 500 Bakteriengenomäquivalente (GE), eine nested PCR ca. 1-2 (!) GE nach. 2) Die PCR - im Gegensatz zu kulturellen Verfahren - unterscheidet nicht zwischen vitalen und abgetöten Erregern, da nur die erregerspezifische Nukleinsäure nachgewiesen wird. Somit ist eine PCR-Anforderung unter Therapie u. U. zum Erregernachweis (teilweise als einziges Verfahren) sinnvoll, nicht jedoch zur Therapiekontrolle (gilt insb. für TBC)! Spezielle Kommentare Eine gegen die 16S rDNA Gene gerichtete universelle eubakterielle PCR ist prinzipiell geeignet eine Vielzahl unbekannter Bakterien nachzuweisen und über einen Sequenzabgleich des PCR-Produktes mit publizierten Daten dieses zu identifizieren. © Universitätsklinikum Rostock / IMIKRO/ Abteilung für Med. Mikrobiologie und Krankenhaushygiene Auszug Einsenderhinweise Seite 1 von 2 Stand: 02.2007 Nukleinsäurenachweis Allgemeine Bemerkungen zur Molekularbiologischen Diagnostik Das Verfahren ist aber nur sinnvoll anzuwenden bei primär sterilen Materialien und der Erwartung eines einzigen Erregers als Krankheitsursache. Bei einer evtl. Mischflora erhält man überlagerte Sequenzen. Diese sind - bei rationalem Ressourcen-Einsatz - mit diesem Verfahren nicht sinnvoll zu untersuchen. Geeignete Materialien für die universelle 16S rDNA PCR sind somit: Liquor EDTA-Blut ggfs Bioptate von primär sterilen Organen wie Leber, Herz, Hirn etc. © Universitätsklinikum Rostock / IMIKRO/ Abteilung für Med. Mikrobiologie und Krankenhaushygiene Auszug Einsenderhinweise Seite 2 von 2 Stand: 02.2007