Proteine
Werbung

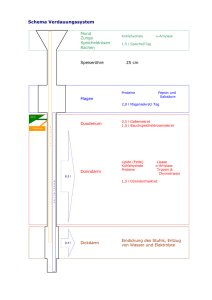
PROTEINE Biochemie-Protokoll Gruppe 1 13.05.16 Julia Gnann & Christoph Hartmann Einleitung Peptidbindung Bezeichnung für die Carbonsäureamid-Gruppierung –CO–NH–, die entsteht, wenn die CarboxyGruppe (–COOH) einer Aminosäure (im folgenden: AS) mit der Amino-Gruppe (–NH2) einer anderen AS unter Wasserabspaltung reagiert. Die Peptidbindung ist das strukturelle Charakteristikum der Peptide und Proteine. Wie andere Amid-Bindungen auch, unterliegt die P.-B. folgender Mesomerie: -O O C N C N+ H Abbildung 1: Mesomerie der Peptidbindung Daraus resultiert zum einen ihre relative chemische Stabilität und zum andern eine eingeschränkte Drehbarkeit um die C–N-Achse infolge eines partiellen Doppelbindungscharakters; die 4 an der Peptidbindung beteiligten Atome liegen in einer Ebene – meist in trans-Konfiguration, außer Aminoseitig von L-Prolin. Diese Beschränkungen sind von Bedeutung für die Sekundärstruktur der Proteine. Die Knüpfung der Peptidbindung erfolgt bei der Protein-Biosynthese in den Ribosomen, kann aber auch durch andere Enzyme katalysiert werden. Die Spaltung der Peptidbindung kann chemisch (z.B. durch halbkonz. Salzsäure, Hydrazin) oder enzymatisch (durch Peptidhydrolasen) erfolgen. Peptide (von griech.: peptos = verdaulich). Kondensationsprodukte von AS. H2N R1 O CH C NH Bezeichnung R2 O CH C .... für durch NH Peptid-Bindungen Rn O CH C verknüpfte OH Abbildung 2: Allg. Strukturformel der Peptide Bauen sich die Moleküle aus 2 AS-Resten auf, so spricht man von Dipeptiden, bei 3 und mehr von Tri, Tetra-, Pentapeptiden etc.; Proteine mit 2–10 AS-Resten faßt man als Oligopeptide, solche mit 10– 100 als Polypeptide zusammen, doch ist der Übergang von den letzteren zu den makromolekularen Proteinen fließend. Proteine mit Bindungen zwischen den seitenständigen Aminogruppen von Diaminocarbonsäuren (z.B. Lys) und seitenständigen Carboxy-Gruppen von Aminodicarbonsäuren (z.B. Glu, Asp) statt der üblichen Peptid-Bindungen zwischen α-NH2 und α-COOH nennt man Isopeptide. Zur Schreibung von Protein-Formeln benutzt man meist die 1- oder 3-Buchstaben-Notationen für die Aminosäuren. So steht z.B. AG bzw. Ala-Gly für L-Alanylglycin; falls nicht anders gekennzeichnet, steht links die Amino-Gruppe und rechts die Carboxy-Gruppe. Bedeutung: Auf die Bedeutung der makromolekularen Proteine für pflanzliche und menschliche Organismen wird bei Proteinen ausführlich eingegangen. Eine gleichermaßen spezifische Rolle spielen Oligo- und Polypeptide im menschlichen Organismus z.B. als Hormone (Peptidhormone) und Neurotransmitter und Neuromodulatoren (Neuropeptide). Für die physiologische Wirkung der Proteine ist neben der Konfiguration die Konformation und die Dynamik von Bedeutung , und natürlich benötigen die Proteine, um als Mediatoren wirksam werden zu können, spezifische Rezeptoren. Bei der zellulären Immunantwort werden Antigene (Fremd-Proteine) von Antigen-präsentierenden Zellen zu Proteinen abgebaut, und an der Zelloberfläche den TLymphocyten zum „Abtasten“ dargeboten; von außen verabreichte Proteine werden ebenfalls präsentiert. Protein-Ester können für süßen oder bitteren Geschmack verantwortlich sein, und wieder andere Proteine treten als Toxine pflanzlichen oder tierischen Ursprungs in Erscheinung. Auch unter Seite 1/8 Biochemie-Protokoll Gruppe 1 PROTEINE 13.05.16 Julia Gnann & Christoph Hartmann den Antibiotika finden sich eine Reihe von Proteinen (Peptid-Antibiotika), die z.T. AS der „unnatürlichen“ D-Konfiguration enthalten. Viele der physiologisch aktiven Proteine liegen als Cyclopeptide vor. Membran-assoziierte Peptide nehmen amphiphile Sekundärstrukturen (s. Proteine) an . Biosynthese: Die Biosynthese von Peptiden geschieht meist durch enzymatisches „Ausschneiden“ aus Proteinen (Propeptiden), die nach Maßgabe des genetischen Codes in den Ribosomen gebildet werden. Proteine (von griech.: proteuein = „der Erste sein“) Sammelbezeichnung für natürlich vorkommende Copolymere, die sich aus 20 verschiedenen α-Aminosäuren als Monomeren zusammensetzen. Von den nahe verwandten Polypeptiden werden sie aufgrund ihrer molekularen Größe unterschieden: Ab etwa 100 Monomer-Einheiten (AS-Resten) spricht man meist von Proteinen. Es ergeben sich Molmassen von 10000 bis mehrere Millionen. Nach der Zusammensetzung trifft man die Einteilung in einfache Proteine, deren Hydrolyse nur AS gibt, und zusammengesetzte Proteine, die außer AS essentielle Nichtproteinkomponenten – die prosthetischen Gruppen – enthalten: Nucleoproteine (Nucleinsäuren; z.B. Chromatin), Glykoproteine (Kohlenhydrate; z.B. Immunglobuline, Blutgruppensubstanzen), Lipoproteine (Lipide), Phosphoproteine (Phosphorsäure; z.B. Casein, Vitelline), Chromoproteine (Farbstoffe; z.B. Hämoglobin, Cytochrome), Metallproteine (Metalle; z.B. Transferrin und auch Eisenproteine) u.v.m. Bedeutung und Funktion: Proteine sind in der belebten Welt allgegenwärtig. Neben Kohlenhydraten und Fetten sind sie die dritte große Gruppe von Nahrungs- und Reservestoffen. Auf der Anwesenheit bestimmter Proteine beruhen Struktur, Funktion und Stoffwechsel aller lebenden Zellen und Gewebe. Man findet sie gleichermaßen in Tieren, Pflanzen und Mikroorganismen, so z.B. in den Muskeln (Actin, Myoglobin, Myosin), im Blut (Hämoglobin), usw. Vielfältig sind auch die Funktionen der Proteine im Organismus: Als Enzyme, Transport- und Speichermoleküle (Ferritin, Hämoglobin), molekulare Motoren (Dynein, Kinesin, Myosin), Gerüstsubstanzen (Sklerop., Gerüst-Eiweiß), in der Immunabwehr (Immunglobuline, Komplement), Hormone (Follitropin, Thyreotropin), Hormon- und Neurotransmitter-Rezeptoren, Regulatoren (EnzymInhibitoren, Transkriptionsfaktoren), usw. Eigenschaften: Die meist gut wasserlöslichen Proteine (Ausnahme: Membran-Proteine) sind gegen physikalische und chemische Einwirkung im allgemeinen ziemlich empfindlich. So gerinnt z.B. das Hühner-Eiweiß oberhalb 65°C; man bezeichnet diesen Vorgang als Denaturierung. Er beruht auf einer Zerstörung der Raumstruktur der Proteine unter Aufbrechen der schwachen innermolekularen Wechselwirkungen (vgl. unten den Abschnitt zur Struktur). Denaturierende Agentien sind z.B. Harnstoff, Natriumdodecylsulfat, elektrische Ladungen, Säuren, Schwermetallsalze usw. Schonendere Ausflockungen ohne bedeutende Denaturierung können z.T. durch Alkohol, Ammoniumsalze erreicht werden. Bei dieser Ausfällung erfolgt eine Schwächung der Hydrathülle der Proteine. Bei der Quellung werden Wassermoleküle von den Proteinmolekülen gebunden. Aufgrund der ionisierbaren Seitenketten der sauren AS Asparaginsäure und Glutaminsäure, der basischen AS Lysin, Arginin und Histidin, sowie der freien Amino- und Carboxy-Gruppe an den Enden der Polypeptid-Kette kommt dem Protein ein amphoterer Charakter zu, und es nimmt in Abhängigkeit vom pH-Wert eine jeweils verschiedene elektrische Gesamtladung an; der pH-Wert, bei dem diese verschwindet, heißt isoelektrischer Punkt (IP). Bei ihm ist die Wasserlöslichkeit des Proteins am geringsten. Struktur: Der für Proteine besonders kennzeichnende Stickstoff-Gehalt ist auf ihre Grundbausteine, die AS, zurückzuführen. Mit Hilfe von Säuren, Laugen oder Enzymen lassen sich alle Proteine nahezu restlos hydrolytisch in AS zerlegen. Die Analyse dieser Hydrolysate ergibt, daß Proteine – neben selteneren AS – immer wieder dieselben sog. proteinogenen 20 AS enthalten, wenn auch in unterschiedlichen Anteilen und nicht immer alle zugleich, nämlich Glycin (Gly), L-Alanin (Ala), L-Serin (Ser), L-Cystein (Cys), L-Phenylalanin (Phe), L-Tyrosin (Tyr), L-Tryptophan (Trp), L-Threonin (Thr), LMethionin (Met), L-Valin (Val), L-Prolin (Pro), L-Leucin (Leu), L-Isoleucin (Ile), L-Lysin (Lys), L-Arginin (Arg), L-Histidin (His), L-Asparaginsäure (Asp), L-Asparagin (Asn), L-Glutaminsäure (Glu) und LGlutamin (Gln). Alle optisch aktiven AS der Proteine haben also L-Konfiguration, was im folgenden bei Nennung einzelner AS nicht mehr speziell angegeben wird. Peptid-Bindung (sh.a. dort): Der Zusammenschluß dieser AS zu den hochmolekularen Proteinen geschieht durch die Bildung von Säureamid-Bindungen zwischen den Carboxy- und Amino-Gruppen verschiedener AS-Moleküle. Demnach kann man Peptide und Proteine mit der folgenden gemeinsamen Strukturformel charakterisieren: Seite 2/8 PROTEINE Biochemie-Protokoll Gruppe 1 Amino-Terminus H2N R1 O CH C 13.05.16 Julia Gnann & Christoph Hartmann c) NH Carboxy-Terminus R2 O CH C AS-Rest 1 AS-Rest 2 Wassers toffbrück enbindung NH R3 O CH C AS-Rest 3 NH Rn O CH C OH AS-Rest n Abbildung 3: Verknüpfung der AS in Proteine durch Peptidbindung =C =H =N =O = Seitenket te Die Peptidbindung kann auch zum Ring geschlossen werden. Am Aufbau des fortlaufenden Teils der Peptid-Kette ist also jeder AS-Baustein mit dem gleichen Anteil –CO–C(R)H–NH– beteiligt; nur die außerhalb der Kette liegenden Reste R variieren. Mit Hilfe von in den Seitenketten enthaltenen Aminound Carboxy-Gruppen bilden einige Proteine jedoch auch sog. Isopeptid-Bindungen aus. Die Polypeptid-Ketten eines makromolekularen Proteins sind sowohl in Lösung als auch im Kristall in charakteristischer Weise gewunden und gefaltet und besitzen unter gegebenen Bedingungen eine ganz bestimmte Konformation. Bei der Struktur der Proteine unterscheidet man zwischen Primär-, Sekundär-, Tertiär- und Quartärstruktur. Für diesen Strukturaufbau sind nicht nur die SäureamidBindungen, sondern darüber hinaus kovalente Disulfid-Brücken und verschiedene Arten von Nebenvalenzbindungen (zwischenmolekulare Kräfte) maßgebend, unter diesen besonders die Wasserstoff-Brückenbindung (in Abb. 3 durch Punktlinien dargestellt). Die Primärstruktur wird durch das Zusammentreten der AS zum Protein unter Knüpfung der Peptidbindung ausgebildet und ist durch die jeweilige Reihenfolge (Sequenz) der AS charakterisiert. Sie wird beginnend mit der AS, die eine freie α-Amino-Gruppe besitzt (Amino-Terminus, s. Abb. 1 u. 2) unter Benutzung des Drei- oder Einbuchstabencodes angegeben. sserstoffbrückenbindungen des Typs N–H···O=C zwischen den Atomen des Peptid-Rückgrats sind für die Ausbildung der Sekundärstruktur verantwortlich. Darunter versteht man gewisse regelmäßige, d.h. vom „Zufallsknäuel“ abweichende lokale Faltungsmuster, v.a. die schraubenförmige, rechtsgewundene α-Helix und das durch parallele oder antiparallele Anordnung mehrerer Abschnitte der Polypeptid-Kette zustande kommende β-Faltblatt (Abb. 3). Die Struktur der α-Helix ist wie folgt zu charakterisieren: 3,6 Aminosäure-Reste pro Windung, 0,54 nm Ganghöhe, 1,05 nm Gesamtdurchmesser. Die Seitenketten weisen nach außen, Wasserstoffbrücken bilden sich ungefähr in Richtung der Helix-Längsachse. Durch Prolin-Reste wird die Konformation der α-Helix gestört. Beim β-Faltblatt liegen die Peptid-Ketten in nahezu gestreckter Konformation vor, Seitenreste stehen abwechselnd nach beiden Seiten senkrecht von der Faltblatt-Ebene ab, die Wasserstoff-Bindungen liegen in dieser. Abbildung 4: Helix- und Faltblattstruktur Seite 3/8 Biochemie-Protokoll Gruppe 1 PROTEINE 13.05.16 Julia Gnann & Christoph Hartmann Unter Tertiärstruktur versteht man die jeweilige räumliche Anordnung der Peptid-Kette sowie der ASSeitenreste, die durch Disulfid-Brücken, durch Wasserstoffbrückenbindungen, durch ionische und durch hydrophobe Wechselwirkungen, meist zwischen AS-Seitenketten, stabilisiert wird. Die HBrückenbindung zwischen AS-Seitenketten, besitzt ihre größte Bedeutung im Inneren des Proteins. Dort befinden sich aufgrund hydrophober Bindung v.a. unpolare (hydrophobe) AS-Reste, während sich die polaren und geladenen Seitenketten in wäßriger Lösung eher nach außen wenden. Auch ohne Disulfid-Brücken können allein durch Nebenvalenz-Stabilisierung mehrere Polypeptid-Ketten zu einer funktionellen Einheit verbunden sein. Eine Quartärstruktur liegt dann vor, wenn ein Protein nicht aus einer einzigen Polypeptid-Kette besteht, sondern aus einer definierten Anzahl solcher Ketten (Untereinheiten), die untereinander nicht durch Peptidbindungen, sondern durch intermolekular wirkende Kräfte zusammengehalten werden. Dabei können sich Konformationsänderungen der einen Untereinheit den übrigen mitteilen und auch bei diesen zu Veränderungen führen (Kooperativität). Die vier genannten Strukturniveaus (Primär-, Sekundär-, Tertiär- und Quartärstruktur) sind voneinander abhängig. So kann man – wenn auch noch mit mangelnder Treffsicherheit – die Elemente der Sekundärstruktur (Helix, Faltblatt) eines bestimmten Proteins aus dessen AS-Sequenz ableiten . Die Raumstruktur der Proteine darf man sich übrigens nicht vollkommen starr vorstellen. Ihre Flexibilität ermöglicht es erst, ihre Funktionen als Enzyme, Rezeptoren usw. zu erfüllen. Biosynthese: Die Biosynthese der Proteine aus AS oder Translation findet in eukaryotischen Zellen an den Ribosomen des Cytoplasmas, des rauhen endoplasmatischen Retikulums (rER) und der Kernhülle statt. Der Zyklus für jede AS verläuft in folgenden sich wiederholenden Einzelschritten: 1. Bindung der AS an ihre zugehörigen Transfer-Ribonucleinsäuren (tRNA), unter Katalyse spezifischer Aminoacyl-tRNA-Synthetasen. 2. Bindung derjenigen Aminoacyl-tRNA an den Nucleoprotein-Komplex des Ribosoms, die mit ihrem Anticodon zu einem 3 Nucleotide großen Bereich (Codon) der Ribosom-gebundenen MessengerRibonucleinsäure (mRNA) paßt. Letztere bestimmt also die Aufeinanderfolge der AS. 3. Übertragung eines Peptids einer gleichfalls am Ribosom gebundenen Peptidyl-tRNA des Ribosoms auf die Aminoacyl-tRNA (Elongation). 4. Dissoziation der aus der Peptidyl-tRNA freigesetzten tRNA vom Ribosom, Einnahme ihres Platzes am Ribosom durch die neue Peptidyl-tRNA und Verschiebung der Messenger-tRNA um 3 Nucleotid-Einheiten (Translokation). Die Synthese der Proteine beginnt am Amino-Ende und endet am Carboxy-Terminus. Man nimmt an, daß sie sich schon während des Synthesevorgangs zu falten beginnen . Viele sekretorische Proteine tauchen auch schon während ihrer Synthese mit ihren aminoterminalen Erkennungssequenzen (Signalpeptiden) in die Membran des rER ein und werden dort eingeschleust. Im Cytoplasma synthetisierte, für den Import vorgesehene Proteine besitzen ebenfalls Signalsequenzen. Während der Translation finden mit der Acetylierung und/oder Entfernung eines Methionin-Rests bei Eukaryonten bereits Modifizierungen des Amino-Terminus des entstehenden Proteins statt. Im rER, im Golgi-Apparat aber auch im Cytoplasma erfolgen etliche posttranslationale Modifikationen, (Acylierung , Carboxylierung, Glykosylierung, usw.). Proteohormone und sekretorische Enzyme müssen schließlich noch aus Vorstufen (Prohormonen bzw. Proenzymen oder Zymogenen) „herausgeschnitten“ werden . Abbau: Von außen mit der Nahrung zugeführte Proteine werden im Verdauungstrakt, körpereigene dagegen meist intrazellulär zu AS abgebaut. Bei der Verdauung erfolgt im Magen und Darm eine Aufspaltung in Peptide bzw. AS unter dem Einfluß Eiweißspaltender Enzyme (Proteasen), die allerdings zuvor erst aus ihren Zymogenen freigesetzt werden müssen. Bei den Proteasen unterscheidet man zwischen Endopeptidasen, die Proteine nur innerhalb der Peptid-Kette spalten (Pepsin im Magen, Trypsin und Chymotrypsin im Dünndarm) und Exopeptidasen, die nur am Ende der Peptid-Ketten angreifen (Carboxypeptidase, Aminopeptidase, Dipeptidase im Darm). Die Spaltprodukte wandern durch die Darmwand und werden in den arbeitenden Zellen zu CO 2 und H2O oxidiert (Katabolismus) oder aber mit Hilfe von Nucleinsäuren und Enzymen zu arteigenen Eiweißstoffen zusammengefügt (Anabolismus). Beim vollständigen Abbau ergibt 1 g Protein die Energie von etwa 17,2 kJ (4,1 kcal.). Bei Proteolyse innerhalb von tierischen Zellen spielen auch Lysosomen eine wichtige Rolle. Dabei werden Proteine, die bestimmte AS-Sequenzen enthalten, schneller abgebaut als andere. Seite 4/8 PROTEINE Biochemie-Protokoll Gruppe 1 13.05.16 Julia Gnann & Christoph Hartmann Versuche Denaturierung und Löslichkeit von Proteinen Fällung durch Neutralsalze Mittels Fällung durch Neutralsalze kann man z.B. verschiedene Proteine des Blutserums fraktionieren, da sie bei verschiedenen Salzkonzentrationen ausfällen. 2ml Serum + 2ml Ammoniumsulfatlösung Globuline fallen aus. Zum Filtrat gibt man 2 Messerspitzen Ammoniumsulfat Albumin fällt aus. Diese Art der Denaturierung ist in beiden Fällen reversibel, man benötigt beim GlobulinNiederschlag viel H2O. Hitzedenaturierung Irreversible Denaturierung des Serums durch Kochen im Wasserbad. Alkoholfällung 2ml Serum + 5-10ml 96%iger Alkohol Niederschlag. Diese Fällung ist reversibel. Bei der Hitzedenaturierung wird die Tertiärstruktur des Proteins irreversibel zerstört. Bei der Alkoholfällung kommt es zu einer reversiblen Veränderung der Hydrathülle und bei Fällung mit Salzen zur Konkurrenz um Hydratwasser. Bestimmung des isoelektrischen Punktes (IP) von Casein (Eichgerade sh. Anhang) RG-Nr. 1 berechneter 3,99 pH-Wert gemessener 6,01 pH-Wert beobachtete 0 Fällung E595nm Proteingehalt [μg/ml] 2 3 4 5 6 7 8 9 3,38 3,68 3,53 3,375 3,22 3,07 2,92 2,77 5,7 5,3 5 4,6 4,25 4 3,68 3,35 0 0 0 +++ ++ + 0 0 1,288 1,289 1,402 1,367 0,074 0,154 0,849 1,268 1,279 390 391 >400 399 15 27 219 381 384 ↑ Isoelektrischer Punkt von Casein Fragen Warum weichen die gemessenen pH-Werte von den berechneten ab? Pufferwirkung von Casein Welche Werte sind für die Bestimmung des IP relevant? Der pH, bei dem die geringste Proteinkonzentration gemessen wurde, d.h., bei dem die stärkste Ausfällung (=schlechteste Löslichkeit) erfolgte, entspricht dem IP des Caseins. Warum ist die Löslichkeit eines Proteins am IP am niedrigsten? Aufgrund der ionisierbaren Seitenketten der sauren AS Asparaginsäure und Glutaminsäure, der basischen AS Lysin, Arginin und Histidin, sowie der freien Amino- und Carboxy-Gruppe an den Enden der Polypeptid-Kette kommt dem Protein ein amphoterer Charakter zu, und es nimmt in Abhängigkeit vom pH-Wert eine jeweils verschiedene elektrische Gesamtladung an; der pH-Wert, Seite 5/8 PROTEINE Biochemie-Protokoll Gruppe 1 13.05.16 Julia Gnann & Christoph Hartmann bei dem diese Gesamtladung verschwindet, heißt isoelektrischer Punkt (IP). Bei ihm ist die Wasserlöslichkeit des Proteins am geringsten, da es ungeladen/hydrophob ist. Trennung eines Proteingemisches mit der Gelfiltration Die Gelchromatographie ist eine als Säulenchromatographie durchgeführte Flüssigkeitschromatographie (s. Abb.4). Die stationäre Phase besteht aus Perlen mit einem heteroporösen gequollenen Netzwerk, dessen Porengrößenverteilung über mehrere Größenordnungen variiert, so daß die Fraktionierung nach Molekülgröße (Molekularsieb-Effekt) erfolgt. Eine flüssige Phase mit dem gelösten Polymeren wird durch das Gel gegeben, wobei die Polymer-Moleküle in alle Teile des Netzwerkes diffundieren, die ihnen aufgrund ihrer Größe versperrt sind. Folglich dringen die kleineren Moleküle des Gelösten tiefer ein und werden länger in der Säule zurückgehalten als die größeren Moleküle. Solche Moleküle, die größer sind als die größten Poren des gequollenen Gels, können die Gelkörner nicht durchdringen und wandern an diesen vorbei; sie verlassen die Säule zuerst. Die Moleküle erscheinen daher im Eluat in der Reihenfolge abnehmender Größe. In unserem Fall besteht das aufzutrennende Gemisch aus Hämoglobin und Fluoreszin, wobei Hb mit 64kDa das große und Fluoreszin mit 332Da das kleine Molekül ist. Zuerst verläßt das rotbraune Hb die Säule, da es nicht in die Gelporen eindringen kann, und zuletzt erreicht das gelbe Fluoreszin das Ende der Säule (s.Abb.6). Abbildung 5:Gelchromatographie Abbildung 6 Bestimmung freier SH-Gruppen im nativen und denaturierten Ovalbumin DTNB reagiert im Alkalischen mit SH-Gruppen-haltigen Substanzen unter Freisetzung eines farbigen Anions. Die Konzentration des freigesetzten Anions ist gleich groß wie die Konzentration der freien SH-Gruppen in der Analyse und läßt sich bei λ = 412nm mit ε = 13600 moll*cm bestimmen. E V d Masse Protein mol Protein = Molekulargewicht mol SH-Gruppen = Zahl der SH-Gruppen pro Molekül = mol SH Gruppen mol Protein Häufig liegen SH-Gruppen nicht direkt zugänglich an der Oberfläche des Proteins. Damit möglichst alle SH-Gruppen mit DTNB reagieren können findet die Reaktion zusätzlich unter denaturierenden Bedingungen durch Zugabe von Harnstoff, Guanidin und SDS statt. Berechnung für RG 1: MGOvalbumin = 44000 Masse Protein = 1mg (0,1ml von 10mg/ml) 0,053 mol cm 0,003l = 1,17∙10-8mol 13600l 1cm 0,001g mol mol Protein = = 2,27∙10-8mol 44000 g mol SH-Gruppen = SH-Gruppen = 1,17 10 8 mol = 0,5 2,27 10 8 mol Seite 6/8 Biochemie-Protokoll Gruppe 1 PROTEINE 13.05.16 Julia Gnann & Christoph Hartmann RG-Nr. 1 2 3 4 Harnstoff X Guanidin X SDS X H2O X Ovalbumin X X X X E412nm 0,053 0,136 0,317 0,194 SH-Gruppen 0 1,32 3,08 1,89 Bei Glas 1 ist keine denaturierende Substanz zugegeben und die Zahl der Bindungsstellen ist gering. Nach Zugabe von denaturierenden Substanzen – RG 2-4 – steigt die Zahl der SH-Gruppen. dies läßt darauf schließen, daß die meisten SH-Gruppen des Ovalbumins unzugänglich in dessen Inneren liegen. Guanidin hat in diesem Fall die Tertiärstruktur des Ovalbumins am meisten zerstört, da in diesem Ansatz die meisten SH-Gruppen nachzuweisen waren. Fragen Welche Effekte sind die treibenden Kräfte für die Faltung einer PolypeptidKette zur nativen Konformation? Für den Strukturaufbau sind die Säureamid-Bindungen, kovalente Disulfid-Brücken und verschiedene Arten von Nebenvalenzbindungen (zwischenmolekulare Kräfte) maßgebend, unter diesen besonders die Wasserstoffbrückenbindung. Zusätzlich falten sich Proteine mit Hilfe sog. Chaperone. Welche Funktionen haben SH-Gruppen in Proteinen? Die SH-Gruppen bilden vornehmlich Disulfid-Brücken aus. Durch Disulfid-Brücken können in der Sequenz voneinander entfernte Bereiche einer Polypeptid-Kette oder mehrere Polypeptid-Ketten kovalent miteinander verbunden werden. Im Insulin finden sich z.B. zwei Disulfidbindungen zwischen den beiden Polypeptid-Ketten und eine dritte zwischen den AS-Resten 6 und 11 der sog. A-Kette. Spektrale Titration der Bromkresolgrün-Bindung an Albumin Gibt man zu einer konstanten Menge Bromkresolgrün steigenden Konzentrationen von BSA (Rinderalbumin) und mißt die ΔE-Werte (spektrale Titration), so erhält man beim Auftragen von ΔE gegen cBSA eine Kurve, die den Verlauf der Sättigung einer bestimmten Bromkresolgrün-Menge mit BSA wiedergibt. RG-Nr. 1 2 3 4 5 6 7 8 9 10 11 12 cBSA 0 0,3 0,6 1 1,6 3,3 3,3 6,6 10 16,6 33,3 50 ΔE630nm 0,012 0,044 0,062 0,1 0,163 0,165 0,230 0,251 0,257 0,261 0,27 (Kurve s. Anhang) Die beiden Geraden schneiden sich bei cBSA = 4,5μmol/l. cL tot = 12,5μmol/l Anzahl der Bindungsstellen = c L tot 12,5 = = 2,7 ≈ 3 Bindungsstellen. 4,5 c BSA Fragen Nennen Sie Beispiele, wo die Bindung niedermolekularer Substanzen an Serumalbumin beim Transport im Serum eine wichtige Rolle spielt. Albumin transportiert folgende Substanzen: Fettsäuren, Vitamin B12, Mg2+, Cu2+, Bilirubin, Cholesterin, Pharmaka, u.v.m. Seite 7/8 Biochemie-Protokoll Gruppe 1 PROTEINE Abbildungsverzeichnis: Abbildung 1: CD-Römpp, Chemielexikon; Thieme Abbildung 2: CD-Römpp, Chemielexikon; Thieme Abbildung 3: CD-Römpp, Chemielexikon; Thieme Abbildung 4: CD-Römpp, Chemielexikon; Thieme Abbildung 5: ckjh Abbildung 6: ckjh Quellen: CD-Römpp, Chemielexikon; Thieme Pschyrembel, 258. Auuflage; WdeG Taschenatlas Biochemie; Thieme Seite 8/8 13.05.16 Julia Gnann & Christoph Hartmann