Enzym
Werbung

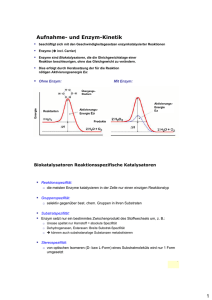
Pflanzenphysiologie Enzyme 7. Enzyme Enzyme sind Proteine (Proteide) mit katalytischen Eigenschaften. Sie ermöglichen den Ablauf vieler chemischer Reaktionen, die ansonsten unmerklich langsam verlaufen würden. Enzyme zeichnen sich insbesondere durch die Spezifität ihrer Katalyse aus. 7.1 Spezifität der Enzymkatalyse - Reaktionsspezifität: von mehreren Reaktionen, denen eine Substanz unterliegen kann, katalysiert ein Enzym immer nur eine. Beispielsweise können Aminosäuren a) decarboxyliert, b) transaminiert und c) isomerisiert werden; Enyme katalysieren nur eine dieser Reaktionen. - Substratspezifität: eine Reaktion erfolgt meist nur an einer von zahlreichen ähnlichen Verbindungen (z.B. nur einer von 21 Aminosäuren) z.B. Alanin + -Ketoglutarat (AAT) = Pyruvat + Glutamat. - Stereospezifität: Die substratspezifischen Reaktionen erfolgen stereospezifisch, es entsteht nur eine von zwei möglichen mesomeren Formen, z.B.: Pyruvat + NADH2 (LDH) = L-Lactat + NAD+. - pH-Optimum, Temperatur-Optimum Am aktiven Zentrum sind oft dissoziable Aminosäuren beteiligt. Ihre Ionisierungsform entscheidet über das Ausmaß der katalytischen Aktivität, deshalb haben Enzyme ein pH-Optimum. Aber auch das Ionenmilieu und die Ionenstärke beeinflussen die katalytische Aktivität. - Selbstkontrolle: DNA-Polymerase I katalysiert die Polymerisation von Nukleotiden nach Vorgabe eines DNA-Einzelstranges. Hierbei wird in weniger als einem von 106 Fällen ein falsches Nukleotid in den neuen DNA-Strang eingebaut, da falsch eingebaute Nukleotide sofort hydrolysiert werden. 7.2 Einteilung der Enzyme in sechs Klassen 1. Oxidoreduktasen (Oxidations-Reduktions-Reaktionen) z.B.: Alkohol + NAD (ADH) = Acetaldehyd + NADH2 2. Transferasen (Übertragung von funktionellen Gruppen) z.B.: ATP + Cholin (Cholinkinase) = ADP + Phosphocholin 3. Hydrolasen (Spaltung von Molekülen unter Wasseraufnahme) z.B.: Ester + H2O (Esterase) = Alkohol + Säure 4. Lyasen (Lösen von C-C, C-O, C-N u.a. Bindungen) z.B.: Isocitrat (Isocitrat Lyase) = Glyoxylat + Succinat 5. Isomerasen (intramolekulare Umlagerungen) z.B.: Fructose-6-phosphat (Hexosephosphat Isomerase) = Glucose-6-phosphat 6. Ligasen (Knüpfen kovalenter Bindungen zwischen Molekülen unter ATP-Verbrauch) z.B.: Acetat + CoA + ATP (Acetyl-CoA-Synthetase) = Acetyl-CoA + AMP + PPi Disk: File: Pflanzenphysiologische Vorlesung Kurzfassung PFLA-7K 1 Pflanzenphysiologie Enzyme Jedes Enzym erhält von einer internationalen Kommission eine vierstellige IdentifikationsNummer (EC-Nummer), z.B.: Lactat Dehydrogenase (EC 1.1.1.27) 1. Oxidoreduktase 1.1 Wirkung auf CH OH Gruppen 1.1.1 NAD als Coenzym, und 1.1.1. 27 als Individual-Nummer. 7.3 Regulatorische Enzyme Enzyme wirken nicht nur als Katalysatoren, sondern auch als Stellglieder im Stoffwechsel, insbesondere die regulatorischen Enzyme. - Endprodukt-Kontrolle: das Endprodukt eines Syntheseschritts wirkt (hemmend) auf das Eingangsenzym der Reaktion zurück. Die Hemmung ist reversibel, geht die Konzentration des Endproduktes zurück, wird das Enzym wieder entsprechend aktiver (Beispiel: ThreoninDesaminase, Hemmung durch Isoleucin). - Isoenzym-Kontrolle und Rückkopplung: Am Beginn eines verzweigten Biosynthese-Weges stehen mehrere (Iso)-Enzyme, die spezifisch von den jeweiligen Endprodukten gehemmt werden (Beispiel: Aspartokinase 1, Hemmung durch Lysin; Aspartokinase 2, Hemmung durch Threonin; Aspartokinase 3, Hemmung durch Methionin). - Aktivierung von Enzymreaktionen durch Ca-Calmodulin, Inaktivierung durch Ca-freies Calmodulin. - Covalente Modifikation von Enzymen durch andere, regulatorische Enzyme (z.B. Aktivierung/Inaktivierung von Stärke-Phosphorylase durch Phosphorylierung/ Dephosphorylierung des Enzyms durch zwei verschiedene regulatorische Enzyme. - Erzeugung von inaktiven Proformen, die am Wirkort katalytisch aktiv werden (z.B. viele Verdauungsenzyme). 7.4 Aktivierungsenergie Enzyme beschleunigen chemische Reaktionen (ohne daß sie dabei verbraucht werden), indem sie die Aktivierungsenergie der Reaktion herabsetzen. Beispiel: Der spontane Zerfall von Wasserstoffperoxid benötigt eine Aktivierungsenergie von 75,4 kJ/mol, die Katalase katalysierte Reaktion (H2O2 + H2O2 (Katalase) = 2 H2O + O2) nur 13 kJ/mol. Die Aktivierungsenergie kann nach Arrhenius über folgende Gleichung ermittelt werden: v = A . e –Ea/RT, wobei v = Reaktionsgeschwindigkeit ( - S /t; P/ t; S = Substrat; P = Produkt), Ea = Aktivierungsenergie (J/mol), A = Proportionalitätsfaktor, e = Basis der natürlichen Zahlen, R = 8,315 (J/ mol. K) und T = absolute Temperatur (K). Nach Logarithmieren erhält man eine Geradengleichung ln v = - Ea/ RT + ln A; d.h. es besteht ein linearer Bezug zwischen ln v und 1/T. Aus der Steigung der Geraden in einem Plot von ln v gegen 1/T ergibt sich die Aktivierungsenergie zu: Steigungswert = Ea . R (J/mol). Disk: File: Pflanzenphysiologische Vorlesung Kurzfassung PFLA-7K 2 Pflanzenphysiologie Enzyme 7.5 Molekulare Masse einiger Enzyme Mr g/ mol Enzym Ribonuclease aus Aspergillus oryzae Cellulase aus Polyporus - Chymotrypsin Papain Trypsin Peroxidase aus Tulpen Peroxidase aus Merrettich -Amylase, Pankreas Malat Dehydrogenase (Ochsenherz, Mitochondrien) Phosphoglucomutase Peroxidase (Milch) Luciferase (Leuchtkäfer) Aldolase (Leber) Succinat-Dehydrogenase Glycerat-Kinase (Hefe) Urease (Pferdebohnen) ATPase (Kaninchenmuskel, Myosin) Pyruvat Decarboxylase 11.000 11.400 15.000-25.000 20.700 23.800 34.300 40.000 45.000 62.000 74.000-78.000 82.000 100.000 159.000 200.000 251.000 480.000 540.000 1.000.000 Vgl. zu Kapitel Enzyme auch Fig. 32 Modell der Adenylat Kinase, Fig. 3.33 a Modell der Adenylat Kinase, Abb. 50 Modell des Lysozyms, Abb. 52 Hydrolyse des an Lysozym gebundenen Mischpolymeren, Disk: File: Pflanzenphysiologische Vorlesung Kurzfassung PFLA-7K 3 Pflanzenphysiologie Enzyme 7.6 Enzymkinetik 7.6.1 Katalytische Aktivität Die katalytische Aktivität von Enzymen wird nach der Menge umgesetzten Substrates (gebildeter Menge Produkt) pro Zeiteinheit beurteilt. Unter optimalen Reaktionsbedingungen und bei Substratsättigung gilt: 1 Katal (kat) ist diejenige Menge Enzym, die 1 mol Substrat pro Sekunde umsetzt. Daneben ist noch die internationale Einheit IE bzw. Unit (U) in Gebrauch. 1 IE (U) = Enzymmenge, die pro Minute 1 mol Substrat umsetzt = 16,67 nkat 1 nkat = 0,06 IE (U). Als abgeleitete Einheiten werden verwendet: die Volumenaktivität (kat/Liter) und die spezifische Aktivität (kat/g Protein). Die Wechselzahl gibt an, wieviele Moleküle Substrat (Produkt) von einem Enzymmolekül pro Sekunde umgesetzt werden. Zwischen Reaktionsgeschwindigkeit und Enzymkonzentration besteht bei optimalen Bedingungen ein linearer Zusammenhang. 7.6.2 Michaelis-Menten-Kinetik Enzyme mit Michaelis-Menten-Kinetik sind solche, die einen hyperbolischen Zusammenhang zwischen Substratkonzentration und Reaktionsgeschwindigkeit (bei konstanter Enzymmenge) zeigen. Die Michaelis-Menten-Gleichung beschreibt diesen Zusammenhang: v = vmax / (1 + KM / S) bzw. v = vmax . S / (S + KM) Bei Substratkonzentrationen kleiner als KM gilt: v = S . vmax / KM d.h. die Reaktionsgeschwindigkeit ist der Substratkonzentration direkt proportional. Bei hohen Substratkonzentrationen, wenn S sehr viel größer als KM ist, wird v = vmax ; ab einer bestimmten, hohen Substratkonzentration nimmt die Reaktionsgeschwindigkeit nicht mehr zu. Der KM-Wert wird üblicherweise über einen Lineweaver-Burk-Plot ermittelt, d.h. nach Umformung der Michaelis-Gleichung in eine Geradengleichung: 1/ v = KM / vmax . 1/ S + 1/ vmax Die katalytische Wirkung von Enzymen verläuft über die Bildung eines labilen EnzymSubstrat-Komplexes: E + S - k1 ES - k2 E + P k2 E = Enzym, S = Substrat, ES = Enzym-Substrat-Komplex, P = Produkt, k1, k2, k3 = Geschwindigkeitskonstanten. Disk: File: Pflanzenphysiologische Vorlesung Kurzfassung PFLA-7K 4 Pflanzenphysiologie Enzyme Erfolgt ein Zerfall des ES-Komplexes sehr langsam in E + P aber schneller in E + S, so hat die Michaelis-Konstante die Bedeutung einer Dissoziationskonstanten: KES = E . S / ES = k2 / k1 Ein hoher KM-Wert zeigt unter diesen Bedingungen eine schwache Substratbindung an, ein niedriger eine feste Bindung. 7.6.3 Wechselzahl Die Wechselzahl eines Enzyms kann ermittelt werden, wenn alle Enzymmoleküle mit Substrat gesättigt sind und sowohl die umgestzte Substratmenge als auch die Enzymkonzentration im Reaktionsansatz bekannt sind (vmax = k3 ET; ET = Enzymmenge). Eine 10-6 M Lösung von Kohlensäure-Anhydratase katalysiert die Bildung von 0,6 M H2CO3 (aus CO2 und Wasser) pro Sekunde, die Wechselzahl ist demnach 0,6/ 10-6 = 6 x 105 (1/sec). Jeder Katalysevorgang erfolgt im Zeitintervall 1/ k3, für Kohlensäure-Anhydratase bedeutet dies 1/ 6 x 105 sec = 1,7 Mikrosekunden. Übliche Wechselzahlen: 1 bis 104 sec. 7.6.4 Allosterische Enzyme Allosterische Enzyme zeigen, im Gegensatz zu Enzymen mit Michaelis-Menten-Kinetik, eine sigmoide Beziehung zwischen Substrat-Konzentration und Reaktionsgeschwindigkeit. Allosterische Enzyme sind Stellglieder im Stoffwechsel, ihre Aktivität wird von allosterischen Effektoren positiv oder negativ beeinflußt. Das Enzym Aspartat-Transcarbamoylase katalysiert die folgende Reaktion: Carbamoylphosphat + Aspartat (AT) = N-carbamoyl-L-aspartat + Pi. Es ist ein allosterisches Enzym. Es steht am Anfang des Biosyntheseweges des Pyrimidinrings. In. E. coli wird das Enzym durch Cytidintriphosphat (CTP) gehemmt, dem Endprodukt des Biosyntheseweges. ATP ist dagegen Aktivator. CTP verstärkt den sigmoiden Verlauf zwischen Reaktionsgeschwindigkeit und Substratkonzentration, ATP mildert ihn ab. Die (unbeeinflußte) sigmoide Zunahme der Reaktionsgeschwindigkeit mit zunehmender Substratkonzentration zeigt, daß die Bindung der Substrate (Carbamoylphosphat und Aspartat) kooperativ erfolgt. Die kinetische Gleichung für allosterische Enzyme lautet allgemein: v = vmax . Sn / (KM + Sn) mit n = Gebrochene Zahl. Die Aspartat Transcarbamoylase besteht aus 6 (großen) katalytischen Untereinheiten und 6 (kleinen) regulatorischen Untereinheiten. Spaltet man die regulatorischen Einheiten ab, so reagiert das Enzym nicht mehr auf die Effektoren CTP und ATP. Disk: File: Pflanzenphysiologische Vorlesung Kurzfassung PFLA-7K 5 Pflanzenphysiologie Enzyme Prosthetische Gruppen und Zwischensubstrate Verbindung Funktion Vitamin Nicotinamid-adenindinucleotid (NAD+) Nicotinamid-adenindinucleotid-phosphat (NADP+) Flavinmononucleotid (FMN) Flavin-adenin-dinucleotid (FAD) Thioctansäure (-Liponsäure) Wasserstoffübertragung Nicotinamid Wasserstoffübertragung Nicotinamid Wasserstoffübertragung Wasserstoffübertragung Riboflavin Riboflavin Wasserstoffübertragung Hämine Biotin Tetrahydrofolsäure Thiaminpyrophosphat Elektronenübertragung CO2-Übertragung C1-Gruppenübertragung Aldehydübertragung, Decarboxylierung Acetyl-Übertragung Aminosäurendecarboxylierung, Transaminierung Methylgruppenübertragung Thioctansäure (Bakterienwuchsstoff) Biotin (Hefewuchsstoff) Folsäure Thiamin (Aneurin, Vitamin B1) Pantothensäure Pyridoxin Coenzym A Pyridoxalphosphat Adenosin-methionin Adenosintriphosphat (ATP) Phosphoadenylsäuresulfat Adenosindiphosphat (ADP) Uridindiphosphat (UDP) Cytidindiphosphat (CDP) - Ketoglutarat Glucose-1,6-diphosphat 2,3-Phosphoglycerinsäure Cobalamin Transfer-RNS Disk: File: Phosphat- und PyrophosphatÜbertragung, Adenylierung Schwefelsäureübertragung Zuckerübertragung Zuckerübertragung Phosphocholinübertragung NH2-Übertragung Phosphatübertragung Phosphatübertragung Isomerisierung (Verschiebung von Carboxylgruppen) Spezifische AminosäurenÜbertragung Pflanzenphysiologische Vorlesung Kurzfassung PFLA-7K Methionin (essentielle Aminosäure) Cobalamin - 6