Nomenklatur von Koordinationsverbindungen
Werbung
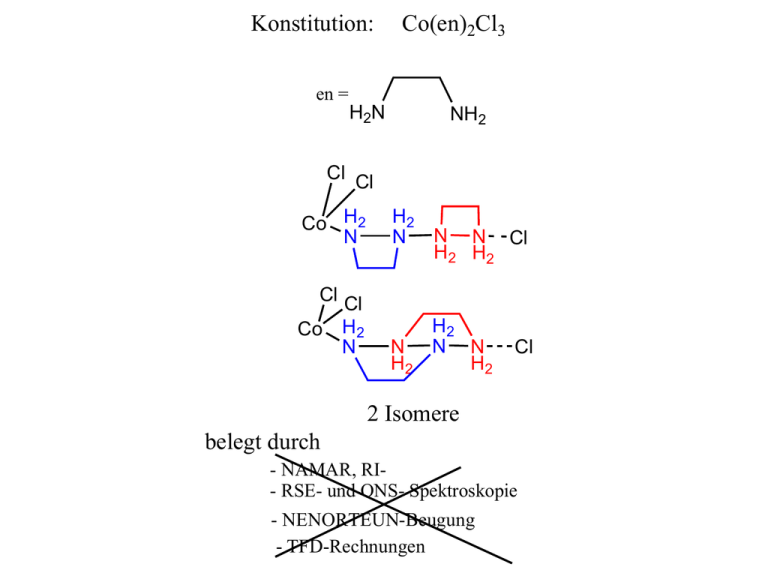
Konstitution:
Co(en)2Cl3
en =
H2N
NH2
Cl Cl
Co H2
N
Cl Cl
Co H2
N
H2
N
N
H2
N N Cl
H2 H2
H2
N N
H2
Cl
2 Isomere
belegt durch
- NAMAR, RI- RSE- und ONS- Spektroskopie
- NENORTEUN-Beugung
- TFD-Rechnungen
Werner-Typ Komplexe
Cl
H2N
Co
H2N
Cl
H2
N
N
H2
+
+
Cl
H2N
H2N
Alfred Werner Nobelpreis 1913
Co
NH2
Cl
NH2
[Me3NBF3]
BF3 + NMe3
F
F
F
F
B
B
F
F
+
Me
N
Me
N
Me
Me
Lewis-Säure
Akzeptor
Lewis-Base
Donor
Me
Me
BH3 + CO
[H3BCO]
O
C
B
H
H
H
IR((CO): 2167 cm-1
freies CO: IR((CO): 2143 cm-1
J. Am. Chem. Soc. 1996, 118, 12159
J. Chem. Phys. 1957, 26, 1118
Neutronenbeugungsstruktur H3N-BH3
B-H-Abstand: 115 pm
N-H-Abstand: 96 pm
H-H-Abstand 202 pm
J. Am. Chem. Soc. 1999, 121, 6337
EtMgI + PhBr
Et-Ph + MgIBr
Br
MgI
[Kat]
+
+ MgIBr
Ausbeute 0 %
[Kat] = dppeNiCl2 =
Ph2
P
P
Ph2
Ni
Cl
Cl
Ph2
P
P
Ph2
Ni
Cl
Cl
[Kat] = dpppNiCl2
Koordinationsverbindung
[Me3NBF3]
BF3 + NMe3
F
F
F
F
B
B
F
F
+
Me
N
Me
N
Me
Me
Me
Lewis-Säure
Akzeptor
BF3
+ F-
Me
Lewis-Base
Donor
[BF4-]
-
F
B
F
F
F
Definitionen
Komplexbildung:
- Änderung der Zahl der Atome oder Atomgruppen
=> Koordinationszahl & Liganden
- die an ein Zentralatom gebunden (koordiniert sind)
Geometrieänderung
z.B.
BF3 => keine Koordinationsverbindung/Komplex
hingegen: [BF4 ]
CoCl3 => Festkörper kein Komplex
hingegen [Co(NH3)6]3+
Koordinationssphäre
Koordinationszahl, CN
Oxidationszahl, OX
Komplex
CN Liganden Ox
[Ni(NH3)6]2+ 6
5 NH3
II
[CoCl4]24
4 ClII
[HCo(CN)5]3- 6 1 H-, 5 CN- III
[Fe(CO)5]
5
5 CO
0
[MnO4-]
5
4 O2VII
[Mn(CO)4]3+ 4
4 CO
III
Co(en)33+
3+
NH2
H 2N
H 2N
H2N
N
N
N
N
Co
H2N
NH2
H2N
NH2
H2N
"take home lesson"
NH2
Co
N
N
3+
NH2
NH2
N
N
N
N
N
N
- oktaedrisch Co(III) d6
- konfigurationsstabil da inert (t2g voll besetzt, eg* leer)
- chiral - Enantiomere
- Enantiomere separierbar
homoleptische Komplexe
- nur ein Ligandtyp
z.B. [NiCl4]2- [Fe(CN)6]3- [Fe(CN)6]4-
heteroleptische Komplexe
- mehr als ein Ligandtyp
z.B.
[MoCl4O2]2-
Exkurs:
2-
O
Cl
Mo
O
2-
O
Cl
Cl
Cl
Cl
Mo
Cl
Cl
Cl
O
cis
trans
IR-Spektroskopie (2 (Mo=O) Banden (880 und 920 cm-1)
cis-konfiguriert
Nomenklatur von Koordinationsverbindungen
a Aufstellung von Komplexformeln
Regel 1: Das Kation steht in der Formel immer vor dem Anion.
Komplexe werden in eckigen Klammern geschrieben
z.B. K4[Fe(CN)6]
Kaliumhexacyanoferrat(II)
[NBu4][BPh4]
Tetrabutylammoniumtetraphenylborat
Nomenklatur von Koordinationsverbindungen
Regel 2: Das Zentralatom steht in der Formel an erster Stelle.
z.B. [Fe(CN)6]4-, [PF6-]
Regel 3: Anionische vor neutralen Liganden.
Regel 4: Alphabetische Reihenfolge innerhalb der Ligandklassen.
Regel 5: Molekulare Komplexliganden sowie Abkürzungen in runde
Klammern.
Regel 6: Oxidationszahl als Exponent hinter Zentralatom.
z.B. [FeII(CN)6]4-, [CoIII(NO2)3(NH3)3], [CoIIBrCl(en)2]Cl
Nomenklatur von Koordinationsverbindungen
b Name
Regel 1: Liganden in alphabetischer Reihenfolge vor dem Namen
des Zentralatoms (als letztes!) mit di, tri,.. als Präfix
für Mehrfachliganden.
Regel 2: a) Angabe der Oxidationszahl des Zentralatoms als
römische Ziffern (z.B. I, II) oder
b) der Ladungszahl der Koordinationseinheit als
arabische Ziffern + Ladung (z.B. +3)
hierbei jeweils hinter dem Namen.
Regel 3: Namen anionischer Liganden enden auf -o, neutrale und
formal kationische ohne Zusatz.
Regel 4: Neutralliganden werden in runden Klammern.
Nomenklatur von Koordinationsverbindungen
Regel 1: Liganden in alphabetischer Reihenfolge vor dem Namen
des Zentralatoms (als letztes!) mit di, tri,.. als Präfix
für Mehrfachliganden.
z.B.
[Co(NH3)6]3+
Hexaammincobalt(III)
Ligand
Metall Ox.Stufe
Nomenklatur von Koordinationsverbindungen
Regel 2: a) Angabe der Oxidationszahl des Zentralatoms als
römische Ziffern (z.B. I, II) oder
b) der Ladungszahl der Koordinationseinheit als
arabische Ziffern + Ladung (z.B. +3)
hierbei jeweils hinter dem Namen.
[PtCl4]2Tetrachloroplatinat(II)
[Ni(CN)5]3
Pentacyanonickelat(3-)
Anionische Komplexe enden auf at
Beachte: Cu: Cuprat
Au: Aurat
Fe: Ferrat
Sn: Stannat
Pb: Plumbat
Ag: Argenat
Nomenklatur von Koordinationsverbindungen
Regel 3: Namen anionischer Liganden enden auf -o, neutrale und
formal kationische ohne Zusatz.
z.B. Br = Bromo
Mit folgenden Ausnahmen behalten Neutralliganden den Name des
Moleküls:
Ammoniak NH3
ammin
Kohlenmonoxid, CO
carbonyl
Wasser, H2O
aqua
Häufig verwendete Liganden
Formel
Ligandname
Formel
Ligandname
FClO2S2(SH)(SO4)2(C2O4)2N3P3(CN)(NCO)(NCS)(NH2)(NH)2(PH2)N3(NO3)(NO2)-
Fluoro
Chloro
Oxo, Oxido
Sulfido, Thio
Hydrogensulfido
Sulfato
Oxalato, Ethandionato
Nitrido
Phosphido
Cyano
Cyanato
Thiocyanato
Amido, Azanido
Imido, Azandiido
Phosphanido
Azido
Nitrato
Nitrito
H2
O2
H2O
H2S
H2S2
CO
CS
N2
NH3
PH3
P4
(CH3)3N
(CH3)3P
HN=NH
HP=PH
NO
NS
N2O
Diwasserstoff
Disauerstoff
Aqua
Sulfan, Hydrogensulfid
Disulfan, Hydrogendisulfid
Carbonyl
Thiocarbonyl
Distickstoff
Ammin
Phosphan
Tetraphosphor
Trimethylamin
Trimethylphosphan
Diazen
Diphosphen
Nitrosyl
Thionitrosyl
Distickstoffoxid
Nomenklatur von Koordinationsverbindungen
Ligand mit Präfix
Verwendung von:
z.B.
[Co(en)3]Cl3
Tris(ethylendiamin)cobalt(III) chlorid
3
Ligand
name
Monodentate Liganden
O
P
N
H
O
THF
N
Pyridin
S
H
ambident
H
Ammin
Trimethylphosphan
dmso
Dimethylsulphoxid
Bidentate Liganden
F3C
O
N
N
bipy, bpy
2,2´-Bipyridin
P
N
-
O
O
-
N
phen
1,10-Phenanthrolin
P
dppe
Diphenylphosphinoethan
O
CF3
P
acac
Acetylacetonat
P
dmpe
Dimethylphosphinoethan
N
N
daad
Diazadienderivat
H2N
hfac
Hexafluoroacetylacetonat
NH2
en
Ethylendiamin
N
N
tmeda
Tetramethylendiamin
Terdentate Liganden
H
-
H
N
N
H
B
N
N
N
N
N
N
H
Tp
Trispyrazolylborat
tacn
Triazacyclononan
Ir
N
N
N
N
N
terpy
2,2´,2´´-Terpyridin
N
N
Diimin-Pyridin
[Ir]=O
"eigene Küche"
O
Tetradentate Liganden
2N
2N
N
O
O
N
N
N
salen
por
Porphyrin
Tetradentate Liganden
2-
+
N
N
N
Mn
N
N
O
O
O
N
salen
por
Porphyrin
N
N
N
N
tren
-
O2CCH2
-
N
O2CCH2
N
CH2CO2-
CH2CO2-
EDTA
Ethylendiamintetraacetat
hexadentat
extrem guter Komplexligand
Benennung
[PtCl2(PPh3){SC(NH2)2}]
Dichloro(thioharnstoff)(triphenylphosphan)platin(II)
beachte: - zuerst Liganden, dann Zentralmetall
- unabhängig von Ladung
- Einhaltung der Ladung
- di-, tri; bis, tris etc. unberücksichtigt
[Mo(CO)4(PPh3)2]
Tetracarbonylbis(triphenylphosphan)molybdän(0)
bis,tris anstelle von di oder tri
zur Unterscheidung von Mehrdeutigkeiten
Nomenklatur
Cl
Cl
OC
PPH3
OC
PPh3
PPh3
PPH3
OC
Ru
Ru
H
Cl
fac
Ph3P
PPH3
Ru
H
Ph3P
PPh3 mer
[RuIIClH(CO)(PPh3)3]
Carbonylchlorohydridotris(triphenylphosphan)ruthenium(II)
nicht vollständig beschrieben Isomere/Stereochemie
H
PPh3
mer
Nomenklatur
3+
H2N
NH2
H2N
Co
NH2
H2N
H2N
3+
NH2
H2N
NH2
Co
H2N
NH2
NH2
Enantiomere
Tris(ethylendiamin)cobalt(III)
-
CN
NC
Ni
CN
CN
CN
NC
CN
NC
Ni
CN
CN
trigonale-Bipyramide quadratische Pyramide
Pentacyanonickelat(1-)
Strukturen und Isomere
• Isomere: Verbindungen mit gleicher
Molekülformel aber anderer Anordnung:
2 Varianten
Strukturen und Isomere
I) Konstitutionsisomere - verschiedene Verbundenheit
II) Stereoisomere - gleiche Verbundenheit
verschiedene Anordnung im Raum
Strukturen und Isomere
I) Konstitutionsisomere
z.B.
[Co(NH3)5(SO4)]Br
[Co(NH3)5Br]SO4
rote Verbindung
violette Verbindung
Stereoisomere
cis-Platin
trans-Platin
orangegelb
hellgelb
cancerostatisch
> 95 % Erfolg
Hodenkrebs
Löslichkeit 0.252 g/100 ml H2O 0.037 g/100 ml H2O
Hauptgruppenchemie VESPR-Regel
VESPR = Valence Shell Electron Pair Repulsion
sp2
H
H
N
VESPR-Vorhersage
Koordinationszahl
2
3
4
N
5
sp3
H
6
H
H
7
freie e--Paare mehr Platzbedarf als
8
bindendene e--Paare
9
H
Polyeder
lineare Anordnung
gleichseitiges Dreieck „Y“
Tetraeder
trigonale Bipyramide
Oktaeder
pentagonale Bipyramide
Dodekaeder
dreifach überkapptes Prisma
Methode von Kepert et al.
= Ligand
= Metallzentrum
inter-Ligand-Wechselwirkung
abstoßend !(repulsiv)
WW:
r-n –Abhängigkeit (n=6, 12) Optimierung
D.L. Kepert Inorganic Stereochemistry, Springer, Berlin 1982
Kepert Modell
richtige Vorhersage der Koordinationspolyeder
unabhängig von Metall!!!!
bis auf KZ = 4 hier Tetraeder immer bevorzugt!
0.2e15 pm
Korrektur durch zwei Punktladungen
15 pm
2Cl
„dz2-lone pair“
Cl
Pt
Cl
Cl
dz 2
d8 ML4
0.2e-
Koordinationszahlen
KZ = 1
sehr selten, nur mit sperrigen Liganden möglich,
z.B. 2,4,6-Triphenylphenylkupfer
Ag
Ag
Kristallstruktur
Strähle et al. Angew. Chem. Int. Ed. 1988, 27, 436
Haalan et al. Angew. Chem. Int. Ed. 1994, 33, 2443
„The Crystal Structures of … 2 Cases of Mistaken Identity“
Br!
MO-Theorie - Beispiel H2
s*
E
Ei
H
1s
H Ej
E
1s
s
S
2
1. Ordnung Störungstheorie:
S
E
(Ei = Ej)
Ei E j
2. Ordnung Störungstheorie
Li
E
H s*
s c1 2 s ,Li c2 1s ,H
*
antibindendes MO:
EN2 > EN1 c1 > c2
Li
2s
H
E
1s
-
+
Li
H
s
bindendes MO:
EN2 > EN1 c2 > c1
s c1 2 s ,Li c2 1s ,H
KZ = 1 – Modell MO-Schema: Cu-H
y
E
Cu
4p
Cu
H
H
pz
x
z
nicht-bindende WW S
Cu H
Cu H
py
px
=0
+
4s
LUMO
3d
y
Hybridisierung
dz2
=
dyz
dx2-y2
y
H
dxy
1s
+
+
H
x
x
z
dxz
x
3 dz2
4s
s*
4 pz
Cu
Hybridisierung - Konsequenzen
L
Cu-H
bessere Überlappung
S2 groß, E groß
KZ = 2 !!
H
KZ = 2
nur mit sperrigen Liganden oder mit Elementen der
Gruppen 11 und 12 (d10). Struktur: linear oder
nahezu linear
H 3N
Ag
NH3
Cl
Cu
[Ag(NH 3 )2 ]+
N
C
Hg
C
[Hg(CN) 2 ]
Cl
[CuCl 2 ]-
N
N
C
Au
C
[Au(CN)2 ]-
N
MO-Theorie AH2 (H2O)
Walsh-Diagramm AH2
VE: 2 gewinkelt LiH2+
4 linear
BeH2
"HOMO diktiert das Geschehen"
6 gewinkelt CH2 (S = 1)
8 gewinkelt OH2
Walsh Diagramm ML2-Fragment
4a1 LUMO
2b1 HOMO
S2 = 0
L
L
Pt
L
Pt
L
Chemie
Grenzorbitale = HOMO/LUMO
s*
E
LUMO = Electrophil
- MO´s Größe/Richtung
- je tieferliegend desto besser
Estab = S2/E
L
L
Pt + H2
Pt
L
L
HOMO = Nucleophil
- MO´s Größe/Richtung
- je höherliegend desto besser
s
H2
H
H
synthetischer Chemiker
Erzwingen ML2 gewinkelt ?
bidentater Ligand
L
q
M
L
q Bißwinkel (bite angle) durch Liganden steuerbar
Bite Angle
PR2
L
=
(CH2)n
L
PR2
n=1-4
PPh2
P(tBu)2
PPh2
PMe2
PR2
PPh2
PPh2
Fe
P(tBu)2
dbpm
n=1
bite angle [o]
75
PPh2
PMe2
dmpe
dppe
n=2
90
Ph2P
PPh2
dppp
n=3
90-100
PR2
> 110
PPh2
transphos 175o
Fe
PPh2
90-120
PPh2
bidentat höhere Stabilität durch Chelateffekt
[Ni(H2O)6]2+ + 6NH3
[Ni(H2O)6]2+ + 3en
[Ni(NH3)6]2+ + 6H2O KK = 2 . 109
[Ni(en)3]2+ + 6H2O
KK = 3.8.1017
Bildung des [Ni(NH3)6]2+-Komplexes Teilchenzahl bleibt gleich
Bildung des [Ni(en)3]2+-Komplexes Teilchenzahl nimmt zu S > 0
- Komplexbildung mit einem Chelatliganden führt zur Entropiezunahme!
H ist für beide Fälle ungefähr gleich.
G°=H°-TS° und G° = -RT ln KK (KK = Bildungskonstante)
(G° = 0 - TS°)
Bildung von [Ni(en)3]2+ negativeres G° grösseres KK
KZ = 3
selten, nur mit sperrigen Liganden oder Metallen der
Gruppen 11 und 12 (d10).
Strukturen: meist trigonal planar 3x 120o
T-Form möglich 2x 90o, 1x 180o (L-M-L Winkel) (d8)
P Au
P
P
P
P
d10- [Au(PPh3)3]+
Tris(triphenylphosphan)gold(I)
Cl
d10- [Au(PPh3)2Cl]
Chloro-bis(triphenylphosphan)gold(I)
d10
N
P
N
d10
Fe
Cu
S
N
S
d10
P
S
P
Fe
d5
Fe(III)
[Fe(N(Si(CH 3 )2 )3 ]
d10
Tris(hexamethyldisilylamin)eisen(III)
d10
[Cu(SP(CH 3 )3 )310
]+
d
Tris(trimethylphosphansulfid)kupfer(I)
L
M
L
M
L
L
L
L
D4h-ML4
L
L
=
+
+
2a1g
nicht-bindend
b1g
b1g
eu
eu
a1g
a1g
D4h
a1g: dz2, s b1g: dx2-y2
b2g: dxy; e1g: dxz, dyz
a2u: pz
eu: px, py
L
L
M
L
L
L
-L
L
Übergang ML4 ML 3
M
L
s*
„T-Form“ d8-ML3
Aktivierung kleiner Moleküle
p
H2, CH4
C2v
D3h
2b2
C2v
2a1
Barriere
b1
PPh3
Ph3P
Rh
e´
156o
trigonal
PPh3
1a1
b11
1b2, a2, b1
d8
d10
"T-Form"
2a1
1a1
1b2, a2
2a1
KZ = 4
- Tetraedrische Koordination bevorzugt bei d7 und d10.
Beispiele: [CoCl4]2-, [Cu(Pyridin)4]+
sterisch bevorzugt (4 x 109.50 Winkel)
- Quadratisch planare Koordination bevorzugt bei d8 (4 x 900 Winkel).
Cl
NH3
Cl
Pt
Cl
NH3
Pt
NH3
cis-[PtCl2(NH3)2]
H3N
Cl
trans-[PtCl2(NH3)2]
Isomerisierung
Cl
2-
Ni
Cl
Cl
2-
4K
Cl
Cl
Cl
Cl
Cl
Cl
M
Cl
Cl
M = Pd, Pt
Ni
Cl
2-
dx2-y2
LUMO
t2
d8-ML4
dz 2
HOMO
S=1
S=0
symmetrieverboten (S=0)
weitere Beispiele: quadratisch-planare Komplexe
Wilkinson´s Katalysator
- Hydrierung von Olefinen
- Hydroformylierung
Vaska´s Komplex
- „Drososphila für mech. Studien“
CO + C2H4 + H2 C2H5C(=O)(H)
trans-Einfluß
thermodynamische Größen, Grundzustand
Auswirkung:
- Bindungsabstand des trans-ständigen Liganden
- IR-Banden
- NMR Kopplungskonstanten
R- ~ H- PR3 > CO ~ C=C ~ Cl- ~ NH3
trans-Einfluß abnehmend
2.37 Å
2.38 Å
Cl
Cl
Pt
Et3P
Cl
IR
Cl
Cl
Cl
2.31 Å
Cl Cl
Cl
Pt
Pt
Cl Cl
Cl
L=
CO SMe2 C2H4 SEt2
(Pt-Cl) [cm-1] 322 310
309 307
Cl
Pt
L
PPh3
279
PMe3 AsMe3
275 271
trans-Einfluß
Orbitallappen ausgedehnter
S größer
LUMO
stärkere Bindung
zum trans-ständigen
Liganden
L
dx2-y2
(E ~ S2/(Ei- Ej))
Cl
H
Cl
-
Pt
Cl
starker trans-Ligand
Cl
Pt
L
Cl
schwacher trans-Ligand
trans-Effekt
Kinetik (Substutionsgeschwindigkeit)
+
Et3P
T
Cl
Pt
PEt3
k2
+
Et3P
Pt
T
N
N
PEt3
-
+ Cl
Reaktion 2. Ordung: - 1. Ordnung in [PtClT(PEt3)2]
(überwiegend) 1. Ordnung bzgl. Pyridin
T=
PEt3
H-
CH3-
C6H5-
Cl-
k2 [M-1s-1]
3.8
4.2
6.7.10-2
1.6.10-2
4.10–4
CO, CN-, C2H4 > PR3, H- > CH3- > C6H5- , NO2-, I, SCN- > Br-, Cl-> py, NH3, OH-, H2O
trans-Effekt nimmt ab
trans-Effekt >> cis-Effekt!
L
L
T M X+Y
Y+X
T M
L
L
Ligandsubstitution
L
E
T
Y
L
M
L
T
M
X
trig.-bipy.
L
L
T
M
Y
X
p-Akzeptor stabilisiert
X + Y G#
L
L
T
M
L
Y +X
Anwendung trans-Effekt Cl - > NH3
2+
NH3
+ Cl
H3N Pt NH3
NH3
+
NH3
-
- NH3 H3N Pt Cl
NH3
+ Cl
NH3
-
- NH3
Cl
Pt Cl
NH3
trans
trans Effekt: Cl- > NH3
Cl
Cl
Pt Cl
Cl
2+ NH3
- Cl-
NH3
Cl
Pt Cl
Cl
+ NH3
- Cl
-
NH3
Cl
Pt NH3
Cl
cis
d0-Übergangsmetallverbindungen
Cl
Ti
IV
Cl
VE:
O
Cl
O
Cl
O
O
O
O
Cl
p-Donor/-Bindung!
Mo
O
VII
VI
8 e-
Os
2
O
O
O
O
16 e-
Mn
VIII
O
O
O
16 e-
O
O
16 e-
Cl
Ti
Os
Cl
Cl
Cl
Ti
Cl
Cl
Cl
Abstoßung
d3-Mo(III)
starke Metall-Metallbindung
größere Substituenten ?
keine Mo-Mo-Bindung!
(R)ArN
N(R)Ar
Mo
[Mo{N(R)Ar)3}]
N(R)Ar
N(R)Ar
(R)ArN
+ N2
- N2
[Mo{N(R)Ar)3}]
N2
(R)ArN
Mo
Mo
N(R)Ar
N
N
2
N
N(R)Ar
Mo
N(R)Ar
(R)ArN
Mo
(R)ArN
N(R)Ar
N(R)Ar
N(R)Ar
N(R)Ar
bei RT!!
L
L
L
L
M
L
L
L
tetraedrisch
L
LUMO
L
L
M
L
M
L
PMe3
L
oder
L
s*
M
L
q.-pl.
Me3P
Fe
Me3P
L
c2v-ML4
L
PMe3
d8-Fe(0)
L
M
L
L
H
HOMO
sH
H
H
H
H
C
C
c2v-ML4
H2C
isolobal: Grenzorbitale: gleiche Symmetrie
& Ausdehnung
„vergleichbare Chemie“
L
L
M
L
L
trigonal-pyramidal
ML6 Oh-Symmetrie
z
L
y M
x
L
M
L
t1u
L
L
L
L
L
L
L
L
L
t1u
p z px
a1g
py
a1g
a1g
s
eg
t2g
eg
eg *
eg*
t2g
M-L-antibindend!!
nb
dz2 dx2-y2
eg
eg
t2g
dxy
dxz dyz
eg
t1u
a1g
t1u
a1g
t1u
a1g
L
L
M
L
L
L
ML6 Oh-Symmetrie - Grenzorbitale
L
eg*
LUMO
dz 2
dx2-y2
o
HOMO
t2g
dxy
dxz dyz
o: je größer je stärker Ligand-Metall-Wechselwirkung
S2
E ~
Ei - E j
spektrochemische Serie (empirisch):
E groß: CO > CN- > PR3 > CN- > NO2- > NCS- > H2O RCO2- >
OH- > F- > NO3- > Cl- > NCS- > S2- > Br- > I- (E klein)
Deutung: starke Donoren großes o
keine Erklärung für CO
Erklärung ?
ML6 Grenzorbitale
bislang: Berücksichtigung der Liganden als reine s-Donoren
Liganden: p-Akzeptor- und pDonor-Eigenschaften
CO
O2O
C
O
p*-Akzeptor
p
O C C O
p
p
s
s
C
O
O
p-Donor!
p
ss*-Donor
s s-Donor
C
8 VE
p-Akzeptor
L
L
PiPr3
L
M
L
OC
OC
L
W
CO H
H
84 pm
PiPr3
eg*
freies H2: 74 pm
+ p-Wechselwirkung
H
p*
dx2-y2
eg
t2g
dx-y
Absenkung
Rückbindung
ab d1-Konfig.
H
s*
H2 analog !
p
H
Absenkung
Hinbindung
H
s
L
M
L
L
p-Donor
L
O
L
eg*
dx2-y2
eg
dx-y
t2g
p-Donation
stabilisierend für d0-d4 Konfig
= + (Hybrid)
ML5
O
Auffüllen: Hund´sche Regel = höchste Multiplizität
eg*
eg*
E
S=3/2
S=1/2
t2g
t2g
l
2
1
0
2 S+1 = 2.3/2+1 = 4 Quartett
high spin
2 S+1 = 2.1/2+1 = 2 Dublett
NB: Atom:
4F-Zustand (L= 3 F)
ML=Sl, j=L+S ... L-S (Bahn- & und Gesamtdrehimpuls)
2H-Zustand
(L= 5 G)
L 0 1 2 3 4 5
S P D F G H
eg*
*
eg
E
S=5/2
S=1/2
t2g
t2g
Sextett
Paarbildungsenergie
Paarbildungsenergie & e-/e- Wechselwirkung:
abhängig von Hauptquantenzahl (Racah Parameter)
Trend 3d > 4d > 5d (Ausdehnung der Orbitale)
Überlapp S (M-L) größer E größer
5d Relativistik & Spin-Bahn-Kopplung
höhere Homologe low spin!
Faustregel: E > 100 kJ/mol
low spin
ML6 - Thermodynamik
L L
G
M
L
stabil
L
L
L
L
L
GR
M
L
L
L
L
Oktaeder stabiler als trigonales Prisma (GR < 0)
d3- (high spin) und d6- (low spin) Konfiguration sehr stabil:
t2g-Satz: halb- und vollbesetzt
eg*
d7
t2g
antibindendes MO besetzt
M-L Bindung geschwächt
z.B. Co(II)-Komplexe (vs Co(III))!
ML6 - Kinetik
G
L
inert
L
M
L
L
G#
L
L L
L
M
L
L
L
L
z.B. Oktaeder inert (Kinetik) G# groß isomerisiert sehr langsam!
d3- (high spin) und d6-(low spin) Konfiguration inert:
NB: G#(298K) = 100 kJ/mol t½ 1 Tag
Eyring-Gleichung:
kB T
k
e
h
G #
RT
k: Geschwindigkeitskonstante, kB: Boltzmann-Konstante, h: Planck´sches
Wirkungsquantum, T: Temperatur, R: Gaskonstante
L
L
-L
+L
L
M
L
L
L
L
L
M
L
L
L
d7 häufig KZ 5 d8 vorwiegend KZ 4 & 5
eg *
b1
a1
dx2-y2
dz2
d8 KZ 5 stabiler!
dxz dyz
t2g
dxy O -ML
h
6
e
b2
dxz dyz
dxy
c4v-ML5
cis-labilisierender Effekt
L
M
X
18 e-
-L
L'
M
L'
M
X
18 e-
X: cis-labilisierender Ligand
X = -O-R, NR2
X
18 e-
L
M
b1
dx2-y2
L
b1
L L
e
a1
dz2
e
d6
e
b2 dxz
dxz,dyz
dyz
a1
dxy
b2
p-Donation
dxy
90
100
110
120
L
KZ = 5
A
A
B
B
90°
B
B
A
90+x °
B
B
E
E
E
B
B
A
100 - 120°
A: apikal B: basal
quadratisch-pyramidal
A: apikal
E: äquatorial
trigonal-bipyramidal
3-
CN
NC
NC
Ni
CN
CN
NC
Ni
CN
3CN
CN
CN
Trigonal-bipyramidale Koordination
Beispiele: [CuCl5]3-, [MnCl5]2-, [Pt(SnCl3)5]3-, [Fe(CO)5]
[CuCl 5 ]3-
[Fe(CO) 5 ]
Berry-Pseudorotation
Bei d0-, d4-, d8- und d10-Komplexen findet man oft Berry-Pseudorotation
4
1
5
‡
2
3
4
1
5
2
3
2
1
4
3 5
4
1
5
2
3
3
1
2
4
5
trigonal bipyramidal
Stereochemie
L
L
L
cis
L
trans
L
L
L
L
L
facial
L
meridional
15 Stereoisomere!
Stereodeskriptoren:
Priorität: 1) höchste Ordnungszahl
2) höhere Massenzahl
2
1
O2N
Br
Pt
3
4
NMe2H
N
Konfigurationsindex: Priorität des Liganden in trans-Stellung
zum Liganden höchster Priorität
quadratisch-planar SP-4-3
[SP-4-3]Bromo(dimethylamine)nitrito(pyridin)platin(II)
Tetraeder T-4 R/S Nomenklatur
Stereodeskriptoren:
Priorität: 1) höchste Ordnungszahl
2) höhere Massenzahl
1
Cl
2
N
3
NH3
Pt
1
Cl
[SP-4-1]
1
Cl
2
N
1
Cl
Pt
3
NCCH3
[SP-4-3]
trans-Maximaldifferenz
Stereodeskriptoren: Oktaeder
OC-6-43
OC-6-43
3
1OH
2
NC
+
2
NH3
Co
3
NC
2
1Cl
3
2
OC
1
PPh3
Ru
4
OH2
H
2
NH3
2
2
3
NC
PPh3
PPh3
OC-6-24
OC-63-2
1OH
2
C
NH3
2
lockwise
3
OC
4
H
2
PPh3
2
PPh3
NH3
OC-63-2-C
OC-6-24-A
A
nticlockwise
NH2
NH2
H2N
NH2
Co
H2N
NH2
H2N
NH2
Co
H2N
NH2
NH2
H2N
O
NH2
Br
S
NH2
OH
Pt
NH2
HN
H2N
OH
NH
S
O
NH
HN
Co
O2N
O
O
H2N
M
NH2
links oben rechts unten
H2 N
M
NH2
links unten rechts oben
![[Mo(CO)6], [Ru(CO)5] und [RhCl(CO)(PPh3)2]](http://s1.studylibde.com/store/data/005606366_1-015cff068bd8932129b59ee7ad5cb2c5-300x300.png)