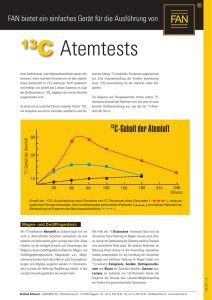
Peptidyl-cystein- und 4
Werbung

Biofocus B I O S P E K T R U M • 2. 0 1 • 7. J A H R G A N G 129 Habilitierte stellen sich vor Eine neue Familie von Flavoproteinen: Peptidyl-cystein- und 4‘-Phosphopantothenoylcystein-Decarboxylasen Thomas Kupke, Lehrstuhl für Mikrobielle Genetik, Eberhard-KarlsUniversität-Tübingen A Proteine und Peptide können auf unterschiedliche Weise posttranslational modifiziert werden, zum Beispiel glykosyliert, phosphoryliert und acyliert. Auch die von Gram-positiven Bakterien synthetisierten Lantibiotika („lanthionine-containing antibiotic peptides“, eine ausführliche Zusammenfassung über die Biosynthese und die biologischen Aktivitäten der Lantibiotika findet sich in [1]) enthalten ungewöhnliche Aminosäurereste, etwa die Thioether-aminosäure Lanthionin, D-Aminosäurereste und dehydratisierte Aminosäurereste, die durch posttranslationale Modifikationen an ribosomal synthetisierten Vorläuferpeptiden eingeführt werden. Einige Lantibiotika wie Epidermin, Mersacidin und Mutacin I/III enthalten am C-Terminus ungesättigte und decarboxylierte Thioetheraminosäurestrukturen, die als 2-Aminovinyl-D-cystein beziehungsweise 2-Aminovinyl-3-methyl-D-cystein bezeichnet werden (Abb. 1). Die Biosynthese des 2-Aminovinyl-D-cysteinrestes von Epidermin wurde näher untersucht. Es stellte sich heraus, dass der einleitende Schritt eine oxidative Decarboxylierung des C-terminalen Cysteinrestes des Vorläuferpeptids EpiA ist (Abb. 1) und durch das Flavinmononukleotid-haltige Enzym EpiD katalysiert wird [2, 3]. Durch die oxidative Decarboxylierung des Peptidyl-cysteins entsteht ein Peptidyl-aminoenthiol [3, 4, 5, 6] und durch Addition der Thiolgruppe an einen 2,3-Didehydroalaninrest dann der 2-Aminovinyl-D-cysteinrest. Das Flavoprotein EpiD besitzt keine absolute Substratspezifität, sondern kann auch andere Peptide als das natürliche Substrat EpiA oxidativ decarboxylieren. Durch die Umsetzung von synthetischen Einzelpeptiden und Peptidbibliotheken konnte das entsprechende Motiv der Peptidsubstrate ermittelt werden. Die C-terminale Aminosäure des Peptids muss ein unmodifizierter Cysteinrest sein (so wird weder ein Serin- noch ein Homocysteinrest noch ein Alkylthioetherderivat des Cysteinrestes umgesetzt), der vorletzte Aminosäurerest muss ein kleiner Aminosäurerest sein und der drittletzte ein hydrophober, voluminöser Aminosäurerest [5]. Eine Reihe von Proteinen, wie etwa die bakteriellen Dfp-Proteine, und das Halotoleranzprotein AtHAL3a von Arabidopsis thaliana weisen eine signifikante Sequenzähnlichkeit zu EpiD auf [7] und sind als Flavoproteine beschrieben worden. Eine molekulare Charakterisierung von EpiD zeigte, dass es sich bei den konservierten Aminosäuren zum Teil um Reste des aktiven Zentrums handelte. Zu nennen ist beispielsweise der konservierte Rest His67, dessen Austausch gegen Asn zum Verlust der enzymatischen Aktivität führte [7]. Charakteristisch für die genannten Proteine sind außerdem die Sequenzmotive PASANT und PXMNXXMW, die an der Kofaktorbindung beteiligt sind [7, 8]. Ausgehend von der molekularbiologischen Charakterisierung der Peptidyl-cystein Decarboxylase EpiD wurde die Frage untersucht, welches die Substrate der bakteriellen Dfp-Proteine sind. Es konnte dann gezeigt werden, dass die zu EpiD homologe aminoterminale Domäne des E. coli-Dfp-Proteins die Decarboxylierung von 4‘Phosphopantothenoylcystein zu 4‘-Phospho-pantethein katalysiert [7, Abb. 1]. Diese Reaktion war bisher einem Pyruvoyl-abhängigen Abb. 1: Die katalytische Funktion der HFCD-Proteine EpiD und Dfp. Das Flavoprotein EpiD katalysiert die oxidative Decarboxylierung von Peptidyl-cysteinen zu Peptidyl-aminoenthiolen (Aminoenthiolgruppe in rot hervorgehoben) und ist an der Biosynthese des 2-Aminovinyl-D-cysteinrestes des Lantibiotikums Epidermin beteiligt (A), während das in Bakterien ubiquitäre DfpProtein die Decarboxylierung von 4´-Phosphopantothenoylcystein zu 4´Phosphopantethein katalysiert. 4´-Phosphopantethein (in blau hervorgehoben) ist Bestandteil des Kofaktors Coenzym A (B). Enzym zugeschrieben worden, welches allerdings nie zur Homogenität gereinigt und nicht kloniert worden ist. Das Dfp-Protein katalysiert also zumindestens in vitro einen Schritt der CoenzymA-Biosynthese, indem es den reaktiven Cysteaminrest einführt. Neben der FMN-Bindung und der enzymatischen Aktivität weisen EpiD und Dfp eine weitere Gemeinsamkeit auf: Gelfiltrationsexperimente zeigten, dass diese Proteine Homododekamere bilden [7]. Vor kurzem gelang es, die Kristallstruktur von EpiD und von EpiD H67N mit gebundenem Peptidsubstrat DSYTC aufzuklären [8]. Die Kristallstrukturdaten bestätigten die dodekamere Struktur von EpiD; die zwölf Monomere sind dabei in vier tetraedrisch angeordneten Trimeren organisiert. Die Aminosäurereste Pro143-Met162, die die „substrate binding clamp“ von EpiD darstellen, und das Peptidsubstrat bilden ein dreisträngiges β-Faltblatt. Die Kristallstrukturdaten bestätigen die experimentell bestimmte Substratspezifität. So bildet der C-terminale Cysteinrest zahlreiche spezifische Kontakte zum Enzym aus, und der Tyrosinrest liegt in einer hydrophoben Tasche. Die Kristallstruktur liefert auch erste Hinweise auf den Reaktions- Biofocus 130 mechanismus von EpiD. Der Abstand der Thiolgruppe des Substrats zum N5-Atom des FMN beträgt 3,4 Å, während die Cα- und Cβ-Atome des Cysteinrestes weiter entfernt liegen. Dies deutet auf eine direkte Oxidation der Thiolgruppe und einen Thioaldehyd als Zwischenstufe der oxidativen Decarboxylierung hin. Die gebildete β-Thioketosäure decarboxyliert dann wahrscheinlich spontan zum Peptidylaminoenthiolat. Die Charakterisierung der Flavoproteine EpiD und Dfp lässt den Schluss zu, dass auch andere Proteine, etwa das homologe Flavoenzym AtHAL3a aus Arabidopsis thaliana, die Decarboxylierung von Cysteinresten katalysieren. Daher werden diese Proteine in einer Familie mit dem Namen HFCD-Proteine (homooligomere Flavin-haltige Cys-Decarboxylasen) zusammen-gefasst [7, 8]. Ziel ist es, die Substrate dieser Enzyme zu identifizieren, die Reaktionsmechanismen aufzuklären und die in-vivo- Funktionen der HFCDProteine in Beziehung zu ihrer katalytischen Aktivität zu setzen. Danksagung Mein Dank gilt Prof. Dr. Friedrich Götz, allen Kooperationspartnern und der DFG, die die vorgestellten Forschungen unterstützt hat. Literatur [1] Sahl, H.-G., and Bierbaum, G. (1998) Lantibiotics: Biosynthesis and biological activities of uniquely modified peptides from Gram-positive bacteria. Annu. Rev. Microbiol. 52: 41-79. [2] Kupke, T., S. Stevanovic, H.-G. Sahl, and Götz, F. (1992) Purification and characterization of EpiD, a flavoprotein involved in the biosynthesis of the lantibiotic epidermin. J. Bacteriol. 174: 5354-5361. [3] Kupke, T., C. Kempter, V. Gnau, G. Jung, and Götz, F. (1994) Mass spectroscopic analysis of a novel enzymatic reaction: oxidative decarboxylation of the lantibiotic precursor peptide EpiA catalyzed by the flavoprotein EpiD. J. Biol. Chem. 269: 56535659. [4] Kupke, T., C. Kempter, G. Jung, and Götz, F. (1995) Oxidative decarboxylation of peptides catalyzed by flavoprotein EpiD: determination of substrate specificity using peptide libraries and neutral loss mass spectrometry. J. Biol. Chem. 270: 11282-11289. [5] Kempter, C., T. Kupke, D. Kaiser, J. W. Metzger, and Jung, G. (1996) Thioenols from peptidyl-cysteines: oxidative decarboxylation of a 13C labeled substrate. Angew. Chem. Int. Ed. Engl. 35: 2104-2107. [6] Kupke, T., and Götz, F. (1997) The enethiolate anion reaction products of EpiD: pKa value of the enethiol side chain is lower than that of the thiol side chain of peptides. J. Biol. Chem. 272: 4759-4762. [7] Kupke, T., M. Uebele, D. Schmid, G. Jung, M. Blaesse, and Steinbacher, S. (2000) Molecular characterization of lantibiotic-synthesizing enzyme EpiD reveals a function for bacterial Dfp proteins in coenzyme A biosynthesis. J. Biol. Chem. 275: 31838-31846. [8] Blaesse, M., T. Kupke, R. Huber, and Steinbacher, S. (2000) Crystal structure of the peptidyl-cysteine decarboxylase EpiD complexed with a pentapeptide substrate. EMBO J. 19: 6299-6310. Korrespondenzadresse PD Dr. Thomas Kupke Lehrstuhl für Mikrobielle Genetik Universität Tübingen Auf der Morgenstelle 15/ Verfügungsgebäude D-72076 Tübingen Tel.: 07071-2977608 eMail: [email protected] B I O S P E K T R U M • 2. 0 1 • 7. J A H R G A N G Thomas Kupke geboren 1963, 1983-1989 Studium der Biochemie in Tübingen und München, 1989-1992 Promotion bei Prof. Dr. Friedrich Götz am Lehrstuhl für Mikrobielle Genetik in Tübingen, 1993 bis 1995 als wissenschaftlicher Angestellter und seit 1995 als wissenschaftlicher Assistent am Lehrstuhl für Mikrobielle Genetik, 1997/8 beurlaubt für einen Forschungsaufenthalt an der University of Kent (Arbeitsgruppe von Prof. Dr. R. B. Freedman, EMBO long term fellowship), 1998 Habilitation für das Fach Mikrobiologie.