Praktikum der Biochemie / Zusammenfassung
Werbung

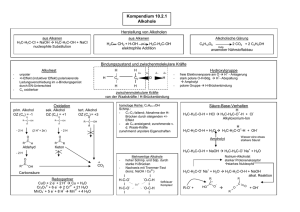
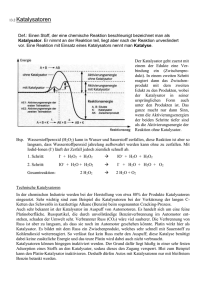
Praktikum der Biochemie / Zusammenfassung Teil 1. / Biochemie (Stoffwechsel) 1. Biochemische Methoden: - Methoden Abmessen von Flüssigkeiten - Herstellen von Lösungen definierter Zusammensetzungen - Methoden zur Fehlerabschätzung - Photometrie, Absorptionsspektrum, Absorptionskoeffizient (monochromatisches Licht mit der Intensität I0 ; Farbstoffmoleküle enthalten e- welche durch Aufnahme von Licht in e-n Zustand höherer Energie übergehen; Transmission T= I0/I wobei A = -log T = ε x d x c ) 2. Enzyme - Enzymklassen: „(Frau) Ochse trifft Herr Lüers in Lettland“ - v=(Vmax x [S] / KM + [S]) / KM = Halbsättigungskonstante; mit de Form e-r rechtwinkligen Hyperbel (y=ax/b+x) / Rkt. 1. Ordung Æ Rkt. 2. Ordnung - Lineweaver-Burk Verfahren: „Überführung“ der Hyperbel in e-e Gerade mittels der Gleichung 1/v = 1/Vmax x 1/[S] + 1/Vmax - kompetitive Hemmung: KM erhöht - nicht-kompetitive Hemmung: Vmax erniedrigt - „unkompetitive“ Hemmung: Vmax und KM erniedrigt (Rkt. Mit dem ES-Komplex) - Versuch beinhaltet die Untersuchung zur Hemmwirkung von Phosphat bzw. Fluorid auf die Phosphatase (Hydrolyse von Phosphosäuremonoestern zu Alkohol und freien organischem P) / chromogenes Substrat ist 4- Nitrophenylphosphat, dessen Anion zu e-r Gelbfärbung führt (Photometrie s.o.) 3. Aminosäuren - Grundlagen und Struktur, sowie ein enzymatischer Pyridoxalphosphattest (PALP) - Identifizierung von AS durch DC (stat. Phase: Kieselgel; Laufmittel: n-Butanol, Essigsäure, Wasser 4:1:1 / Trennung beruht auf der unterschiedlichen Polarität der Substanzen)/Rf –Werte=Wanderungsstrecke AS/Wanderungsstrecke Lösungsmittel / engl. retention-factor) Farbreaktion (AS Umsetzung mit best. Reagenzien zu Farbstoffen / SakaguchiTest: spez. Arg / Pauly-Rkt.: Tyr, His / DTNB: Cys / Cole-Hopkins-Rkt.: Try ) Aminotranferasen (AST/GOT)–Test: rel. hoher Anteil in Herz-, Skelettmuskel, sowie Leberparenchym / bei Schädigung erhöhte Aktivität in Serumproben (I): a-Ketoglutarat + Asp -> Glutamat + Oxalacetat (II): Oxalacetat + NADH+H -> Malat + NAD (Malat-DH) a-Ketoglutarat Oxalacetat Pyruvat Glyoxylat Phenylpyruvat His Glutamat Asp Alanin L-Dopa Serin Gly Phenylalanin Tryptophan Histamin GABA β-Alanin Dopamin Ethanolamin Serotonin 4. Verdauung/Stoffklassen - Versuchsbeschreibung: Untersuchung der Wirkung einiger Verdauungsenzyme im Reagenzglas (Verdauungsenzyme ausnahmslos Hydrolasen!) - pH-Optima der Proteinspaltung durch Trypsin und Pepsin (Azoalbumin=Farbstoffgekoppelt / „unverdautes“ Azoprotein als Maß für die Aktivität) - Hydrolyse von Stärke durch Speichel-Amylase (Einschlussverbindung mit Iod-Molekülen) - Spaltung der TAG durch Pankreas-Lipase (freie FS mittels Titration bestimmt / alkalisches Milieu benötigt) 5. Lipide - Versuchsbeschreibung: Extraktion von Lipiden aus biologischen Material / Trennung und qualitativer Nachweiß durch DC und Photometrie - Identifizierung DC: Platten mit Ammoniummolybdat-Phosphat-Reagenz besprüht, es bildet sich ein Phosphomolybdat-Komplex (Molybdänblau) mit den P-haltigen Verbindungen (Lecithin, Cholesterol, Triacylglycerol, Cholesterolester, Ganliosid, Cerebrosid) - Identifizierung Photometrie (Gesamt-Cholesterol im Serum): gekoppelt enymatischer Test: (I): Cholesterol + O2 -> Cholestenon + H2O2 (Cholesterol-Oxidase) (II):farblose Verb. + H2O2 -> Farbstoff (blau) + H2O (Peroxidase) 6. Organstoffwechsel/Kohlenhydrate - Versuchsbeschreibung: exemplarische Untersuchung von Stoffwechselumstellungen wichtiger Organe in Phasen der Nahrungsaufnahme und Verwertung („Resorptionsphase“ / „Postresorptionsphase“) / Verwertung von KH im Vordergrund - oraler Glucose-Toleranz-Test (OGT™): Blutglucose- und Blutlactat-Profil mit Entnahme aus der Fingerbeere und „Säure-Stopp“ mit PCA (Perchlorsäure) / nach Zentrifuge alle löslichen Bestandteile im klaren Überstand / Metabolite nun ermittelt über optisch-enzymatischem Test: Lactat: (I): L-Lactat + H2O + O2 -> Pyruvat + H2O2 (Lactat-Oxidase) (II):H2O2 + red. Chromogen -> ox. Chromogen (rosa) + H2O (Peroxidase) Glucose: (I): Glu + H2O + O2 -> Gluconsäure + H2O2 (Glucose-Oxidase) (II): H2O2 + red. Chromogen -> ox. Chromogen (rosa) + H2O (Peroxidase) - Zufuhr komplexer KH (Brötchen): s.o. - Zufuhr Protein/Fett (Eier): s.o. - Übergang zu mittelschwerer Arbeit (Treppenlauf, ca. 150 W): s.o. 7. Entgiftung/Auscheidung - Versuchsbeschreibung: Analyse verschiedener Harnbestandteile, damit Einblick in die ernährungsabhängige Regulations des N-Stoffwechsels, Abbau Purinbasen und Nierenfunktion (24h Sammelurin) - Ascorbinsäurebestimmung: Titration / Iod-Lsg. oxidiert Ascorbinsäure in äquimolarer menge quantitativ zu Dehydroascorbinsäure (Stärke-Lsg. wird als Indikator verwendet, dabei resultierender Farbumschlag am Äquivalenzpunkt aufgrund Einschlussverbindung (s.4.) mit überschüssigem Iod) (I): I2 + Ascorbinsäure -> 2 I- + Dehydroascorbinsäure + 2 H+ - Bestimmung der Gesamt-N-Ausscheidung - (a) Harnstoffbestimmung: optischer enzymatischer Test (I): NH2-CO-NH2 + 2H2O + H+ -> 2NH3+ + HCO3- (Urease*) (II):NH4+ + -OOC-CO-(CH2)2 -> -OOC-HCNH3+-(CH2)2-COO- (Glu-DH) +NADH+H+ +NAD+ + H2O * evtl. Kopplung mit e-r Titration der Base NH3+ - (b) Kreatininbestimmung : Jaffe-Rkt. (+ Pikrinsäure, es entsteht ein rotoranger Farbkomplex) - (c) Harnsäurebestimmung: gekoppelt enzymatischer Test. (I): Harnsäure in Allatoin (Uricase) + H2O2 (II): H2O2 (Peroxidase) zu Chinonimin-Farbstoff - Urinteststreifen: Multistix 10 SG™ / Glu, Bilirubin, Ketonkörper, spez. Gewicht, Erythrozyten/Hämoglobin, pH, Eiweiß, Urobilinogen, Nitrit, Leukozyten Teil 2. / Molekularbiologie 1. Mutation - Versuchsbeschreibung: Identifikation e-r Punktmutation in e-r cDNA Sequenz des humanen Cyclins (= pos. regulatorische Untereinheit von CDKs / s.a. mitose promovierender Faktor (MPF) / Progression der Mitose) - Restriktionsverdau: Linearisierung der Plasmid-DNA mit EcoRI / Fragmentierung mit NotI - Agarose-Gelelektrophorese zur Auftrennung der DNA-Fragmente und Detektion der DNA-Banden unter UV-Licht 2. P C R - Versuchsbeschreibung: Vorstellung der PCR - a) PCR: Nachweiß der Expression amplifizierter myc-Gene in zwei Bronchialkarzinomlinien (Anzahl der PCR-Produkte definierter Länge nimmt ab dem3. Zyklus exponentiell zu) b) anschl. Gelelektrophorese: die Intensität der Banden korreliert mit der Menge des jeweiligen DNA-Fragmentes (abhängig von der ursprünglichen Anzahl der coder myc- cDNA Moleküle in der Ausgangsprobe) 3. In vitro-Translation - Versuchsbeschreibung: Untersuchung der Expression eines Proteins in vitro und Wirkung von Antibiotika (verwendetes Enzym: β-Galaktosidase) - Plasmid pBac: Gen unter Kontrolle e-s bakteriellen Promoters - Plasmid pT7: Gen unter Kontrolle e-s viralen Promoters - in beiden Versuchsteilen erfolgt anschl. E-e photometrische Bestimmung des Chromophor Nitrophenol (durch β-Galaktosidase hydrolytisch aus ONPG freigesetzt) 4. Protein-Expression - Versuchsbeschreibung: Expression e-s humanen Serumproteins CBG in E. coli und Nachweiß von Einfluss der CBG-Synthese auf die Expression (beeinflusst sie negativ, weil Energie-Mangel aufgrund der hohen Synthese) - Vorbereitung (nicht teil des Versuchs): Klonierung (cDNA), Einbau in e-n Expressionsvektor, Tranformation des Vektors in E. coli - Induktion der Serumproteinexpression in e-r Bakterienkultur (IPTG) - Überprüfung der Expression mittels SDS-Gelelektrophorese bzw. Überprüfung des exp. Wachstums durch Aufnahme e-r Wachstumskurve Regulation der Transkription: Prokaryonten - Promoter-Operator-Gen - neg. Kontrolle: Repressorprotein auf O / Ablösung bei Bindung e-s Induktors / Polymerase an P - pos. Kontrolle: Aktivatorprotein an O Eukaryonten - „house keeping genes“: Promotor sequenz und Transkriptionsfaktor der RNA-Polymerase - Enhancer: spez. Basensequenz an die Liganden binden = cisaktivierende Elemente z.B. lac-Operon/-Repressor (Induktion z.B. Hormonrezeptorkomplex/ Zink durch Allactose, 1-Isopropyl-β-D-Thio- fingerproteine galctopyranosid IPTG - Achtung: die Expression e-s eukaryontischen Proteins in E. coli benötigt bakteriellen Promoter und eine Erkennungssequenz für die Ribosomen (eukaryontischen Genen fehlen diese für Bakterien typischen Regulationssequenzen = Promotoren und die Erkennungssequenz = „Shine-Dalgarno-Sequenz“) 5. Hormone - Analyse der Inhaltsstoffe Antibaby-Pille mittels DC: Ergebnisermittlung mit Hilfe von Referenzbanden - Nachweiß von Cortisol mittels ELISA: ELISA arbeitet nach dem Kompetitionsprinzip, d.h. zu bestimmende Menge an Corstisol in der Probe und definierte Menge an Cortisol das mit Peroxidase gekoppelt ist (Enzymkonjugat) konkurrieren um die Bindestellen e-s fixierten polyklonalen anti-Cortisol Antiserums / Nachweiß des umgesetzten chromogenen Subtrats per Photometrie („je geringer der Farbumschlag und damit die Absorption bei 450 nm, desto höher die Cortisolkonzentration der Probe“, also umgekehrt proportional)