Gelenkte in vitro-Evolution enantioselektiver Enzyme
Werbung

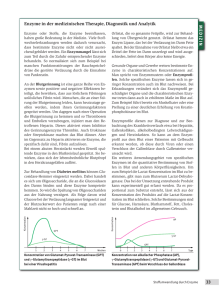
681 Gelenkte in vitro-Evolution enantioselektiver Enzyme Eine Automationslösung für Hochdurchsatz-Screening von Enzymbibliotheken Manfred T. Reetz und Marcus Hermes Max-Planck-Institut für Kohlenforschung, Mülheim an der Ruhr Die linke und rechte Hand: Chiralität und Enantiomere In der Natur gibt es eine Vielzahl von spiegelbildlich aufgebauten Molekülen, so genannte Enantiomere, die wie linke und rechte Hand räumlich nicht zur Deckung gebracht werden können – man nennt sie daher auch chirale Moleküle. Auch viele synthetische Wirkstoffe kommen als Enantiomere vor, wobei meist nur eines der Enantiomere die gewünschte biologische Wirksamkeit zeigt, während das spiegelbildliche Gegenstück oft unwirksam ist oder gar negative Wirkungen zeigt. Das vielleicht bekannteste Beispiel hierfür ist das Schlafmittel R-Thalidomid und das teratogene, starke Missbildungen bei Neugeborenen hervorrufende S-Thalidomid. In dem berüchtigten Arzneimittel Contergan war ein Racemat enthalten, eine Mischung aus beiden Enantiomeren. Bei der chemischen Synthese von Molekülen entstehen solche Race- mate, schwer trennbare Enantiomerengemische, deren Auftrennung einen sehr hohen Aufwand erfordert. Die Entwicklung von Enzymen, die eine enantioselektive Stoffumwandlung ermöglichen, ist daher hochinteressant für die Wirkstoffentwicklung. Das Prinzip der gelenkten in vitro-Evolution Vor einigen Jahren wurde am Max-Planck-Institut für Kohlenforschung ein grundsätzlich neuer Ansatz zur Entwicklung enantioselektiver Enzyme erarbeitet, der auf der „Evolution im Reagenzglas“ beruht. Das Verfahren besteht aus einer Kombination molekularbiologischer Methoden für Zufallsmutagenese und Genexpression mit einem effizienten Screening-System, das es ermöglicht, tausende von enantioselektiven Mutanten sehr schnell zu durchsuchen. Dabei wird zunächst die für eine Stoffumwandlung am bes- Abb. 1: Schema der gelenkten in vitro-Evolution enantioselektiver Enzyme Industrie-Applikationen 682 Der Arbeitsablauf Abb. 2: Marcus Hermes vor Gerät 1. Hier wird die Analytik-Vorbereitung durchgeführt. Tecan Genesis Workstation 150/8 mit Robotic Manipulator Arm (rechts), Inkubator links hinten, Tecan Multipipettierer TeMO 96 (Mitte) und Tecan Mikroplatten-Karussell (ganz rechts). Eine Lipase aus Pseudomonas aeruginosa und die Epoxidhydrolase aus Aspergillus niger wurden im experimentellen Ansatz als Beispielenzyme verwendet. Der Arbeitsablauf besteht aus zwei grundlegenden Verfahren: 1. Zufallsmutagenese und Genexpression 2. Hitpicking und Hochdurchsatz-Screening auf Enantioselektivität Die Automationslösung ten geeignete Enzym-Mutante identifiziert und dann das entsprechende Enzym-codierende Gen erneut der Mutagenese und einem anschließenden Screening unterworfen, ein Vorgang, der beliebig oft wiederholt werden kann (s. Abb. 1). Die Mutagenesen liefern jeweils eine Bibliothek mit einer großen Zahl von Genen, die dann in E. coli eingeschleust werden. Die Bakterien produzieren tausende mutierter Enzyme mit randomisiert ausgetauschten Aminosäuren, die im Hochdurchsatz-Screening auf Enantioselektivität getestet werden. Die zyklische Wiederholung des Vorgangs erzeugt den „evolutionären Druck“, der schließlich zu der Gen-EnzymKombination mit der gewünschten Enantioselektivität führt. Da hierfür tausende von Mutanten untersucht werden müssen, wurde eine Automationslösung auf der Basis von Tecan-Laborrobotern etabliert. Abb. 3: Gerät 2. Hier wird das Hitpicking von aktiven Klonen und das Screening auf Enantioselektivität durchgeführt. Tecan Genesis Workstation 150/8 mit 8-fachem Liquid Handling Arm (links), Robotic Manipulator Arm (rechts hinten, verdeckt), zwei Inkubatoren (links hinten), Tecan Multipipettierer TeMO 96 (Mitte), Tecan Sunrise Absorptionsreader (rechts daneben) und Tecan Mikroplatten-Karussell (ganz rechts). Gerät 1: Hier wird die Probenvorbereitung für das Screening durchgeführt. Dies geschieht auf einer Tecan Genesis Workstation 150/8 mit Robotic Manipulator Arm (RoMa), mit Inkubator, Tecan Te-MO 96 (96-Kanal Multipipettierer) und einem Tecan Mikroplatten-Karussell (s. Abb. 2). Gerät 2: Auf diesem Gerät wird das Hitpicking von aktiven Klonen und das Screening auf Enantioselektivität durchgeführt. Dies geschieht auf einer Tecan Genesis Workstation 150 mit 8-fachem Liquid Handling Arm, RoMa, zwei Inkubatoren, Te-MO 96, Mikroplatten-Karussell und einem Tecan Sunrise Absorptionsreader (s. Abb. 3). Die Anlage wird von Tecans FACTS-Software gesteuert. Der 1. Schritt: Zufallsmutagenese und Genexpression Die error-prone PCR (epPCR) erlaubt es, bei der DNA-Replikation gezielt „Fehler“ einzubauen, eine einfache Möglichkeit, Gen-Mutanten zu erzeugen, wobei die Mutationsrate empirisch eingestellt werden kann. Auch mit der Sättigungsmutagenese, bei der gezielt ausgesuchte Positionen im Enzym gesättigt werden, und mit DNAShuffling, einer rekombinanten Methode, lassen sich Genmanipulationen durchführen. Die mutierten Gene werden in E. coli-Expressionsvektoren kloniert, die Kolonien mittels eines Kolonie-Pickers (z. B. Genetix) aus- gesucht und in 96-Deep-well Mikroplatten angezogen. Nach der Zentrifugation kommen die Platten mit den E. coli-Pellets auf die Tecan Genesis Workstation, wo sie mittels des Te-MO 96 Multipipettierers resuspendiert und dann in 96-well Mikroplatten auf Enzymaktivität gescreent werden. Für die Epoxidhydrolase werden dabei verschiedene Substrate getestet, deren Umsatz sich mit einfachen, qualitativ auswertbaren Assays nachweisen lässt. Dazu eignen sich z. B. die in der Literatur beschriebenen PNP- und Adrenalin-Tests. Aktive Klone ergeben bei PNP eine gelbe Färbung, beim Adrenalintest wird von aktiven Klonen der rote Farbstoff abgebaut. Die Farbveränderung wird im Sunrise Absorptionsreader gemessen und die Daten an die SelActive-Software übergeben, welche einen definierten Cut-off Wert anlegt und die Hits in eine Pipettierliste für das selektive Zusammenfügen von aktiven Klonen auf neue Platten überführt (Hitpicking). Von den gescreenten Klonen sind 5–35 % aktiv. Damit kann aber noch keine Aussage über die Enantioselektivität der Klone gemacht werden, sondern es sind lediglich aktive Klone selektiert worden. Der 2. Schritt: Hitpicking und Hochdurchsatz-Screening auf Enantioselektivität Das Screening auf Enantioselektivität der Enzyme erfolgt mittels Massenspektrometrie (MS). Hierzu wird eines der beiden Enantiomere eines Enzymsubstrates in einem Racemat (50 % R- und 50 % L-Enantiomere) deuteriert. Diese Mischung wird zu den Bakterienlösungen gegeben und über Nacht inkubiert. Zur Vorbereitung der MS-Analytik werden die Deep-well Platten auf das zweite Tecan-Gerät gebracht. Eine Probe der Bakterienkultur wird in eine Glas-Mikroplatte pipettiert und mit Essigester versetzt. Mittels Flüssig-FlüssigExtraktion werden die organischen Moleküle in die Essiges- terphase überführt. Die übrigen Bestandteile verbleiben in der wässrigen Phase. Die Enzymsubstrate werden dann mittels MS vermessen. Da eines der Substrat-Enantiomere deuteriert wurde, können R- und L-Enantiomere des Substrats aufgrund der unterschiedlichen Massen unterschieden werden. Bis zu 10.000 Proben können an einem Tag geprüft werden. Ein Verhältnis von 50 % Rund 50 % L-Enantiomeren bedeutet, dass das vom Bakterienklon exprimierte Enzym keinerlei Enantioselektivität zeigt. Wird ein Überschuss eines der beiden Substrat-Enantiomere gemessen, zeigt dies, dass eines der Substrate besser als das andere vom exprimierten Enzym umgesetzt worden ist. Solche „verbesserten“ Klone werden dann weiteren MutageneseZyklen unterworfen, um sie weiter zu verbessern. Die automatisierte „evolutionäre in vitroEnzymoptimierung“ Die Stärke des Verfahrens liegt darin, dass weder die Struktur des Enzyms, noch der Mechanismus der Enzymkatalyse bekannt sein muss. Das Verfahren ermöglicht eine automatisierte „Enzymoptimierung“, wobei versucht wird, die natürliche Evolution im Reagenzglas nachzuahmen. Durch Tecans Automationslösung konnte das Verfahren sehr effizient in die Praxis umgesetzt werden. Korrespondenzadresse: Prof. Dr. Manfred T. Reetz Max-Planck-Institut für Kohlenforschung Abteilung Synthetische Organische Chemie Kaiser-Wilhelm-Platz 1 D-45470 Mülheim an der Ruhr Tel.: 0208-306 2000 Fax: 0208-306 2985 [email protected] BIOspektrum · 5/05 · 11. Jahrgang