Genetik Bachelor 3. Semester 2010
Werbung

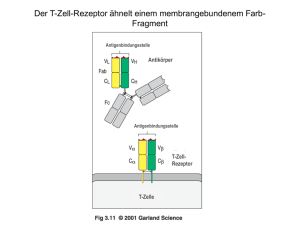
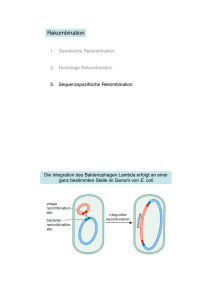
Genetik im Bachelorstudiengang, 3. Semester E. Buchner Anmerkung zur Klausur: Da dies die letzte Genetik-Lehrveranstaltung im Bachelor-Studiengang ist, wird erwartet, dass der Stoff der Vorlesung anhand der Kapitel „Genetik“ in den Lehrbüchern Campbell oder Purves vertieft und erweitert wird. Die Klausur geht also über den Inhalt dieser Kapitel und die Ergänzungen, die wir in der Vorlesung besprechen. Empfehlung: Fertigen Sie parallel zur Vorlesung von Tag zu Tag eine 1-2 seitige Zusammenfassung des Stoffs an, die Sie dann zur Vorbereitung auf die Klausur im Januar verwenden können. Hinweis: Die Wiederholungsfolien aus dem 2. Semester sind im Skript vom 2. Semester zugänglich (aus Platzgründen entfernt). Genregulation Chromatin-Remodeling Heterochromatin Euchromatin Transkriptionskontrolle Capping, poly-A, alternatives Spleißen Translationskontrolle Posttranslationale Modifikation Proteinstabilität Chromatin Umorganisation (remodeling) Remodeling Faktoren können sequenzspezifisch Nukleosomen verdrängen Genregulation durch Histonmodifikation (ATP-abhängig): Transkriptionsaktivatorkomplexe besitzen oft HAT Aktivität Transkriptionsrepressorkomplexe besitzen oft HDAC Aktivität (Deacetylase) Sequenz-spezifische Modifikation: Rekrutierung der HAT durch DNA-Bindungsproteine Histon-Modifikation eigener „Code“ (Histon-Code), definiert Art des Chromatins. H3 H4 Lys-4 Lys-9 Methylierung Methylierung Lys-9 Ser-10 Lys-14 Lys-79 Acetylierung Phosphorylierung Acetylierung Verhindert Methylierung an Lys-9 Methylierung Telomerfunktion Arg-3 Lys-5 Lys-12 Lys-16 Methylierung Acetylierung Acetylierung Acetylierung Chromatin Kondensierung Voraussetzung für DNA-Methylierung Nucleosom Assemblierung X-Chromosom Aktivierung bei Drosophila Nukleosomenfreie Bereiche der DNA Diese Bereiche von ~200bp sind frei von Nucleosomen und daher für mehrere Enzyme zugänglich. Wie entstehen nukleosomenfreie Bereiche? Huhn--Globin Gen in Plasmid kloniert, Zugabe von Histonen und Extrakt von Erythrozyten-Kernen nukleosomenfreie Bereich entsteht nur wenn Extrakt vor Histonen zugegeben wird. DNA –Bindungsproteine (z. B. Transkriptionsfaktoren) verhindern die Nucleosomenbildung an nukleosomenfreien Bereichen Wo? Promotor, Enhancer, Locus Control Region, Origin, Centromer Modifikation der DNA 2-7 % der C Reste tierischer DNA (Ausnahme: Dipteren) sind methyliert. Methylierungsgrad von Spezies zu Spezies unterschiedlich. Satelliten-DNA oft stark methyliert. Säuger: 70% der CpG-Dinukleotide sind methyliert. Meist 5' mCG 3' 3' GCm 5' Paare: CpG-Inseln, vor aktiven Genen weniger methyliert Methylierung einiger Stellen expressionsabhängig: methyliert in inaktiven Genen, nicht methyliert in aktiven Genen. Analyse der Methylierung: Bisulfit-Behandlung und Sequenzierung: 1) Bisulfit Behandlung CH3 5´…CGTTACTGCCGA…3´ desamination HSO3- CH3 BS Behandlung 5´…CGTTAUTGUUGA…3´ PCR 2) PCR 3) Sequenzierung 5´…CGTTAT TGT TGA…3´ Wie wird Methylierungszustand bei Replikation weitergegeben? (Abb.) Änderung des Methylierungszustands: Methyl Transferasen, Demethylasen Haushaltungsgene: schon demethyliert in Gameten. Luxusgene: in Gameten methyliert, in dem Gewebe, in dem sie exprimiert werden, teilweise demethyliert. Demethylierung kann Genexpression auslösen. Beispiel: Cytidin kann durch 5-Azacytidin ersetzt werden. 5-Azacytidin wird nicht methyliert z.B. Bildung von Muskelzellen aus nichtmuskulären Vorläuferzellen; Gene auf dem inaktiven X-Chromosom werden aktiviert. Inaktivierung beruht u.a. auf Methylierung. Kein universeller Mechanismus: in Dipteren wird DNA nicht methyliert. Epigenetisch inaktiv Epigenetisch aktiv Zusammenfassung der Regulation auf Chromatin-DNA-Ebene • 3 Indikatoren für aktive Gene: • Hyperacetylierte Histone im Genbereich • Nukleosomenfreie Bereiche (Promotor, Enhancer) • Untermethylierung der DNA im Promotorbereich Transkriptionsregulation (Transkriptionsfaktoren) Regelung der Transkriptionsfaktoren durch: Synthese Phosphorylierung Dimerbildung Liganden, z.B. Steroidhormone Peptidhormone, Wachstumsfaktoren: Membranrezeptoren und intrazelluläre Signalwege Phosphorylierung von Transkriptionsfaktoren Aktivierter Transkriptionsfaktor a) Promotor Transkriptions-Startpunkt: durch TATA-Box Bindung des Enzymkomplexes Transkriptions-Rate: durch stromauf-Elemente geregelt, kein Einfluß auf Startpunkt Aktivierung der Polymerase Beispiele: Thymidin-Kinase-Gen Module (GC-Box, CAAT-Box) im Promotor binden Transkriptionsfaktoren Kein Modul in allen Promotoren. Wenn TATA-Box fehlt mehrere Startpunkte. b) Terminator: - Haarnadelstrukturen in RNA - Interaktion mit poly-A-Schwanz (Prokaryonten) (Eukaryonten) Wenig bekannt bei Eukaryonten, da 3‘-Ende sehr instabil, prozessiert. Termination bis zu >1000 bp stromab vom 3‘-Ende der reifen mRNA c) Enhancer: ähnliche Funktion wie Promotor, aber: - Position bzgl. Startpunkt kann stark variieren - Orientierung bzgl. Transkriptionsrichtung unwichtig 2 Arten von Enhancer: generelle Enhancer - zellspezifische Enhancer Wirkungsmechanismus eines Enhancers: Bindung eines Transkriptionsfaktors, Schleifenbildung, Interaktion des TF mit Transkriptionskomplex, Aktivierung d) Silencer: ähnliche Struktur wie Enhancer, jedoch Inhibitor der Transkription, unabhängig von Position und Orientierung Beispiel: Hühner Lysozym-Gen E = Enhancer S = Silencer HRE = Hormone responsive element Eileiter: Expr. Induziert durch Steroide E S HRE Makrophagen: Starke Expr. E MakrophagenVorläufer: S HRE Schwache Expr. E Andere Zellen: S HRE Keine Expr. E e) LCR: LCR: Bereichsregulation und Domänenbegrenzung Menschliches β-Globin-Gen in Maus transfiziert Expression < 10% Identifikation weiterer regulatorischer Regionen „Locus Control Region“ = LCR 5‘ HSS (= LCR) 3‘ HSS G A ß1 ß 20 kb f) Isolatoren: blockieren Wirkung von Enhancer, begrenzen Heterochromatisierung Beispiel Drosophila: Aktives Gen zwischen SCS und SCS‘ Gen auch in Heterochromatin aktiv. 87A6 87A7 87A8 5 kb SCS HSS resistent hsp70 hsp70 TF BP E I Pol-II P Tr-Start SCS‘ SCS = specialized chromatin structure = Isolator HSS Isolator zwischen Enhancer und Promotor eines Gens: Gen inaktiv. Fehlt Bindungsprotein (BP) Gen wieder aktiv Weitere Domänenbegrenzungen: MAR = Matrix attachment region: fixiert Chromatin-Domäne an der Kernmatrix SAR = Scaffold attachment region: fixiert Chromatin-Domäne am Chromosomengerüst ?MAR = SAR? Gelegenheit für veränderte Bindung von Transkriptionsfaktoren (= veränderte Expression) bei jeder Replikation Abschalten von Genen z.B. durch Verdünnung von Bindungsproteinen oder Inaktivierung von Transkriptionsfaktoren. Man unterscheidet: Transkribierbarkeit = Determinationsereignis Transkription = Differenzierungsereignis Transkriptions-Faktoren: (definiert durch in-vitro System oder Sequenzhomologie). 3 Gruppen von Transkriptionsfaktoren: 1.) Untereinheiten der RNA-Polymerase, notwendig für alle Promotoren 2.) Proteine, die nach DNA Bindung an Polymerase-DNA-komplex binden, notwendig für alle Promotoren 3.) Proteine, die an Sequenzen von Promotoren binden, nicht an allen Promotoren aktiv Genregulation Transkriptionsfaktoren binden meist an kurze DNA-Sequenz = „Response Element“ Transkriptionskomplex = Transkriptionsfaktoren + Polymerase + DNA Domänenstruktur der Transkriptionsfaktoren Meist 3 Domänen: 1) DNA-Bindungsdomäne 2) Protein-Protein-Interaktionsdomäne (Aktivierungsdomäne) 3) Regulationsdomäne Besitzt ein Transkriptionsfaktor nur DNA-Bindungsdomäne, so wirkt er meist als Repressor Transkriptionsfaktoren ohne DNA-Bindungsdomäne wirken durch Interaktion mit DNA-bindenden Faktoren Ein Transkriptionsfaktor kann an verschiedene Response-Elemente (DNA-Boxen) binden, ein Response Element kann mehrere Transkriptionsfaktoren binden Peptidmotive, die charakteristisch für Transkriptionsfaktoren sind: 1) DNA-Bindungsmotive: - Zink-Finger Motiv (z.B. TFIIIA, Steroid-Rezeptor-Familie) - Helix-Turn-Helix Motiv (z.B. Phagen-Repressor, Homeo-Domäne) - „basische Region“ (z.B. bei Helix-Loop-Helix-, Leucin-Zipper-Proteinen) 2) Protein-Protein-Interaktionsmotive: (DNA-Bindung: oft „basische Region“) - Helix-Loop-Helix Motiv - Leucin-Zipper Motiv - Saure Tropfen Motiv 1) DNA-Bindungsdomänen: - Zink-Finger-Domänen: Konsensus: -Cys-X2-4-Cys-X3-Phe-X5-Leu-X2-His-X3-HisBeispiele: TFIIIA (9 Zn-Finger als Tandem Repeats) SP1 (3 Zn-Finger, Bindung an GC-Box) ADR1 (2 Zn-Finger, aktiviert ADH2-Gen in Drosophila) TDF (13 Zn-Finger, männl. Geschlechtsdetermin. in Säugern) Krüppel (5 Zn-Finger, Segmentierung in Drosophila) Hunchback (4 + 2 Zn-Finger, Segmentierung in Drosophila). Konsensus: Beispiele: -Cys-X2-Cys-X13-Cys-X2-CysSteroid Rezeptoren: Bindungsproteine für Steroid-Hormone: Corticoid-Hormone (Cortison, Aldosteron) (> 30) Nebenniere, Funktion: Homöostase Steroid-Hormone (Östrogen, Testosteron) Geschlechtsentwicklung Thyroid-Hormon (Triiodothyronin, T3) Stoffwechselrate Vitamin D (Vitamin D3) Knochenentwicklung, Ca2+-Regulation Retin-Säure (Vitamin A-Säure) Morphogen Steroide sind fettlöslich Diffusion durch Zellmembran Steroid-Bindung an Rezeptor Wanderung in Kern, Bindung an DNA Rezeptoren: Homologie sowohl in Hormon- als auch in DNA-Bindungsregionen Super-Familie, meist Dimere oder Tetramere Glucocorticoid-Rezeptor (GR, 2 Zn-Finger, Bindung an GRE) Östrogen (engl. estrogen)-Rezeptor (ER, 2 Zn-Finger, Bindung an ERE) Gal4-Regulator (Gal4, Bindung an UAS in Hefe, Regelung durch Gal80-Galactose) Finger-Region legt DNA-Bindungsspezifität fest, aber: Zn-Finger nicht Beweis für DNA-Bindung. Response Elemente: erkannte DNA-Sequenz, meist kurzes Palindrom - Helix-Turn-Helix-Domänen: Bindungsdetails an Phagenrepressor-Operator-Bindung analysiert. Beispiel aus Eukayonten: Homeo-Domäne (60 Aminosäuren), wird in allen Eukaryonten gefunden, wurde in Produkten von homeotischen Genen in Drosophila entdeckt (frühe Entwicklungsgene, >20 in Drosophila, z.B. Antennapedia, >10 in Säugern). Oft kombiniert mit Pou-Domäne = 75 AS 2) Dimerisierungsdomänen: - Helix-loop-helix-Domänen: Zahlreiche DNA-Bindungsproteine besitzen HLHDomänen, die sie zur Dimerisierung befähigen. HLH-Domäne: 40-50 AS bilden zwei amphiphatische Helices verbunden durch Schleife. Benachbarter stark basischer Abschnitt ist für DNA-Bindung verantwortlich --> basische HLH (bHLH)-Proteine. HLH-Proteine bilden Homo- oder Heterodimere. Bindung eines nicht-basischen HLH-Proteins an bHLH-Protein --> Inhibition der DNA-Bindung = TF-Inaktivierung. Beispiele: E12, E47 binden an Immunglobulin-Enhancer MyoD (Inhibitor Id), Myogenin, Myf-5: TFs zur Steuerung der Myogenese Myc-Proteine: Wachstumsregulation -Leucin-Zipper: Dimer-Bildung: Amphiphatische -Helix: hydrophobe AS (Leucin) auf einer Seite, geladene Gruppen (hydrophil) auf der anderen. Alle 7 AS Leucine umeinandergewundene -Helices, 3.5 AS/Windung Beispiele: jun, fos, immediate-early genes, Protoonkogene 5 Leucin-Repeats, dann stark basische Region (DNA-Bindung) AP1-Transkriptionsfaktor, Dimer aus jun und fos jun-Familie, Homodimere oder Heterodimere fos-Familie, FRA (fos-related antigens), nur Heterodimere Beispiele für Regulation: Hitzeschock (HS) -Gene von Drosophila HS-Gene werden durch HS aktiviert, enthalten Consensus-Sequenzen bei -15bp, an die HSTF (Hitzeschock-Transkriptionsfaktor) bindet = HS-Response Element (HSRE). ~ 20 HS Gene. Glucocorticoid-Rezeptor (GR) bindet an Glucocorticoid-Response Element (GRE) Metallothionein Promotor: ~ 12 bp Sequenz (MRE) verantwortlich für Aktivierung durch noch unbekannten Transkriptionsfaktor, der durch MetallIonen (z.B. Zn, Cd) aktiviert wird. (BLE = basal level element, TRE = Phorbol-Ester (TPA) – RE C) Genregulation auf der Ebene der RNA Capping: Stabilität der mRNA, Bindung ans Ribosom Polyadenylierung: Stabilität, Transport aus Kern ins Zytoplasma Spleißen: Ein Gen, viele Protein-Isoformen Spleißen Alternatives Spleißen Regelung durch RNA-Bindungsproteine Spleißen: Beispiele für Exon-Intron Struktur: a) Globin- Gene Exon1 Intron1 Exon2 Intron2 Exon3 Muster konstant in allen Säurern, Vögeln, Fröschen, Details unterschiedlich b) Dihydrofolsäure Reduktase: cDNA 2000 bp, Strukturgen 31 kb Mensch Maus Hamster a)pro-2- Collagen-Gen (Hühnchen) über 50 Exons, zus. 5000 bp, 40 kb Strukturgen. b)Drosophila-rRNAs: mit Introns = inaktiv, ohne Introns = aktiv. Sequenzanalyse Intron- Exon- Verbindungsstelle: 5' Exon Intron ...A64 G73 G100 T100 A62 A68 G84 T63 .... Exon 3' 6Py74-87 N C65 A100 G100 N ... GT-AG- Regel, bei allen höheren Eukaryonten Spleißmechanismus hoch konserviert aber: GT-AG reicht nicht aus. Umgebung muss auch stimmen. Erkennungssequenzen für RNA-bindende Proteine bei alternativem Spleißen. Alternatives Spleißen, alternativer Transkriptionsstart, alternative Polyadenylierung: Erzeugt unterschiedliche Protein-Isoformen entweder in derselben Zelle oder zellspezifisch, Zelltyp A = Isoform 1, Zelltyp B = Isoform 2 etc. Mechanismus für alternatives Spleißen: RNA-Bindungsproteine binden sequenzspezifisch an Exon-Intron-Grenze und blockieren Erkennung durch Spleißosom. Regulatorische Proteine binden an „splice enhancers“ Beispiel: a) Maus Amylase Gen: Speicheldrüsen- Leber-Promotor Exon S Exon L Exon 2 hnRNA aus Speicheldrüse: Translationsstart mRNA aus Speicheldrüse: Exons S 2 3 hnRNA aus Leber: Translationsstart mRNA aus Leber: Exons L 2 3 Exon 3 Extremes Beispiel für alternatives Spleißen: Drosophila Homolog zum „Down Syndrom Cell Adhesion Molecule“ DSCAM 38.016 verschiedene Spleißvarianten möglich, vermutlich wichtig für korrekte Konnektivität der Neurone im Nervensystem. Andere Möglichkeit zur Erzeugung von extrem vielen Proteinvarianten: „Somatische Rekombination“ im Immunsystem: Veränderung der DNA vor der Transkription B-Zell-Rezeptor, T-Zell-Rezeptor (s. u.) Spleißen: 3 verschiedene Mechanismen - prä-mRNA (heterogene nukleäre RNA, hnRNA) von Eukaryonten: Spleißosom - Hefe t-RNA: Enzyme ähnlich Prozessierungsnuklease, Konformation der RNA ist kritisch - Ciliaten rRNA, Klasse I und II mtRNA (Hefe): sind autonom für Selbst-Spleißen (autokatalytisch) Bedeutung der Entdeckung von selbstspleißenden RNAs für Fragen der Evolution: Nukleinsäuren älter als Proteine Anfangs selbstreplizierende RNAs mit Enzymfunktion = Ribozym, später Proteine zur Stabilisierung, dann Übernahme der Enzymfunktion durch Proteine Ribosomen zunächst katalytische RNA, dann mit Proteinen Peptidbindung zwischen Aminosäuren wird im Ribosom durch rRNA katalysiert. RNA-Editierung (3 Beispiele) 1.) Eukaryonten: Adenosin deaminase acting on RNA (ADAR) Führt zu A I Austausch, wird in cDNA und Translation als G gelesen. 2.) Säugerzellen: Substitution einer Base Apolipoprotein-B-Gen, 29 Exons in Leber: Protein von 512 kDa in Darm: C2153 gegen U ausgetauscht CAA (Glutamin) UAA (Stop) Protein von 250 kDa 3.) Mitochondriale Gene von Trypanosomen: Addition und Deletion mehrerer Basen + Rasterschub Nonsense mediated RNA degradation Wenn Stopp codon vor letztem Intron Abbau der RNA Regulationsmöglichkeit auf Ebene der Translation: Translation ist in vitro Translation weder spezies- noch gewebespezifisch. Trotzdem regelbar: “gespeicherte“ mRNPs werden durch miRNAs während Entwicklung an Translation gehindert. Micro-RNAs: Nicht-kodierende Gene mit Sequenzen aus 3‘UTR der Ziel-RNA wird transkribiert, Sekundärstruktur mit Doppelstrang-RNA, wird von Enzym Dicer in kleine dsRNAs zerhackt miRNAs miRNAs RNA-induced silencing complex RISC spaltet Doppelstrang, Strang mit Sequenz komplementär zu 3‘UTR bindet an Ziel-RNA und blockiert Translation Blockade der Translation Gefaltetes Transkript eines miRNA-Gens D) Genregulation auf der Ebene der Proteine Mehrere Polypeptide werden aus einem “Polyprotein“ herausgeschnitten. Erstes Polyprotein wurde bei Retroviren entdeckt. Inzwischen sind viele bekannt. Oft Serien von identischen oder ähnlichen Peptiden, z.B. Enkephalin-Vorläufer enthält 6 Met-Enkephaline und 1 Leu-Enkephalin. Posttranslationale Prozessierung, an Lys-Lys oder Lys-Arg Dipeptid-Stellen (Schnitt-signal) Komplexe Kombinationen: Proopiomelanocortin (POMC)-Gen. Gewebespezifische Proteinprozessierung (Abb.). Mehrere Polyproteine mit gemeinsamen Komponenten z.B. ABC und ABD. Beispiel: EggLaying-hormone Gene von Aplysia 5 Gene, alle enthalten ELH, aber zusätzlich verschiedene andere Peptide. Veränderter Kontext. Polypetide enthalten meist nur kleine Peptide ~ 40 Aminosäuren Wie wird der Bestimmungsort eines Proteins in der Zelle gesteuert? Man unterscheidet “freie“ und „membrangebundene“ Ribosomen. “Freie“ Polysomen sind meist an Cytoskelett assoziiert (Abb.). Aber es gibt auch monomere Ribosomen frei im Cytoplasma. Bindung hängt möglicherweise von mRNA ab, die gerade translatiert wird. Membrangebundene Polysomen translatieren Polypeptide die 1. in subzelluläre Kompartimente (z.B. Lysosomen) sezerniert, 2. in Membranen integriert oder 3. in den Extrazelluarraum sekretiert werden. Mechanismen für Integration in Membran oder Passage durch sie: Transport durch Membranen post- oder co-translational. 1) Post-translationaler Transport, z.B. in Mitochondrien, Chloroplasten Zellkern, Peroxisom: Synthese an „freien“ Ribosomen, Proteine besitzen spezifische Sequenzsignale: Mitochondrien: N-terminale amphipathische Helix 12-30 AS Chloroplast: N-terminale geladene AS (ca. 25 AS) Zellkern: interne kurze basische Aminosäurensequenz (NLS nuclear localization signal), nicht konserviert, aber austauschbar. Peroxisom: C-terminal, 3 AS (SKL) 2) Durch co-translationale Translokation gelangen Proteine, die in den Golgi-Apparat, in Endosomen oder in Lysosomen gelangen sollen oder die von der Zelle sezerniert werden müssen, zunächst ins endoplasmatische Retikulum (ER). Die Synthese erfolgt an ER-Membranassoziierten Ribosomen. Die weitere Zielfindung eines Proteins (protein trafficking) erfolgt durch eine Hierarchie von Signalen, welche die Passage durch das ER und den Golgi-Komplex steuern. Gemeinsam ist dem post-translationalen Transport in die Mitochondrien oder Chloroplasten und dem co-translationalen Transport ins ER, dass die N-terminale Leitsequenz (leader) von einer membranassoziierten Protease abgetrennt wird. Co-translationaler Transport ins ER: Komponenten identifiziert, Prozess in vitro rekonstituiert (Abb). - Leader (16-29 AS) bindet Signal Recognition Particle (SRP) = 11S-Ribonukleoproteinkomplex: 6 Proteine, 7S-RNA (305 b) - SRP bindet an Leader und an Membranrezeptor, dann Bindung des Ribosoms an Membran. - Synthese des Proteins, Leader durchdringt Membran, nimmt naszierendes Polypeptid mit, anschließend wird Signal abgetrennt von Signal-Peptidase. Verankerung von Proteinen in Membran durch Stop-Transfer Signal, beide Orientierungen möglich (Abb.). Mechanismen für Membrantransport bzw. Insertion sind zwischen E. coli, Xenopus Oozyten und menschlichen Zellen kompatibel hochkonservativ Proteine müssen auch aus dem Kern ins Cytoplasma transportiert werden. Protein enthält kurzes Leucin-reiches Kernexportsignal (NES). Durch Assoziation mit spezifischen Proteinen können NLS- und NESSignale alternativ maskiert werden, was zu ständigem Ein- und Ausschleusen des Proteins führt. Weitere Mechanismen zur Regulation auf Proteinebene: Lebensdauer des Proteins, Degradation. a) Durch Proteasen: in Lysosomen werden sezernierte Proteine hydrolysiert. Im Cytosol 4 Proteasen: 2 Ca2+ aktivierte Proteasen, eine neutrale Protease und die Ubiquitin-ATP-abhängige Protease (Proteasom). Postranslationale Modifikationen: Acetylierung, Hydroxylierung, Carboxylierung, Phosphorylierung, Gykosylierung, Acylierung, Ubiquitinierung, Sumoylierung b) Autoproteolyse 3 Möglichkeiten für die Regulation der Autoproteolyse: 1) Globale Eigenschaften: (Größe, Ladungsverteilung, Hydrophobizität, thermische Instabilität, Flexibilität) 2) Sequenz: 23 von 24 untersuchten Proteinen mit biologischer Halbwertszeit <2h enthalten Region, die reich an Prolin (P), Glutaminsäure (E), Serin (S) und Threonin (T) ist. “PEST“-Region, 12-60 Aminosäuren 3) Position des Proteins in Zelle, oder Assoziationszustand: Assoziation eines Proteins mit anderen Proteinen oder zellulären Strukturen verlängern Lebensdauer oft erheblich. Experimentelle Befunde: verkürzte Halbwertszeit bei Veränderung der Tertiärstrukutr, z.B. durch Mutation, Einbau analoger Aminosäuren, Denaturierung, vorzeitigem Kettenabbruch, Verhinderung der Assoziation. Zahlreiche Regulationsmechanismen auf Proteinebene möglich. The Mechanism of RNA Interference (RNAi) Abwehr von RNA-Viren, Transposons (Heterochromatisierung) Zusätzlicher Mechanismus zur Genregulation siRNA = small interfering RNA miRNA = micro-RNA 3 Arten von Interferenzmechanismen: 1) siRNAs (~ 23 bp) entstehen durch Enzym Dicer aus dsRNA, der RNA-induced silencing complex (RISC) spaltet sie in Einzelstrang-siRNAs komplementär zu mRNA, Degradation der mRNA 2) siRNA aktiviert RNA-induced initiation of transcriptional gene silencing (RITS)-Komplex, bindet naszierende mRNA-Transkripte lokale Heterochromatisierung 3) Translationsrepression durch miRNAs, entstehen aus Haarnadelstrukturen, Wirkung über (RISC) Einzelstrang-miRNA, bindet an 3‘UTR, blockiert Translation. Genregulation Chromatin-Remodeling Heterochromatin Euchromatin Transkriptionskontrolle Capping, poly-A, alternatives Spleißen Translationskontrolle Posttranslationale Modifikation Proteinstabilität Bindung von Transkriptionsfaktoren Nukleosome-freie Bereiche (Promotor, Enhancer) Histon Acetyltransferase mit DNA-Bindungsprotein Gen-spezifisch DNA-Demethylase mit DNA-Bindungsprotein Gen-spezifisch Cap: Stabilität, Bindung ans Ribosom Poly-A: Stabilität, Transport ins Zytoplasma Alternatives Spleißen: RNA-Bindungsproteine, blockieren Spleißstellen Translationsrepressor, miRNAs Phosphorylierung, Glykolysierung, Ubiquitinierung Abbau im Proteasom Proto-Onkogen: Ras Onkogen: mutiertes Ras (hyperaktiv) Tumor Tumorsuppressorgen: p53 Dominant Mutiertes p53: fehlende Inhibition des Zellzyklus Tumor Rezessiv Rekombination 3 Arten: Homologe Rekombination: jedes Paar homologer Sequenzen ist Substrat, Verteilung von crossing-over etwa konstant über Chromosom. Drosophila: keine Rekombination in Nachbarschaft zu cross over, keine Rek. in oder nahe Heterochromatin, keine Rekombination in Männchen. Ortspezifische Rekombination: Spezielle Sequenzen + Homologie, z.B. Phagenintegration in Bakterien Genom, somatische Rekombination bei Immunglobulin- und T-Zell-Rezeptor-Genen - Replikative Rekombination: Transposable Elemente 1) Homologe Rekombination Mechanismus (Prinzip): Bruch + Wiedervereinigung, auf Base genau. Voraussetzung: Paarung homologer Sequenzen Eukaryonten: Synapsis (Synaptonemaler Komplex). (Abb.) Chromatin laterales Element zentrales Element laterales Element Chromatin Generelle Strukturen: Laterales Element, zentrales Element. Details von Spezies zu Spezies unterschiedlich. Abstand der Chromosomen ~ 200 nm = 100 x DNA-Durchmesser (2 nm) 2. Ortspezifische Rekombination: - Phagenintegration POP‘ Prophage BOB‘ BOP‘ POB‘ Hefe: Plasmid kodiert für FLP-Rekombinase, diese wirkt auf bestimmte Zielsequenz (FRT), 2 FRTs ( ) auf Plasmid Manipulation des HefeGenoms. Invertierte Orientierung Inversion A D B C A D C B direkte Orientierung im Genom: Exzision Ring wird deletiert Höhere Eukaryonten: keine sequenzspezifische Rekombination bekannt außer somatischer Rekombination in Immunglobulin- und T-Zellrezeptor-Genen. Einsatz des Hefe-Systems in Drosophila: Mutante für kloniertes Gen 1. Transformation mit Hefe-Rekombinasegen unter Hitzeschock-Promotor 2. Transformation mit kloniertem Gen zwischen 2 FRTs. Hitzeschock zu bestimmter Zeit, in bestimmtem Gewebe Mosaiktiere, da kloniertes Gen herausgeschnitten wird (nach Hitzeschock). Wichtig für Funktionsstudien von lebenswichtigen Genprodukten Immungenetik: Entstehung der Antikörpervielfalt Somatische Rekombination (ortsspezifische Rekombination) Somatische Rekombination (DNA) Alternatives Spleißen für konstante Region der schweren Kette: membrangebundenes Protein: B-Zell Rezeptor sezerniertes Protein: Antikörper Ähnliche Mechanismen verantwortlich für Vielfalt des T-Zell Rezeptors Der Immunglobulin-Klassenwechsel Somatische Rekombination (DNA) Alternatives Spleißen (RNA): membrangebundenes Protein: B-Zell Rezeptor sezerniertes Protein: Antikörper Das Hardy-Weinberg Gesetz beschreibt die Beziehung zwischen Allel- und Genotypfrequenzen einer nicht evolvierenden Population Beispiel: Etwa 0,04% der menschlichen Bevölkerung leiden unter Mucovisczidose, einer autosomal rezessiv vererbten Krankheit, bei der das Gen für einen Chloridkanal mutiert ist und sich daher z.B. in der Lunge zäher Schleim bildet, in dem sich Krankheitserreger (z.B. für Lungenentzündung) einnisten. Können wir ausrechnen, wieviele Überträger es in der Population gibt? Die Häufigkeit des gesunden Alles in der Population bezeichnen wir mit p, die des defekten Allels mit q, dann ist p+q=1 Die Wahrscheinlichkeit, dass ein Individuum zwei defekte Allele besitzt („und“), ist (wie beim Würfeln) q x q = q2 also ist q in unserem Beispiel q = 2% (0,02 x 0,02 = 0,0004). Ebenso ist die Wahrscheinlichkeit, dass ein Individuum zwei gesunde Allele besitzt = p2. Die Wahrscheinlichkeit, dass das erste Allel gesund und das zweite defekt = p x q, „oder“ dass das erste defekt und das zweite gesund = q x p, also die Wahrscheinlichkeit für Überträger = 2pq. („und“ Wahrscheinlichkeiten werden multipliziert, bei „oder“ addiert) Diese Beziehungen lassen sich in der Formel ausdrücken (p + q)2 = p2 + 2pq + q2 = 1 In unserem Beispiel bedeutet dies, dass von 100 000 Personen in der Population (z.B. in Würzburg) etwa 40 krank, 3 920 Überträger und 96 040 gesund sind. Nicht evolvierende Population bedeutet: Keine Selektion, keine Neumutationen, keine Migration, sexuelle Fortpflanzung mit zufällige Paarungen, große Zahl von Individuen. Bei kleinen Populationen: zufällige Veränderung der Allelhäufigkeit: „Genetische Drift“ Gentechnik Transgen: eingeschleuste fremde DNA Das Insulingen kodiert für ein Pro-Protein, dieses wird prozessiert zu zwei Ketten, die chemisch modifiziert und wieder gekoppelt werden. Diese Dimere bilden die aktive Form des Insulins. In Bakterien werden die beiden Ketten getrennt von cDNA exprimiert. Produktion von Insulin cDNA cDNA Knock-out Mäuse sterben weil nicht neo-resistent Embryonale Stammzellen sterben weil Thymidin-Kinase aus Ganciclovir Zellgift macht Embryonale Stammzellen mit Mutation „Gentherapie“ Repetitive DNA: Nichtkodierende funktionelle DNA Variable number of tandem repeats: VNTR sind 1-5 kb Fragmente, die aus repetitiven Einheiten bestehen (15 bis 100 Basen lang). Diese Fragmente können zum DNA finger printing verwandt werden. Mit PCR amplifiziert DNA finger printing Transposable Elemente in Eukaryonten Ähnlich in Pro- und Eukaryonten Einfache Transposons: Insertions-Sequenzen = IS-Elemente (Abb.) 1. Haben “inverted repeats“ an Enden, 15-31 bp lang. 2. Kodieren für ihre eigene Transposase = Enzym, das Transposition katalysiert. Inverted repeats = Erkennungssequenz für Transposase. 3. Bei Insertion werden 5-9 bp der Ziel-DNA verdoppelt, Transposon liegt zwischen diesen “direct repeats“. 4. Insertionsstelle beliebig oder mit Sequenzbevorzugung (hotspots) Transpositionsrate 10-3 – 10-4 pro Generation Zielsequenz versetzte Schnitte Einfügen der Transposons IR Transposase-Gen Auffüllen der Lücken, Ligation DR IR = inverted repeats IR DR DR = direct repeats Komplexe Transposons: aufgebaut aus vollständigen unvollständigen IS-Elementen (Abb) “Tn-Elemente“ IS Marker (z.B. Resistenz) komplexes Transposon IS oder 2 Arten von Transposition: konservativ: Donor wird meist zerstört durch Transposition replikativ: Duplizierung des Transposons Transposon Ziel-DNA Transposon Ziel-DNA ? konservativ replikativ Folgen: 1. Zerstörung von Genen durch Insertion eines Transposons (p = 10-5 – 10-7 pro Generation spontane Mutationsrate); Reversion durch Rekombination zwischen direct repeats möglich, aber selten (p = 10-6 – 10-10 pro Generation) 2. Rekombination zwischen 2 Transposons ( ) auf einem Chromosom Deletion, wenn gleichgerichtet Inversion, wenn entgegengesetzt gerichtet Deletion Inversion 3. Rekombination zwischen 2 Transposons auf verschiedenen Chromosomen Translokation Funktion in Evolution oder parasitär (“selfish genes“)? Beispiel für Genregulation am Modellsystem Hefe: Regulation mehrerer Gene gleichzeitig ähnlich wie Bakterienmodell GAL80 GAL4 GAL-Gene: kodieren für Enzyme des Galaktose-Stoffwechsels UASG = Enhancer, wirkt auf 2 Gene (GAL10, GAL1) GAL80 verändert nicht DNABindung von GAL4, blockiert seine Aktivierungsfunktion Enhancer UASG nicht aktiv Galactose GAL80 GAL4: 3 Funktionen: 1) DNA-Bindung (Cys2Cys2) 2) Transkriptions-Aktivierung 3) GAL80-Bindung GAL10 EnhancerUASG aktiv GAL1 Cell-specific expression of transgenes in Drosophila using the UAS/Gal4 system (Brand & Perrimon 1993) Transgenic driver lines: Transgenic effector lines: Cell-specific enhancers of known or unknown genes reporters (e.g. GFP), sensors for calcium (e.g. Cameleon, G-CaMP), synaptic blockers (e.g.tetanus toxin, shits) Gal4 Fly stock GAL4 Cell-specific enhancer sequence X UAS Fly stock UAS cDNA-X polyA UAS/Gal4 flies GAL4 GAL4 UAS Cell-specific expression of effector cDNA-X polyA Cell-specific enhancer sequence Gal4 drivers: ubiquitous, neurons, types of neurons, muscles ... Modified from Martine Simonelig From flybrain.neurobio.arizona.de Ein neues genetisches Werkzeug für die Gehirnforschung Optophysiologie: Channelrhodopsin (Chop) Chlamydomonas modified from: Flannery and Greenberg, 2006