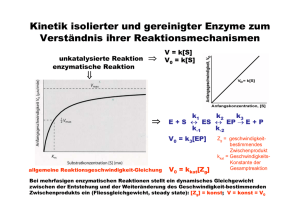
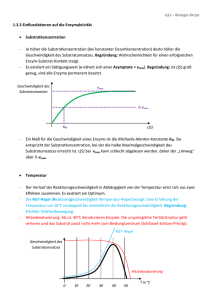
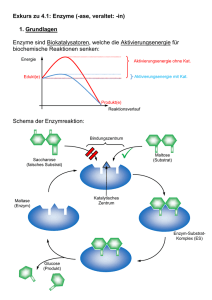
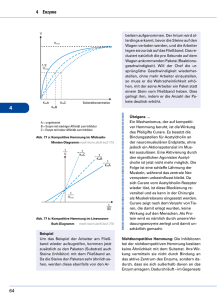
E S ES K - LIMES
Werbung

Vertiefendes Seminar zur Vorlesung Biochemie I 04.12.2015 Bearbeitungg Übungsblatt g 6 Gerhild van Echten-Deckert Fon. +49-228-732703 Homepage: http://www.limes-institut-bonn.de/forschung/arbeitsgruppen/unit-3/ Energiediagramm einer Reaktion Enzym Abhängigkeit der Geschwindigkeit einer Enzym katalysierten Enzym-katalysierten Reaktion von der Substratkonzentration. Wie verändern sich die Konzentrationen der Teilnehmer im Verlaufe einer einfachen MM-Reaktion? Michaelis--MentenMichaelis Menten-Modell E +S frei k+1 k-1 ES k+2 a) b) E +P frei k-2 V0 k2 *[ ES ] k1 :[[ V f k1 *[ E ]*[ S ] Vd k1 *[ ES ] K 2 [ ES ] Anfangsgeschwindigkeiten : k-2 = 0 Fließgleichgewicht : [ES] = C 1 L ] s mol 1 k1 :[ ] s Fließgleichgewicht : [ES] ist konstant Vf=Vd k1 *[ E ]*[ S ] [ ES ](k2 k1 ) [ E ]*[ S ] k1 k2 K M ; Michalis-Menten-Konstante [ ES ] k1 [ ES ] [S ] ; [ E ] [ Et ] [ ES ] [ Et ] K M [ S ] [ ES ] [ Et ]* [S ] K M [S ] [S ] V0 k2 [ Et ]* KM [S ] Bei Substratsättigung [ES]=[Et] Vmax k2 [ Et ] V0 Vmax * [S ] KM [S ] Michalis-Menten-Gleichung Michaelis--MentenMichaelis Menten-Modell Alternative Michaelis-Menten-Gleichung : KM Vmax Vmax V0 Oder : V0 [S ] KM V0 1 In vitro, [S] Überschuss , [E] begrenzend [S ] v Vmax V0 Vmax 1 V max 2 KM [S] [S ] K M [S ] Grenzfälle : [S ] v steigt fast linear mit [S] KM a) Für [S] KM V0 V max b) Für [S] = KM V0 1 2 V max Definition von KM : v halbmaximale Substratkonzentration c) Für [S] KM V0 V max v maximal, Grenzwert Bestimmung von KM und Vmax : Lineweaver Lineweaver--Burk [S ] V0 Vmax * K M [S ] Für 1 1 1 0 V0 [S ] KM Für 1 1 1 0 [S ] V0 Vmax K 1 1 1 M * V0 Vmax Vmax [ S ] Die Geschwindigkeit einer Michaelis-Menten Reaktion als Funktion der Substratkonzentration Lineweaver–Burk i k Auftragung A f Michaelis--MentenMichaelis Menten-Modell E +S frei k+1 k-1 ES k+2 k-2 E +P frei Für k-11 k+2 (ES zerfällt schneller in E + S als in E + P) k1 k2 1 sec KM 1 sec * l mol k1 k 1 Dissoziationskonstante von ES KM k 1 KM-Werte einiger Enzyme Enzym Substrat Chymotrypsin Lysozym -Galactosidase Threonin-Deaminase Carboanhydrase Penicillinase Pyruvat carboxylase Acetyl-L-tryptophanamid Hexa N Acetylglucosamin Hexa-N-Acetylglucosamin Lactone Threonin CO2 Benzylpenicillin Pyruvat HCO3ATP Arginin tRNA ATP Arginin t-RNA Synthetase KM 5x10-3M 6x10-66M 4x10-3M 5x10-3M 8x10-3M 5x10-5M 4x10-4M 1x10-3M 6x10-5M 3x10-6M 4x10-7M 3 10-44M 3x10 Je kleiner KM desto stabiler ES, desto höher Affinität von E für S Vmax k2 *[ Et ] k2 Wechselzahl *[ Carboanhydrase : diffusionskontrolliert Acetylcholinesterase auch : S Moleküle ] k2 kcat E Moleküle *sec HCO3- + H+ H2O + CO2 O O O N Enzym Carboanhydrase Acetylcholinesterase + H2O O 600000 25000 2000 Lactat-Dehydrogenase 1000 DNA-Polymerase I HO N [s-1] Penicillinase Chymotrypsin + 100 15 Tryptophan-Synthetase 2 Lysozym 0,5 Maximale Wechselzahlen einiger Enzyme Michaelis--MentenMichaelis Menten-Modell kcat KM Katalytische Leistungsfähigkeit eines Enzyms Für [S] << KM (in vivo) gilt : Et Efrei und aus V k2 *[ ES ] I in II wird [ ES ] [ Et ]*[ S ] KM (I) (II) k2 *[ S ]*[ Et ] V KM k2 k1 KM [ Et ]*[ S ] [ ES ] K M [S ] ; 1 k1 K M k1 k2 (diffusionskontrolliert), da : Und k2 k2 * k1 K M k1 * k2 k2 k1 k2 stets kleiner als 1 k+2=kkcat kann k maximal i l k+1 erreichen i h (wenn ( k-1<< k+2) ddann iistt E diffusionskontrolliert: diff i k t lli t k+1 max = 108-109 M-1s-1 Kinetische Messungen zum EnzymEnzym-Optimum Plot : v als Funktion von pH • Veränderung der Nettoladung z.B. –COO- zu –COOH • Wendepunkt bei pH ca ca. 6 deuten oft auf ein Histidin • Wendepunkt bei pH ca. 3,5 deuten auf Aspartat oder Glutamatrest Temperatureffekte • bis 40°C Verdoppelung von V bei Erhöhung von je 10°C • Später Inaktivierung durch Denaturierung Substratkonzentration • häufigg „Produkthemmung“ „ g durch unspezifische p Bindung, g, ggf. gg Blockade des aktiven Zentrums • pS = - log [S] Enzymkinetik (Inhibitoren) 1. 2. Irreversible Inhibitoren Suizid-Inhibitoren Reversible Inhibitoren (a) kompetitive Inhibitoren : Substratanaloga (EtOH bei MeOH od.Glykolod Glykol Vergiftung), Vergiftung) Übergangszustandsanaloga (b) nichtkompetitive Inhibitoren Angriffspunkte: Aktives Zentrum, Inhibitoren werden durch das Substrat verdrängt I Bereichen In Be ei he außerhalb ße h lb des de aktiven kti e Zentrums Ze t Keine Verdrängung durch das Substrat Erniedrigung der Wechselzahl Unterschied zw einem kompetitiven und einem nichtkompetitiven Inhibitor Reversible Inhibitoren Kompetitiv K i i (aktives ( ki Zentrum, Z häufigster hä fi Typ) T ): • reversible Bindung, aber kein Umsatz • Erschwerung des Substratzutritts • keine Veränderung des Katalyseeigenschaften Vmax wird nicht verändert Kompetitive Inhibition E k Erkennung d durch h ki kinetische ti h M Messung : KM steigt t i t Vmax unverändert 1 1 = V KM (1 + [I]/KI) 1 Vmax [S] + Vmax Kompetitive Inhibition A double reciprocal plot of enzyme kinetics in the presence (blue, red) and absence (green) of a competitive inhibitor. Vmax is unaltered whereas KM is increased Nichtkompetitive Hemmung Nichtkompetitive Hemmung Unkompetitive Hemmung Erkennung durch kinetische Messung : KM unverändert Vmax fällt • häufig bei zwei oder mehr Substraten • keine Blockade oder Verdrängung des Substrats • Enzym wird auch unabhängig vom Substrat inaktiviert • Effekt : scheinbare Verringerung der Enzymkonzentration • meist kein Effekt auf KM • unkompetitive Inhibitoren binden nur an den ES-Komplex Unkompetitive Hemmung in der Lineweaver-Burk Auftragung Gemischt-kompetitive Hemmung in der Lineweaver-Burk Auftragung Kinetik allosterischer Enzyme in Anwesenheit positiver bzw. negativer allosterischer Effektoren K-Typ: v-Typ: vmax-konstant KM-ändert ä d t sich i h± KM-konstant vmax-ändert sich ± Substratbindung an ein allosterisches Protein: SYMMETRIE-MODELL Jaques Monod Jeffries Wyman Jean-Pierre Jean Pierre Changeux “Alles oder Nichts” “Konzertiertes Modell” Substratbindung an ein allosterisches Protein: SEQUENZ-MODELL Daniel Koshland “Induced Induced fit fit” Wirkung allosterischer Effektoren auf die kooperative Substratbindung eines dimeren Enzyms niedrigaffin hochaffin 1) Michaelis-Menten kinetics and calculation of KM & Vmax KM = 4.6 µM Vmax = 12.8 12 8 nmol/min http://www.graphpad.com/guides/prism/6/curve-fitting/index.htm?reg_michaelis_menten_enzyme.htm 2. Zugabe von 4 µM eines kompetitiven Inhibitors KI = 1.5µM http://www.graphpad.com/faq/file/P4-Competitive%20inhibition.pdf